编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Christoph Bock和Matthias Samwald的一篇评估大语言模型的论文。生物过程的计算模拟可以加速生物医学研究,但通常需要广泛的领域知识和手动调整。最近,如GPT-4等大型语言模型(LLMs)已经证明在生成人类语言方面取得了惊人的成功,可用于各种任务。在这里,作者探讨了利用LLMs作为生物系统模拟器的潜力,建立了一个名为SimulateGPT的文本模拟器,该模拟器利用了LLM的推理能力。作者展示了模型在各种生物医学应用中具有良好的预测性能,无需显式的领域知识或手动调整。因此,LLMs为一类新的通用生物模拟器提供了可能。

大型语言模型(LLMs)具有令人印象深刻的能力,可以利用人类语言生成来解决问题,例如回答复杂问题和逐步构建论证。LLMs通过逐步预测前文中的下一个文本标记来自动生成文本。尽管这个基本过程很简单,但LLMs可以在医学和生物学等各个领域解决任务,有时超过人类专家。新颖的方法,如思维链式推理,进一步通过模仿复杂的因果推理模式增强了它们的能力。在这里,作者描述、实施和评估了一种基于LLMs的新模拟范式。作者的方法将LLMs从模仿人类写作和思维推进到了一个新的层面。它利用LLMs以定性的文本方式来模拟生物系统,无需显式的领域知识或手动调整。计算模拟分子、细胞和生理生物过程可以用于获得科学见解、指导实验研究,并可能促进个性化医学。基于LLMs的模拟可以补充现有的模拟范式,特别是通过利用LLMs的大规模隐式知识库和LLMs所操作的多用途的序列模型。本研究提供了一个基于GPT-4的基于文本的生物模拟器的概念验证,并在各种生物医学场景中对该系统进行了评估,并概述了将LLMs系统化地发展和应用为复杂生物过程的人工智能模拟器的路线图(图1a)。
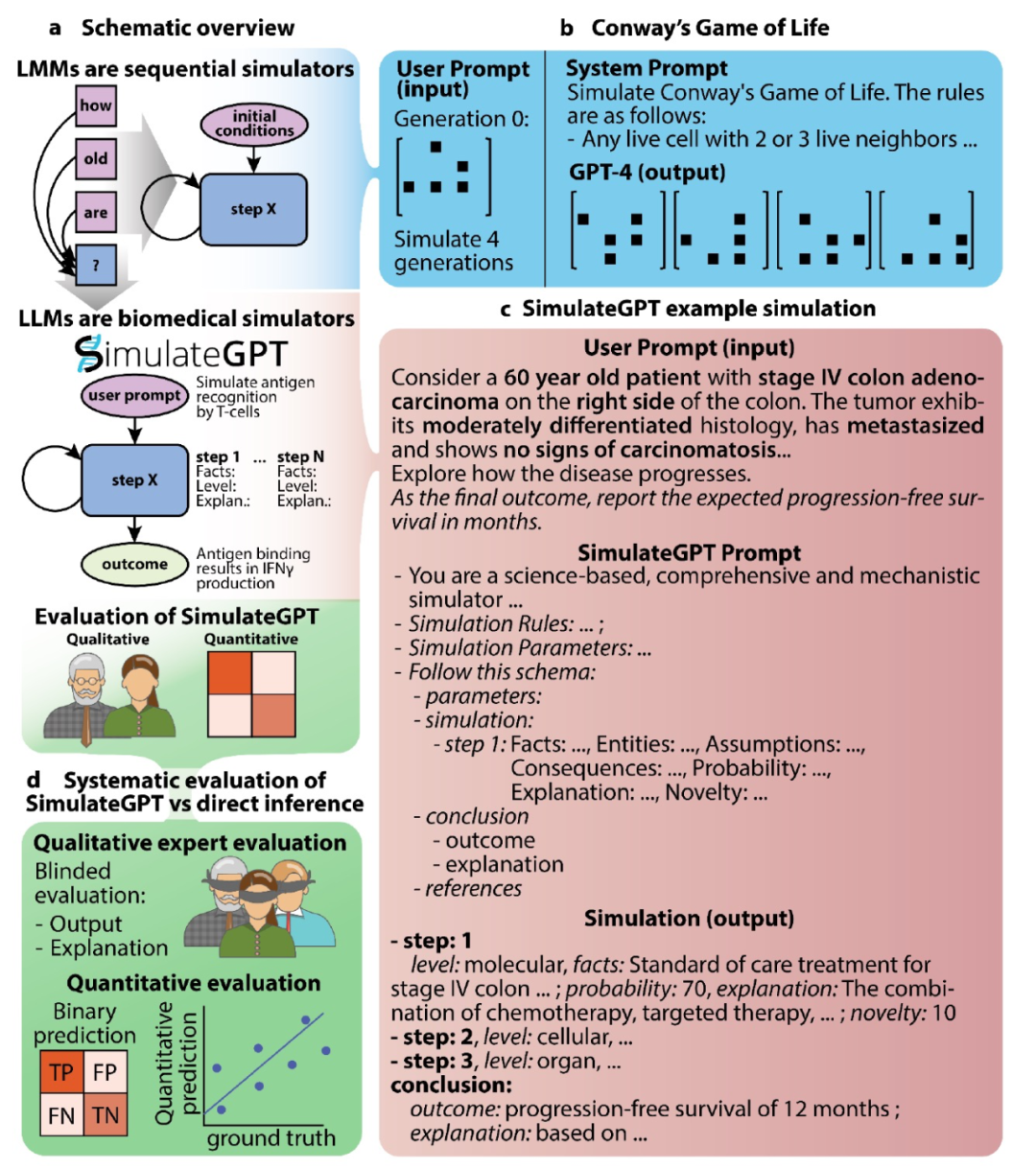
图 1
基于LLM的生物学仿真方法SimulateGPT的开发与评估
作者首先在一个简单明确的测试案例中展示了基于LLM的生物学仿真的可行性,通过在GPT-4中模拟康威生命游戏。与神经网络相比,据报道神经网络在这项任务上表现不佳,而GPT-4能够在不需要手动适应或专门训练的情况下,使用提供的更新规则模拟“滑翔机”模式的整个周期(图1b)。除了这个基于GPT-4的生物学仿真的简单概念验证,预计GPT-4可能特别适用于对复杂的生物学过程进行定性的、基于文本的仿真,这些过程很难或不可能基于它们的物理和化学基础进行建模。GPT-4包含了大量相关知识,因为它已经在大规模文本语料库上进行了训练,其中似乎包括了生物学和医学领域的大部分科学文献,以及关于各种科学和非科学话题的网站和讨论。作者发现,GPT-4可以被激发出对癌症患者癌症进展的有意义的估计。
作者开发了SimulateGPT方法,将GPT-4用作生物医学模拟器。这种方法强制执行逐步仿真,每个步骤由一个设计用于促进在多个生物组织层次上进行仿真的推理结构组成(图1c)。初步测试表明,我们的方法能够提供有意义的解释和概率,并且通常能够提供正确的科学参考来支持其推理(图1c)。为了系统评估SimulateGPT在以步骤方式仿真复杂的生物医学情景,向结果预测方向发展的能力,作者通过基于专家和数据的验证(图1d)将其性能与直接推理提示进行了比较。
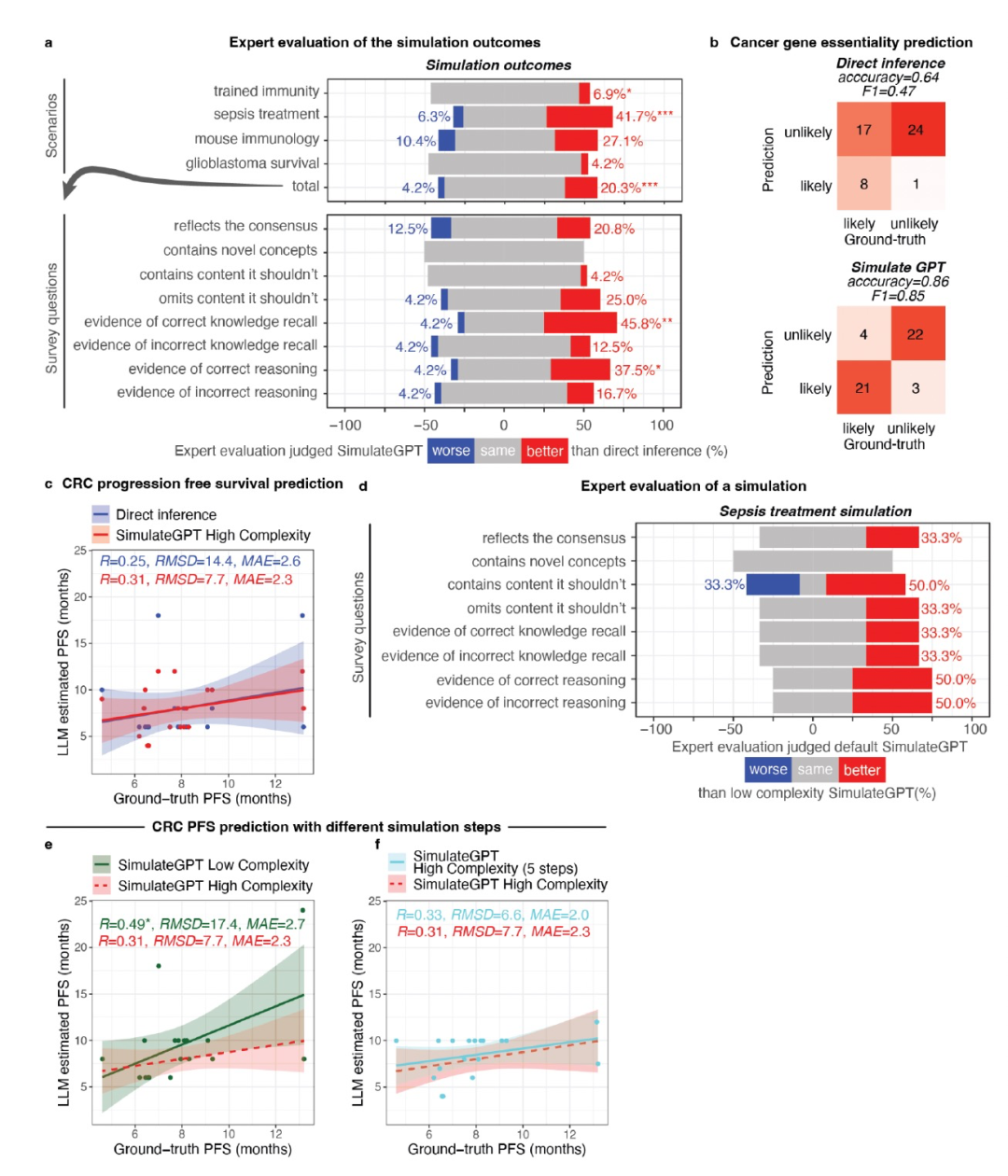
图 2
首先,作者通过带有Likert量表问题的盲目专家调查,评估了SimulateGPT在已有科学知识领域内模拟生物过程的能力,涵盖了仿真的正确性、解释性和完整性。作者制定了四个涵盖不同复杂性水平的情景:(i)具有已知结果的体内小鼠实验;(ii)探索经过训练的免疫,这是一个最近发现的还未充分研究的免疫概念,涉及先天细胞中的免疫记忆;(iii)基于有限临床参数在败血症中的新型治疗决策支持的推理;以及(iv)胶质母细胞瘤患者总体生存期中的肿瘤突变效应。对于每个情景,仿真试图建立特定的结果,以及通向仿真结果的路径的解释。作者为每个情景模拟了一到三种条件,并由博士后生物学家在盲目条件下进行排名。在所有四个情景中,SimulateGPT在结果预测方面的表现优于基于传统GPT-4的推理(图2a)。相比之下,SimulateGPT提供的解释并不比直接推理提示的解释更好。这可能是因为SimulateGPT的最终解释通常并不是自包含的,因为其中的一些部分已经由前面的仿真步骤提供(图1c)。
将SimulateGPT方法应用和扩展到分类和回归任务
大语言模型在语言推理任务方面表现出色,但在结构化预测任务方面表现较差。作者假设SimulateGPT对LLM推理施加的结构有助于解决这一弱点,从而在分类和回归任务方面提高性能。作者模拟了两个场景,其中存在公开验证的结果,但仅通过领域特定的数据存储库才能访问,因此极不可能在GPT-4的训练过程中加入这些信息。为了进一步控制GPT-4的训练过程中是否存在数据泄漏的可能性,对性能进行了与传统GTP-4推理的比较。
在第一个场景中,作者关注分子和细胞机制,通过模拟对癌细胞生存至关重要的单个基因的本质性,构建了一个零样本二分类任务。对应癌细胞系的大规模实验数据提供了真实情况。SimulateGPT表现出高的精度和召回率,在平衡类别的数据集上实现了86%的准确率(图2c)。相比之下,直接推理在预测基因为非必需基因时表现出强烈的偏见,导致准确率仅为64%(图2b)。
在第二个场景中,我们根据患者的临床表现预测结直肠癌患者的预期无进展生存期(图1c)。为了减少癌症数据集之间的巨大变异,作者通过在提示的一部分中将测试数据集中的四个示例结果作为少样本学习的一部分来校准我们的模型。无论SimulateGPT还是直接推理,都显示出低估数据集方差的倾向。尽管如此,与直接推理相比,SimulateGPT实现了较小的误差和更高的相关系数(图2c)。
在调查模拟结果时,作者注意到SimulateGPT通常会在总结之前生成三个步骤。作者想知道更多的步骤是否会提高整体性能,为此推导出一个低复杂性的SimulateGPT变体,减少了输出特征,这个变体生成了更多的步骤,这可能是由于GPT-4生成的响应具有相似的长度倾向。低复杂性的SimulateGPT变体在专家评估中表现较差(图2d),但进展生存期的预测明显改善(图2e)。为了验证这些结果,作者对SimulateGPT进行了修改,以强制至少进行五个模拟步骤。这个修改导致性能改善(图2f),进一步支持在SimulateGPT中采用逐步模拟的方法。
总的来说,作者的实验显示SimulateGPT在各种生物医学场景中具有良好的预测性能,表明LLMs可以配置为可解释的模拟器,可以在复杂的结果预测中优于不遵循SimulateGPT中实现的逐步模拟的模型。作者的方法可以轻松应用于生物学和医学领域的各种过程和应用中,有助于在最小配置下探索和预测各种情景。
参考资料
Schaefer, M., Reichl, S., ter Horst, R., Nicolas, A. M., Krausgruber, T., Piras, F., … & Samwald, M. (2023). Large language models are universal biomedical simulators. bioRxiv, 2023-06.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


