
导语
基础模型正在实现人工智能范式的转变。从传统机器学习时代关注学习算法构建,到深度学习时代转向神经网络架构设计,再到基础模型时代专注模型本身的功能性。——这种范式转变让模型得以摆脱单一任务或领域的局限,极大地推动 AI 技术的广泛应用,特别是在生物医学领域引发变革。将基础模型应用于医学领域的全科医学人工智能(GMAI),拥有跨模态输入输出、动态任务规范、根据结构化领域知识进行推理的强大能力,有望解决当前医疗 AI 的种种局限,为改善医疗健康和增进我们对生命的理解做出革命性贡献。
8月26日,在集智俱乐部与腾讯研究院共同举办的“AIS²系列学术报告和研讨活动”中,我们邀请到斯坦福大学计算机学院教授 Jure Leskovec 进行以“基础模型在全科医学人工智能中的应用潜力”为主题的报告,介绍了一系列最新研究进展,本文由集智社区成员整理自报告内容,感兴趣的朋友可以扫描下方二维码观看视频回放。 斑图地址:https://pattern.swarma.org/study_group_issue/506
斑图地址:https://pattern.swarma.org/study_group_issue/506
集智俱乐部联合西湖大学助理教授吴泰霖,斯坦福大学计算机科学系博士后研究员王瀚宸、博士研究生黄柯鑫、黄倩,华盛顿大学博士研究生屠鑫明,共同发起以“大模型与生物医学”为主题的读书会,探讨该领域的重要问题,共学共研相关文献,读书会从2023年8月13日开始,每周日早上 9:00-11:00 线上举行,欢迎对探索这个激动人心的前沿领域有兴趣的朋友报名参与。
研究领域:人工智能,大语言模型,基础模型,全科医学人工智能,多模态,AI for Science Jure Leskovec | 讲者陶如意、王昊 | 整理吴泰霖、梁金 | 审校
Jure Leskovec | 讲者陶如意、王昊 | 整理吴泰霖、梁金 | 审校 Jure Leskovec,斯坦福大学计算机学院教授,图表示学习方法 node2vec 和 GraphSAGE 的创始人之一,开创了图神经网络领域,并与他人合著了使用最广泛的图神经网络库 PyG。他的研究贡献涵盖社交网络、数据挖掘和机器学习,以及以药物发现为重点的计算生物医学。
Jure Leskovec,斯坦福大学计算机学院教授,图表示学习方法 node2vec 和 GraphSAGE 的创始人之一,开创了图神经网络领域,并与他人合著了使用最广泛的图神经网络库 PyG。他的研究贡献涵盖社交网络、数据挖掘和机器学习,以及以药物发现为重点的计算生物医学。
目录
-
基础模型在生物科学领域的应用
-
SATURN:将不同物种的细胞嵌入到共同空间
-
GEARS:预测多基因组合干扰下的基因表达变化
-
多模态大语言模型在医药领域的应用
-
GMAI:全科医学人工智能基础模型
-
RA-CM3:检索增强多模态模型
-
Med-Flamingo:具有零样本学习能力的多模态医学模型
-
打造作为研究助手的基础模型
(相关论文索引见文末论文列表)
我们目前正处于大语言模型革命中。我们拥有大量的自然语言数据,并使用 Transformer 架构来训练语言模型,以预测在这些庞大的数据集上的下一个单词或 token。这些经过大规模预训练的模型可以通过微调或上下文学习进行调整,最终应用于许多不同的领域,包括问答、文本分类、信息检索等等。对于这些突破,我们都感到非常兴奋。
这些语言模型的令人惊叹之处在于,它们具备执行多种不同任务的能力,可以总结给定的文本段落,理解表情符号并生成有趣的回复。此外,这些模型还扩展到了多模态领域,也就是说可以处理图像和文本。它们能够描述图像,并生成关于图像的文字描述。或者,给定一段文本,可以生成看起来逼真的图像,例如一只泰迪熊在纽约街头玩滑板,或者金门大桥被当成火车桥,火车在上方穿行。
这些语言模型在另一个领域取得了惊人的成功,那就是编写代码。它们能够理解计算机代码,并在文本描述和计算机代码之间进行翻译,以执行这个技术性描述。其中一个最具代表性的例子是 Github 编译器。另外,这类模型也在其他领域产生了巨大影响。尤其值得一提的是 Google 的 AlphaFold,被认为是近几年最重大的科学突破之一。AlphaFold 可以仅依靠接受蛋白质的氨基酸序列信息,预测或估计出蛋白质的空间结构。
基于人工智能、大型语言模型和图神经网络等的这些惊人进展,我们试图将所有这些统一到一个名为“基础模型”(foundation model)的概念中。我们认为基础模型是在广泛数据上训练的模型,基本上是通过自监督的方式进行大规模训练。然后可以灵活适应并用于各种下游任务。对我们来说,基础模型的概念要比仅仅局限于语言模型更加通用。
基础模型概念之所以重要,正是因为它实现了人工智能范式的转变。在传统机器学习时代,问题的关注点主要在于如何进行预测,而核心在于学习算法的构建。在深度学习时代,重点转向了表示学习,即如何设计好的神经网络架构,使其能够学习出良好的特征表示。然而,在基础模型时代,我们上升到了更高层次的思考。我们关注的是模型的功能性,即该模型具备哪些能力,我们讨论的是模型本身,而不仅仅是算法或架构。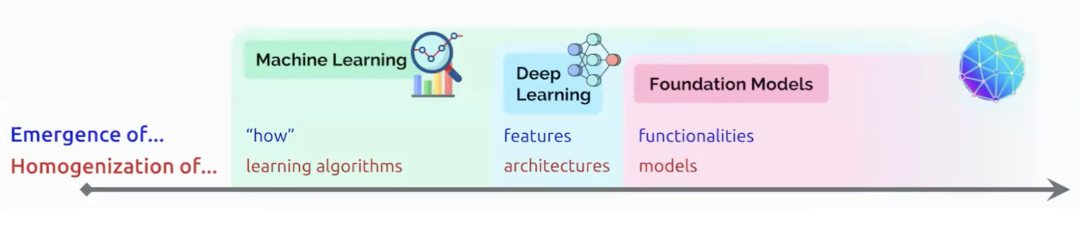 图1. 人工智能的范式转变
图1. 人工智能的范式转变
这种范式转变突出了模型本身的多功能性和适应性,不再局限于单一任务或领域。这也意味着,为了有效地适应各种下游任务,不一定需要从头开始设计一个新模型或算法。相反,我们可以从一个预先训练好的基础模型开始,然后对其进行微调,以适应特定的需求或应用。这不仅大大提高了效率,也推动了AI技术的广泛应用。
在斯坦福大学,我们成立了一个基础模型研究中心,这是一个独特的跨学科团队,由30多名教师、200名学生和博士后组成,涵盖了从计算机到医疗、教育、法律、经济学、哲学以及人文和科学等各个领域。我们的使命是使基础模型更加透明和易于获取。
我们已经撰写了一篇关于基础模型机遇与风险的重要论文,其中汇集了关于机会、能力、应用、技术以及社会挑战的全面清单,探讨了在各个领域采用、实施和部署这些大规模基础模型所面临的问题。
关于基础模型的机遇与风险Bommasani R, Hudson D A, Adeli E, et al. On the opportunities and risks of foundation models[J]. arXiv preprint arXiv:2108.07258, 2021.
在本次分享中,我将探讨基础模型效率的几个不同方面。首先,我会提及一些技术进展,然后介绍它们在生物医药健康领域的应用。最后,我会简要谈论社会责任,并探讨如何将这些大规模模型真正地部署以造福社会并实现重要的社会应用。
1. 基础模型在生物科学领域的应用
在第一部分中,我想真正谈论基础模型在生物科学领域的应用,特别是在分子生物学和药物研发方面。在这个我们共同完成的工作中,我们的目标是构建一个生物学地图。
如果大规模语言模型就像人类语言和知识的地图,那么我们希望建立一个关于生物学的地图。我们希望构建一个真正理解并能够预测人类生物学的模型。这对于医学、药物研发以及理解人类生物基础知识非常重要。
首先,对于构建生物学基础模型的问题,我们需要考虑该领域的基本要素以及可用的大规模数据集。就像语言模型使用单词序列和互联网数据源一样,在视觉领域,我们可以利用图像的像素阵列,并从互联网上获取大量数据。而在蛋白质领域,我们有氨基酸序列,存在着许多可用于训练大规模蛋白质模型的序列。这些描述了分子层面的内容,但可能过于底层。
因此,我们需要确定正确的粒度来建模生命和生物学。我们认为细胞是生命的基本单位,因此希望以细胞作为构建生物学模型的基本单位。然而,关键问题是是否有大规模可用的细胞数据来构建基于细胞的基础模型。在这种情况下,我们需要思考如何准确描述细胞行为。 图2. 基础模型(Foundation Model)分类及基本组成成分
图2. 基础模型(Foundation Model)分类及基本组成成分
下面简单介绍一下生物学过程:基因型(DNA和基因)通过转录过程生成RNA分子,然后通过翻译过程将RNA转化为蛋白质。这个过程对于表型的形成起着重要作用,实现了特定生物的具体存在。而我们真正希望理解和把握的是这个过程如何实现。因为 RNA 作为基因型与调控细胞功能的蛋白质之间的桥梁,所以我们选择在RNA水平开展研究。具体来说,RNA分子是基因信息的转录物,基因活动可以通过 RNA 数量来衡量;细胞中基因的表达可以确定或决定细胞的身份和功能。一旦获取到细胞中 RNA 的含量,就能够测量基因的表达水平,进而知道它如何调节生物功能。基本上说,细胞的身份、功能和状态都可以通过 RNA 的统计数据来确定。
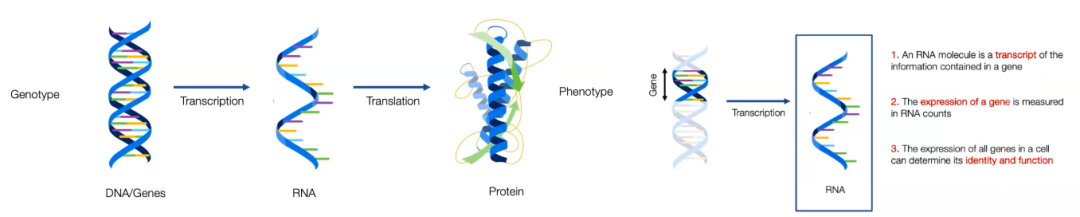
图3. 基因表达示意图
下面回顾一下DNA、RNA的测序方法。传统方法被称为 Bulk RNA-seq,用于基因组关联研究,类似于制作果汁的过程。将样本中所有细胞混合在一起,得到包含给定样本中所有细胞 RNA 计数的结果,这个过程就像将所有水果混合在一起。接下来是单细胞 RNA 测序(scRNA-seq),过程中基本上对每个单个细胞进行 RNA 测序,测量和捕捉每个细胞的状态,可以将其视为了解水果沙拉中每种水果(如菠萝、草莓、奇异果)的含量。最新的技术是空间转录组学(Spatial transcriptomics),不仅可以知道每个细胞的 RNA 计数,还可以了解细胞在空间中的位置和相邻关系。生物学家现在可以理解细胞在特定组织中的排列方式。
我们想要进行细胞行为分析的原因,是人体内所有细胞都具有相同的 DNA。因此有两个问题值得思考:
-
第一个问题是,为什么这些细胞拥有不同的功能,如大脑和肺?
- 第二个问题是,在疾病和衰老过程中,细胞发生了哪些变化?
如果我们能够理解这种状态,或许就能够将患病的细胞转化为健康的细胞。通过 scRNA-seq,我们可以精确地测量每个细胞的特征,从而帮助回答这些非常重要的问题。
下面我们思考一下这些数据是如何表示的。它们通常被表示为矩阵,每一列代表一个不同的基因。我们大约有20000个编码蛋白质的基因,所以大约有20000个基因会被转录成 RNA,然后再被翻译成蛋白质。对于每个细胞,我们都有该特定基因的 RNA 计数。因此,输入基本上就是一个有20000列和尽可能多的细胞的矩阵。我们与 Chen Zuckerberg 合作,这是 Mark Zuckerberg 和他的妻子创立的组织,旨在发现人类健康的机制。他们已经发布了一个最大的人类细胞数据集,其中包括4000万个不同的细胞,涵盖了多个细胞类型和人体组织。所以基本上,我们现在有一个20000列x4000万行的矩阵。这是我们可以使用来训练模型的大规模数据。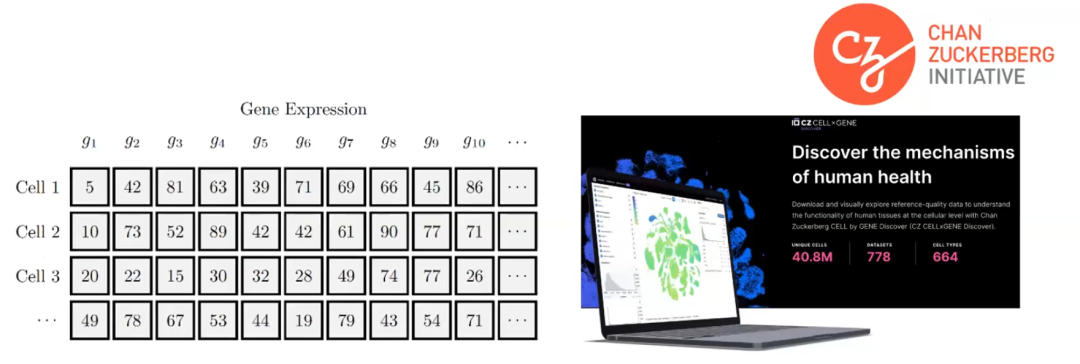 图4. 基因表达形式及大规模RNA数据集,基因表达数据用矩阵表示
图4. 基因表达形式及大规模RNA数据集,基因表达数据用矩阵表示
当我们想要构建一个利用这类数据的生物学模型时,存在两个重大挑战。第一个挑战是,很多既有工作只将基因到细胞矩阵视为索引矩阵,却忽略了基因序列或给定基因编码蛋白质的信息,同时传统的基因处理方法也缺乏对遗传变异和染色体位置的了解。第二个挑战是,如果我们得到了这些由世界各地不同团队生成的矩阵,观察这些RNA计数,并尝试将其投影到一个共同空间中,会发现这些数据集彼此之间完全不同。因此,即使是从同一个人提取的样本,这些数据集在被称为嵌入空间的低维空间中应该有重叠,但实际上实验偏差比生物信号造成的影响更大,现有方法只能学习到数据来自不同数据集,却不能精确地将所有不同人类的细胞映射到一起,而后者才是我们希望实现的目标。
为了解决这个问题,我们需要丰富我们的数据,使其不仅仅包含基因表达信息,还能更多地反映生物功能。我们提出了一种称为“功能表达”的概念,将表达式转化为反映生物功能而非特定基因的方式。通过使用大规模蛋白质语言模型,我们可以将蛋白质序列嵌入到基因功能和角色的描述中。这意味着我们现在能够将具有完全不同基因和蛋白质的不同物种嵌入到同一空间中,从而开始推断不同物种之间的关系。这带来了比以往更为显著的改进。我们特别使用了Facebook团队开发的大规模蛋白质语言模型,ESMFold,来编码整个蛋白质宇宙。该模型经过超过2.5亿个蛋白质序列的训练,并已证明其嵌入能够同时编码结构、分子性质和同源信息。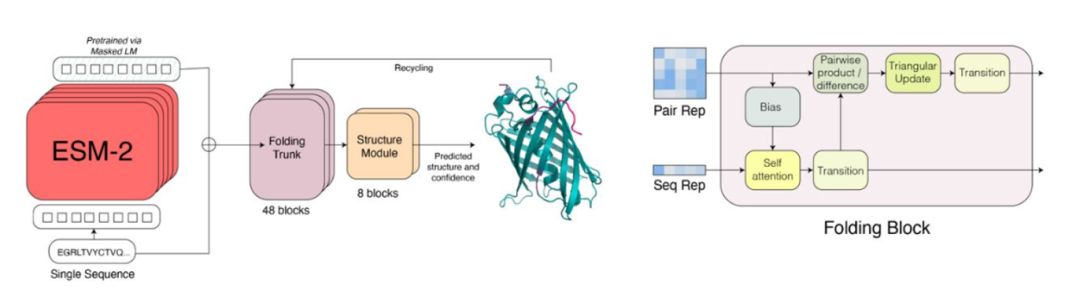 图5. ESM示意图
图5. ESM示意图
Zeming Lin et al., Evolutionary-scale prediction of atomic-level protein structure with a language model. Science 379,1123-1130(2023).DOI:10.1126/science.ade2574相关文章:《Science前沿:大语言模型涌现演化信息,加速蛋白质结构预测》
- SATURN:将不同物种的细胞嵌入到共同空间
此外,我们还开发了一种名为 SATURN 的方法。我们可以获取来自多个不同生物体(如人类、老鼠和狐猴)的 RNA 序列计数以及它们所编码的蛋白质序列,并使用大规模蛋白质语言模型推理这些不同基因在不同生物体中的相互关系,从而创建了所谓的“macrogene”概念。Macrogene 是将现实世界中所有基因连接起来的一种人工基因。通过这种嵌入,我们现在能够将任何生物体的任何细胞嵌入到共同的嵌入空间中。
在此基础上,我们能够解决许多不同的任务,例如创建多种物种的细胞分类、识别差异表达的微基因、研究底层生物学等。我们可以在物种之间传递细胞类型标签,重新定位细胞类型,并识别蛋白质差异等。我们发现采用这个方法能够准确地将模型物种的细胞类型注释传递给新物种。例如,我们可以使用斑马鱼作为模型生物体来训练我们的模型,然后对另一种动物如青蛙的细胞进行注释,比之前的方法提高了120%。因此,利用蛋白质序列的嵌入进行注释使我们取得了巨大的进展。
Rosen, Yanay, et al. “Towards Universal Cell Embeddings: Integrating Single-cell RNA-seq Datasets across Species with SATURN.” Biorxiv: the Preprint Server for Biology (2023).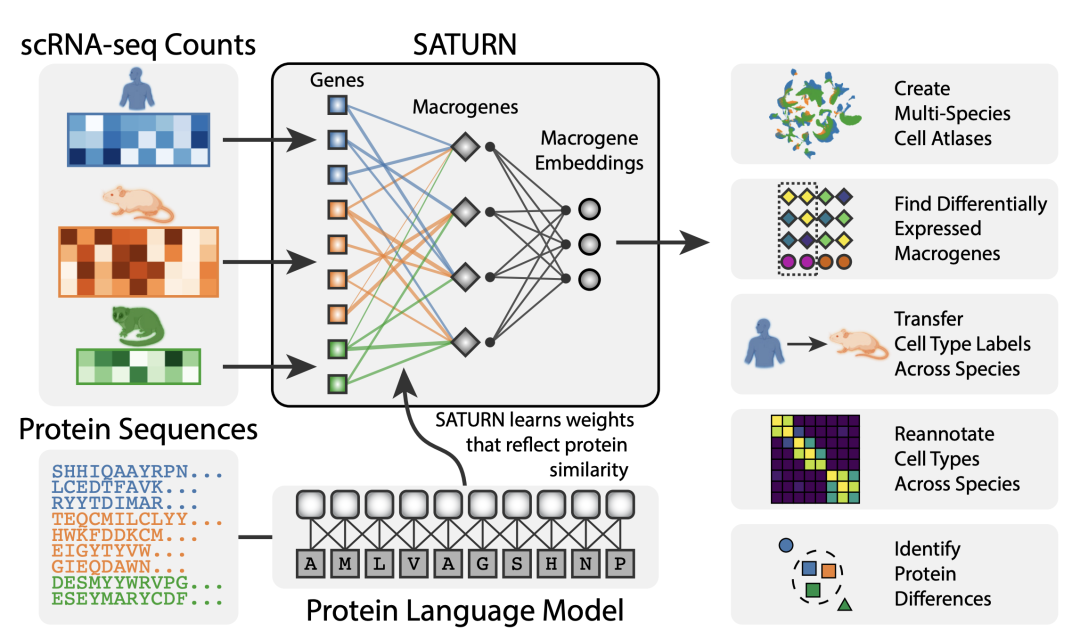
图6. SATURN 模型示意图
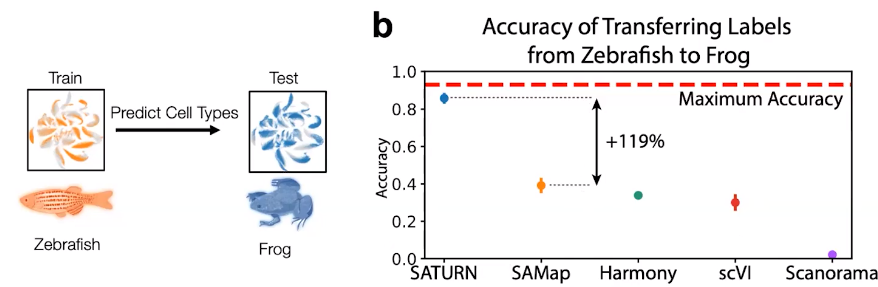
图7. SATURN 相比其他模型(如 SAMap、Harmony、scVI 或 Scandorama)的准确度
我给你展示一些真正令人印象深刻的内容。我们的方法是第一个能够将来自五个不同生物体的细胞进行着色并映射到共同空间的方法。这里每个点代表一个不同的细胞。我们从所有这些生物体中获取细胞,并将它们映射到共同空间中。下图是我们找到的聚类,这些聚类中的颜色是混合的。这意味着基本上相似类型的细胞,无论来自哪个生物体,都映射到了同一个聚类中。例如,这里下面的聚类是所有B细胞,它包括人类的B细胞、狐猴的B细胞、小鼠的B细胞等等。还有一个巨噬细胞的聚类。你可以看到,在颜色方面,它们来自不同的生物体,但在细胞类型方面非常清晰。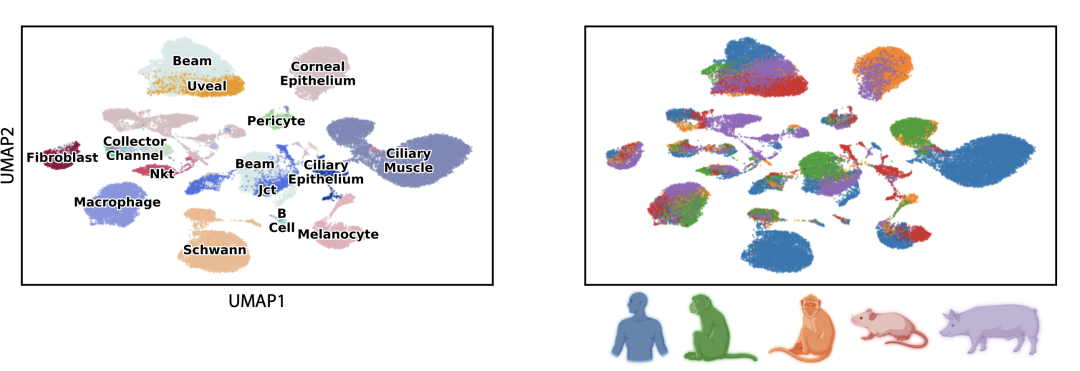 图8. 将来自五个不同生物体的细胞进行着色并映射到共同空间,相似类型的细胞,无论来自哪个生物体,都映射到同一个聚类。
图8. 将来自五个不同生物体的细胞进行着色并映射到共同空间,相似类型的细胞,无论来自哪个生物体,都映射到同一个聚类。
这是我们首次能够将来自非常不同的生物体的数据映射到共同空间中,因此我想首先讨论构建该细胞空间。在这个细胞空间中,来自任何生物体的细胞都可以被映射,以便了解这些细胞的状态、身份和功能等信息。一旦我们理解了这一点,就可以不仅仅停留在识别上,还可以预测细胞的行为,从而设计出我们所期望的细胞。例如,当我们面临一个病态细胞时,需要知道如何使其恢复健康。我们可以将这看作是一种预测任务,即如何预测干扰细胞会产生什么结果。
- GEARS:预测多基因组合干扰下的基因表达变化
再比如,如果给定一个处于特定基因表达状态的细胞,我们可以思考抑制或放大会有怎样的影响。如果干扰基因2和基因4会发生什么,细胞状态会如何改变?比如,如果我服用一种抑制基因2和基因4编码的蛋白质功能的药物,那么细胞的状态会发生什么变化?这些基因在该组合中的表达水平会有何变化?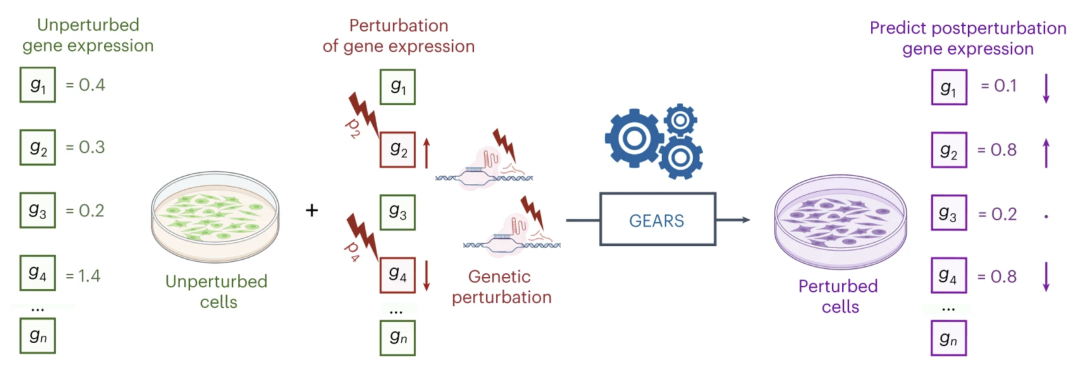 图9. 基因组合干扰下的基因表达变化示意图
图9. 基因组合干扰下的基因表达变化示意图
我们想要回答的问题是,当干扰多个基因的组合时,基因表达会如何响应,结果会是怎样的。为此,我们提出了一种名为GEARS的方法,最近在《自然·生物技术》杂志上发表。该方法利用先前的知识约束深度学习模型,结合了基因关系的知识图谱和通过基因本体论理解的干扰关系图谱所提供的知识。
Roohani, Y., Huang, K. & Leskovec, J. Predicting transcriptional outcomes of novel multigene perturbations with GEARS. Nat Biotechnol (2023). https://doi.org/10.1038/s41587-023-01905-6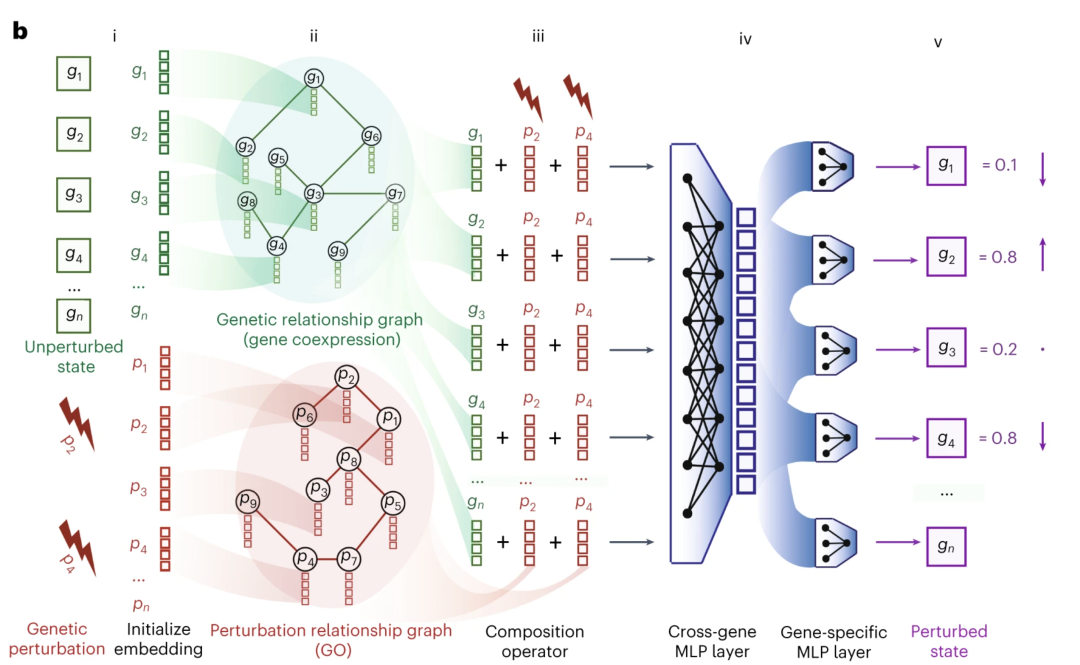 图10. GEARS 架构示意图
图10. GEARS 架构示意图
使用这种方法,我们将细胞的状态作为输入,即未经干扰的细胞状态,并提供希望进行干扰的基因信息。通过神经网络和转换,预测干扰后的基因表达情况。令人振奋的是,尽管这个过程实际上非常复杂,我们可以做出非常准确的预测。
比如,如果我干扰一个细胞的两个基因,然后想知道对该细胞的干扰将如何影响其他基因。下面是图例的说明。灰色条形表示两个基因干扰后的基因表达情况,例如,有些基因的表达显著增加,而其他基因的表达下降了。红色是我们的预测结果,可以看到预测与真实情况非常匹配。黄色是来自线性模型的预测,也就是说,我们预测第一个基因的结果,再预测第二个基因的结果,然后将两者相加并观察结果。可以看到,黄色在许多不同情况下要么严重高估,要么严重低估,这意味着这个基本过程是高度非线性的。我们发现GEARS方法在检测正确的非线性遗传相互作用方面的准确性比其他方法提高了50%,在预测最强的相互作用方面更准确两倍。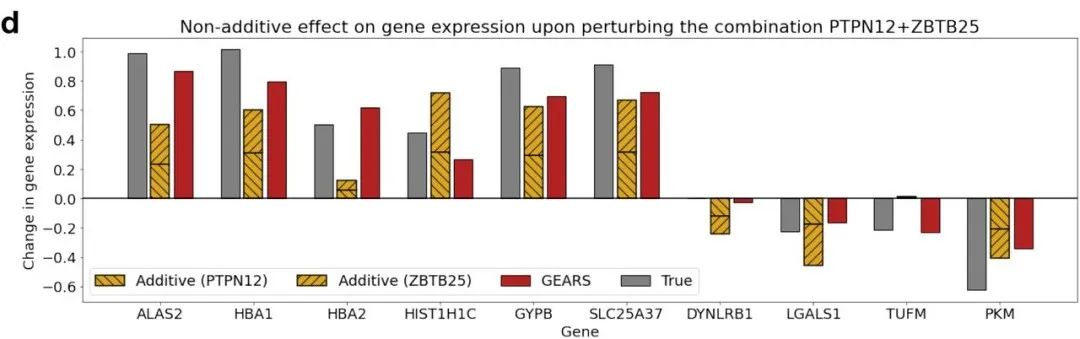 图11. GEARS 可准确预测非加成组合效应和基因相互作用亚型。
图11. GEARS 可准确预测非加成组合效应和基因相互作用亚型。
这种能力非常有用,因为它帮助我们进行靶点发现。我们可以询问:我有一个健康的细胞,应该选择哪些基因作为靶点,才能使其变成一个不健康的细胞?或者,如果我有一个病态的细胞,应该选择哪些基因作为靶点,才能使其恢复健康?这样,我们就能确定治疗疾病表型的可逆转靶点。此外,它还可以帮助我们在计算上确定新的遗传相互作用,以预测遗传相互作用。对于细胞工程来说,我们可以探讨如何将一个处于某种状态的细胞(比如老化的免疫细胞)转变成年轻的细胞。通过计算上确定目标,我们能够提高细胞重编程的精确性和有效性。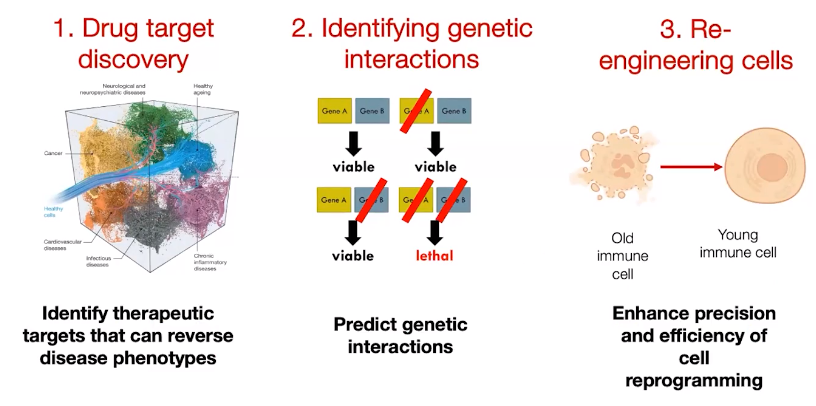 图12. 精准预测的用途
图12. 精准预测的用途
总结一下这部分工作:生物学不是一种语言,更不是自然语言,因此在将自然语言或视觉工具应用到生物学时需要谨慎。通过构建基于生物数据和生物单位(或标记)的基础模型,我们可以构建零样本(zero-shot)工具来理解细胞生物学,并真正为药物发现、医疗保健和我们对生命的理解做出革命性贡献。目前,我们正在研究一些令人兴奋的问题,围绕探索嵌入空间的结构以发现关于细胞层次结构和功能的新见解。我们还在整合患者来源的单细胞数据,以学习临床相关的信息,特别是关于个性化医疗的信息。
2. 多模态大语言模型在医药领域的应用
- GMAI:全科医学人工智能基础模型
在第二部分,我想谈论一下最近我们在《自然》杂志上发表的一篇论文,“关于全科医学人工智能的基础模型”,主要是思考如何构建医学的基础模型。这个领域的主要问题在于,首先医学数据具有稀缺性和异质性的特征,有文本、电子健康记录、测量数据、心电图、图像等等。同时,医学预测任务也非常复杂,需要大量的领域知识。而与此同时,目前的医学AI系统几乎没有利用前沿的医学知识,因为大部分医学知识实际上是写在书籍中的。因此,我们的观点是,具有结构化医学知识的医学基础模型能够解锁泛化能力,并且在多个医学预测任务中找到应用。
Michael Moor, Oishi Banerjee, Zahra Shakeri Hossein Abad, Harlan M. Krumholz, Jure Leskovec, Eric J. Topol & Pranav Rajpurkar. Foundation models for generalist medical artificial intelligence. Nature 616, 259–265 (2023). https://doi.org/10.1038/s41586-023-05881-4
传统的医学AI方法是精选一些带有标签的数据集,然后构建一个预测模型,但这种方法有很多缺点。比如,这些模型只适用于特定任务,其次手动数据标签非常昂贵,并且需要大型的特定任务数据集才能获得良好的性能。那有什么替代方案呢?我们认为预训练模型是一个替代方案。预训练模型具有广泛的适用性,以自监督的方式进行训练,因此不需要任何标签,也不需要大型的特定任务数据集,但可以在大型的任务诊断数据集上进行训练。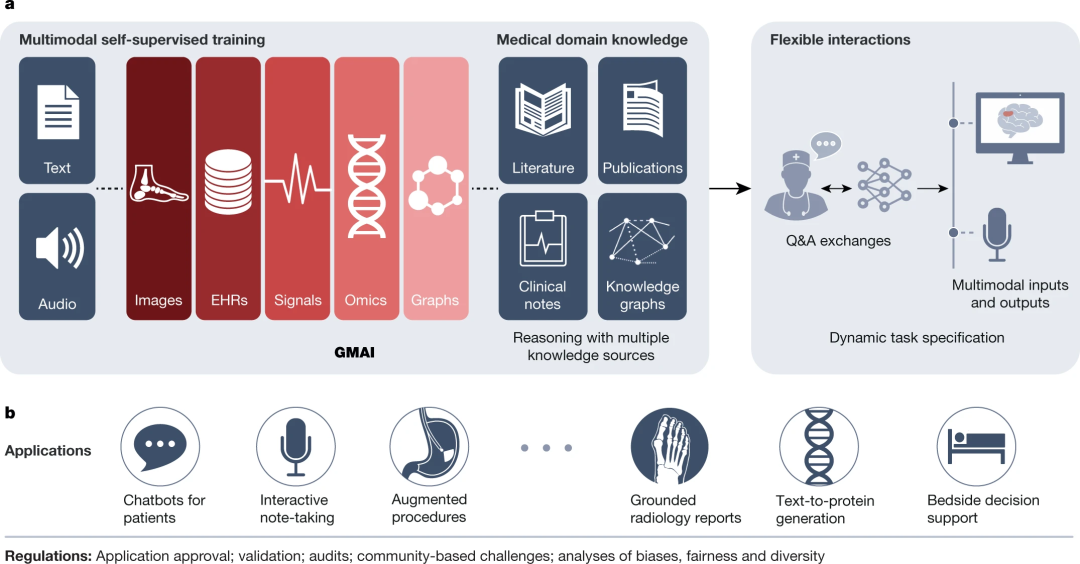 图13. GMAI(generalist medical AI)模型流程图概览
图13. GMAI(generalist medical AI)模型流程图概览
所以我们提出了全科医学人工智能的概念,即多模态的基础模型,它可以训练来自图像、EHR 信号、组学数据(基因组学和转录组学)等等许多不同类型的数据,以及能够摄取医学领域的知识方面的出版物、教科书、临床节点的知识图谱,还具有零样本学习能力,根据几个例子就可以学习去做模型从来没有见过的任务。
这样做的好处是我们得到了灵活的多模态能力,能够自由地组合和交错不同的数据模态。我们允许动态任务规范(dynamic task specification),即根据这些指令即时解决任务。因此基本上是零样本或少样本的上下文类型学习。这些模型具有大量的知识,因为它们允许我们用结构化领域知识进行推理。并且模型有很多不同的使用场景,从床旁决策支持(bedside decision support)到生成基于现实情境的报告,以及在医生决定医疗程序时辅助决策。
当然,这些领域也有很多挑战,包括验证(validation)、确认(verification)、社会偏见(social biases)以及非常重要的患者隐私问题。要解决这些问题以推动全科医学基础模型的出现,真正的技术挑战在于多模态,也就是我们可能有图像的MRI扫描,有关于患者情况的定义或描述,然后需要预测在这种情况下大脑出血的风险。而当前多模态模型的问题在于它们缺乏知识并且容易产生幻觉。
图14. 多模态面临的挑战示意。(第一行)如果你今天问一个多模态基础模型:“亚美尼亚教堂是什么样子的?” 它会给你一个教堂的图片,但其实是错的,亚美尼亚教堂有其特定的风格。(第二行)如果你问这个地方叫什么名字,它可能会说这是“龙和虎塔旁边的烟火”,但实际上是“帝国大厦和烟火”。
- RA-CM3:检索增强多模态模型
如何解决这种幻觉问题并将更多信息和更多领域知识引入这些大型生成模型,灵感来源于增强检索的语言模型。例如,对于仅基于文本的单模型(Uni model),我们可以使用 Wikipedia 并从中检索信息,然后将其作为生成器或语言模型中提示的一部分,以提供准确的答案。所以如果我们问“美国总统是谁?”我们会从 Wikipedia 中检索知识,然后用它作为语言模型生成器的上下文来提供答案。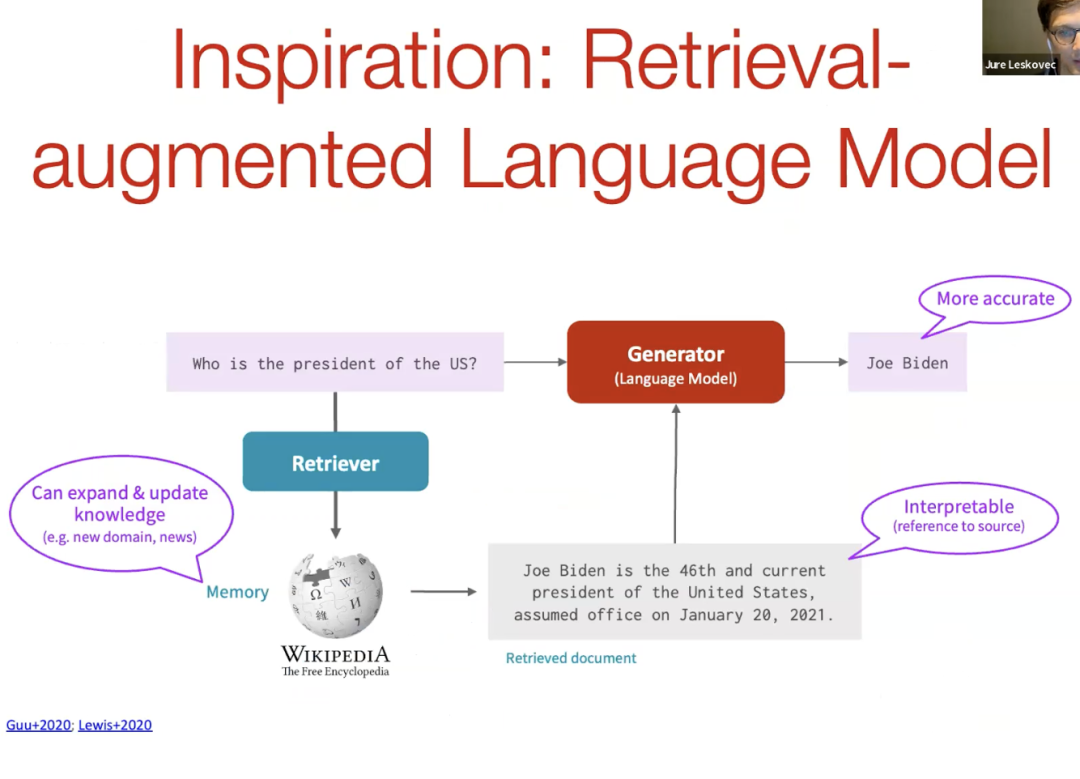 图15. 领域知识检索加上大语言模型
图15. 领域知识检索加上大语言模型
基于这个思路,我们与 Facebook 合作开发了一种首创的检索增强多模态模型(Retrieval-augmented multimodal model),这是一种允许跨多个领域进行推理的检索增强模型。例如,我们可以获取一张图片,说“生成一张拉布拉多犬坐在水边长凳上的图片”。我们会从多模态记忆中检索,可能检索到的是“一只拉布拉多坐在长凳上”,然后用这作为我们多模态模型(生成器)的上下文,生成“一张拉布拉多坐在水边长凳上”的图片。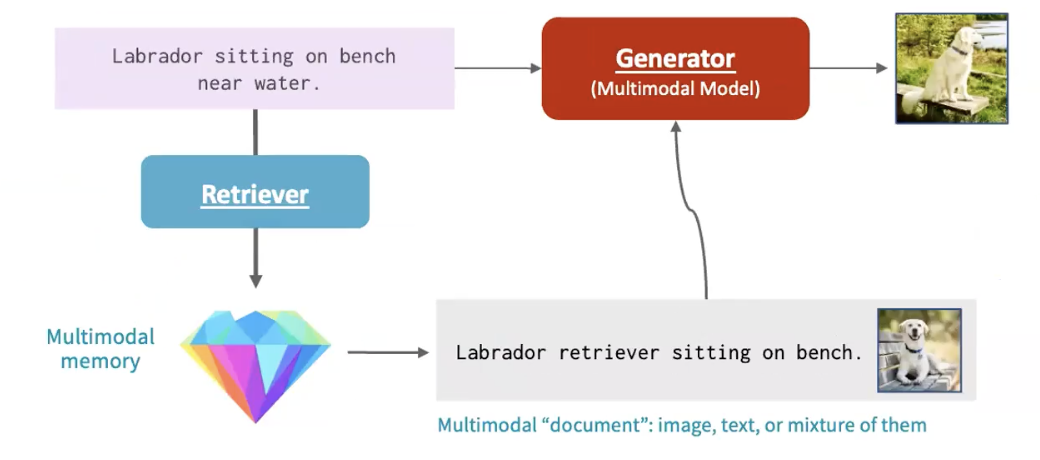
图16. 领域知识检索加上多模态模型,即检索增强多模态模型(Retrieval-augmented multimodal model)
Yasunaga, Michihiro, et al. “Retrieval-augmented multimodal language modeling.” (2023).
这是我们今年在 ICML 上发表的关于 RA-CM3 的论文。我们的创新在于构建了这个多模态检索器,其中有一个混合模态编码器的密集检索器,我们用它来计算查询文本以及从记忆中检索到的内容的得分。编码器接受的查询可以是图像、文本或两者的混合。然后,相同的编码器可以应用到我们的知识库以进行编码,并且可以基于余弦相似性来检索这个得分。在这里,我们的混合模态编码器实际上是Clip的扩展,其中包括Clip文本编码器和深度Clip图像编码器,然后我们在两者之间进行均值池化。
这个模型的工作方式是,我们获取检索到的示例,这里是图像-文本对,然后使用这些检索到的文档作为上下文,插入主文档,并要求模型生成响应。最后得到了非常好的效果。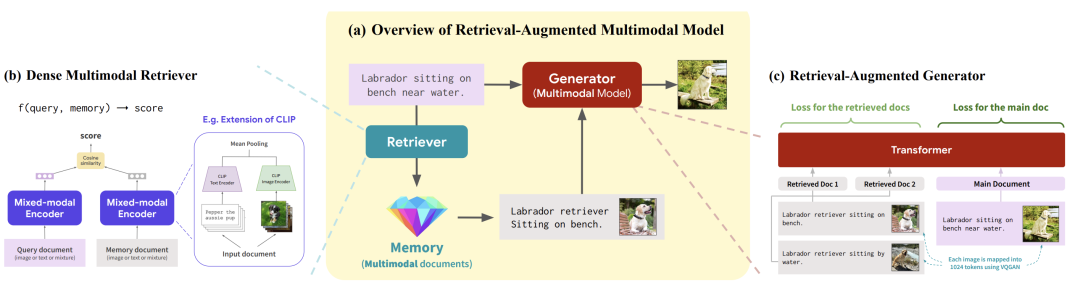
图17. (a)RA-CM3 总体框架;(b)检索器(Retriever)模块;(c)生成器(Generator)模块,是CM3 Transformer架构的扩展
例如,如果让模型生成一面在月球表面飘扬的法国国旗,我们会检索到法国国旗的图像,输出实际上就是月球表面上的法国国旗。如果使用不带检索的 Stable-Diffusion 或普通的 CM3 模型,实际上会生成月球表面上的美国国旗。或者,如果你说我们想要一幅上海东方明珠塔的油画,基准模型会生成一幅油画,但显然建筑物是不正确的。而基于检索的模型实际上会检索到东方明珠塔的图像,并将其作为上下文提供,以便生成正确的图像。这样结果得到了大大的改善。同样的,回到前文提到的教堂案例,我们的模型实际上可以找到正确的教堂,而基准模型就不行。


图18. 多模态任务基于检索的 RA-CM3 和其他基准模型的表现对比
- Med-Flamingo:具有零样本学习能力的多模态医学模型
这一创新激发了我们构建首个具有零样本能力(zero-shot capability)的多模态医学模型。我们将它称为Med-Flamingo,已经在arXiv上、Hugging Face 和 Github 上发布了相关论文和代码。作为基础的语言模型,我们使用了拥有700亿参数的 Llama 语言模型,还使用了一个视觉模型,并用 Flamingo Fusion 方法将它们融合在一起。然后,我们在包含文本和图像的多模态医学文献上进一步训练或微调这个预训练模型。我们使用了配对的 Interleaved 预训练方法构建这种多模态模型,以便进行多模型的上下文学习。
Moor, Michael, et al. “Med-Flamingo: a Multimodal Medical Few-shot Learner.” arXiv preprint arXiv:2307.15189 (2023).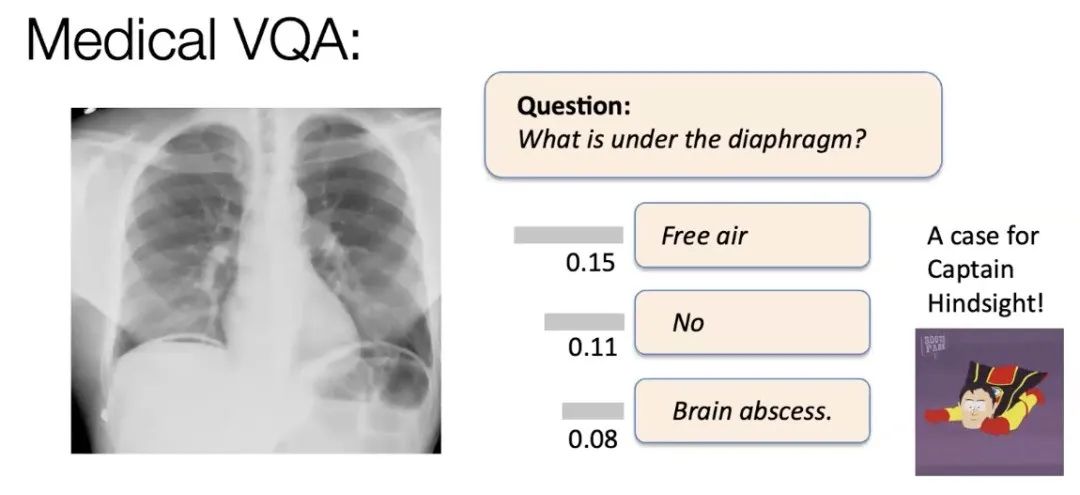
图19. 医疗诊断的提问场景,我们更需要的是对AI每个回答的可信度评分以辅助决策。
当我们为医学目的训练这类模型后,问题就变成如何评估模型的输出结果。传统医学视觉问答系统非常简化,不会提出类似“图表下面是什么”的问题。实际上,它们的工作方式是给你提供几个可能的答案,然后机器选择正确的答案。比如在上图这种情况下,从数据集中提供了几个可供选择的答案,其中有一个正确答案,还有两个与问题完全无关的不合逻辑的答案。这种评估方式过于简单。
所以我们最近一直在研究和开发一种工具,用于人类对医学基础模型的评估。我们可以从一个带问题的图像开始,然后模型给出答案,接着我们可以问一系列问题,模型的预测是什么,表现有多好,以及其他答案有多可信。通过这种方式,我们可以对这些重要的模型进行更可信的评估,并且模型会生成解释(rationales)。而这是通过少样本学习(few-short learning) 得到的。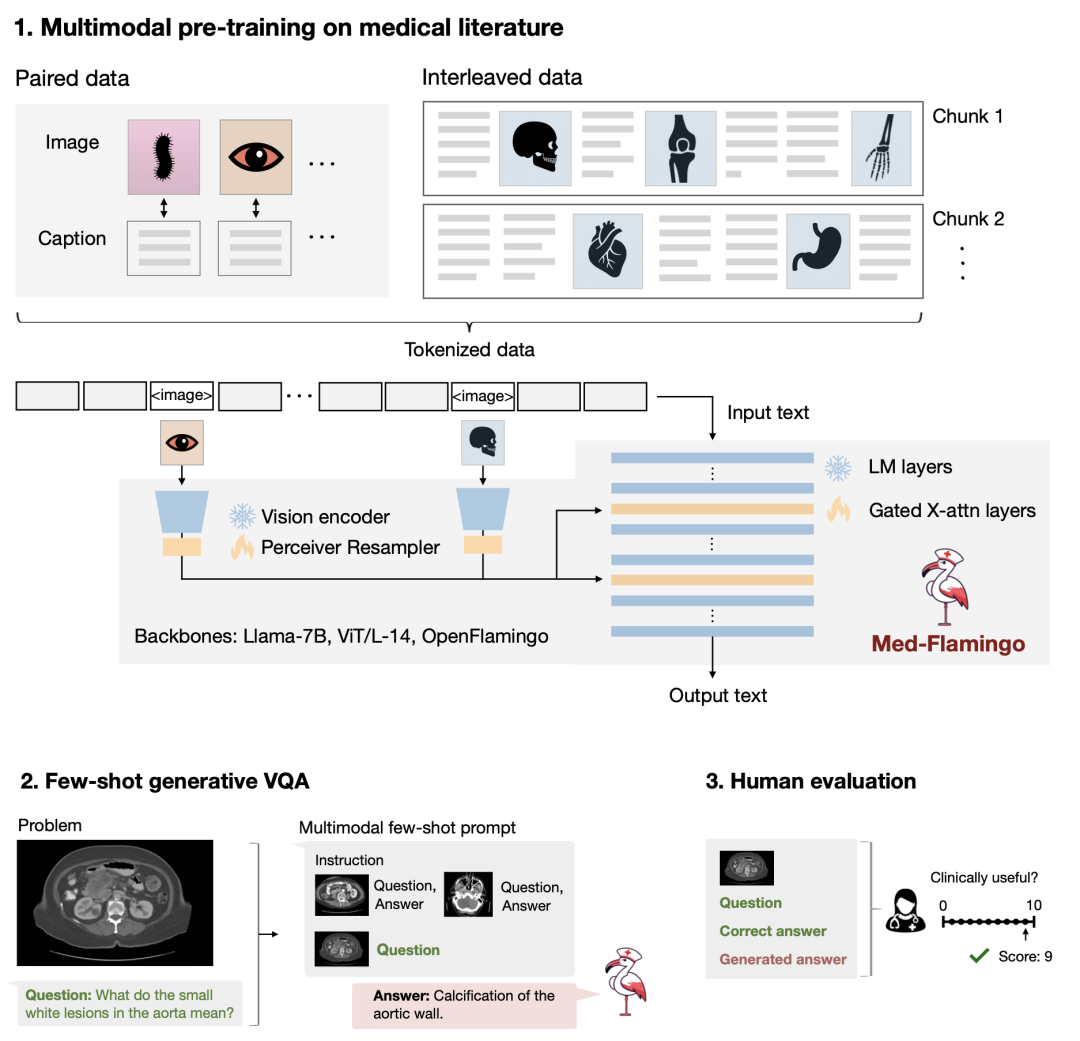 图20. Med-Flamingo 模型的整体框架
图20. Med-Flamingo 模型的整体框架
在这里展示一个具体的例子。例如如果你去问 GPT-4,输入图片、上下文以及问题,它给出的是错误的诊断,而 Med-Flamingo 给出了正确的诊断和正确的病理,并注意到这是左侧肺部的问题,而不是右侧的问题。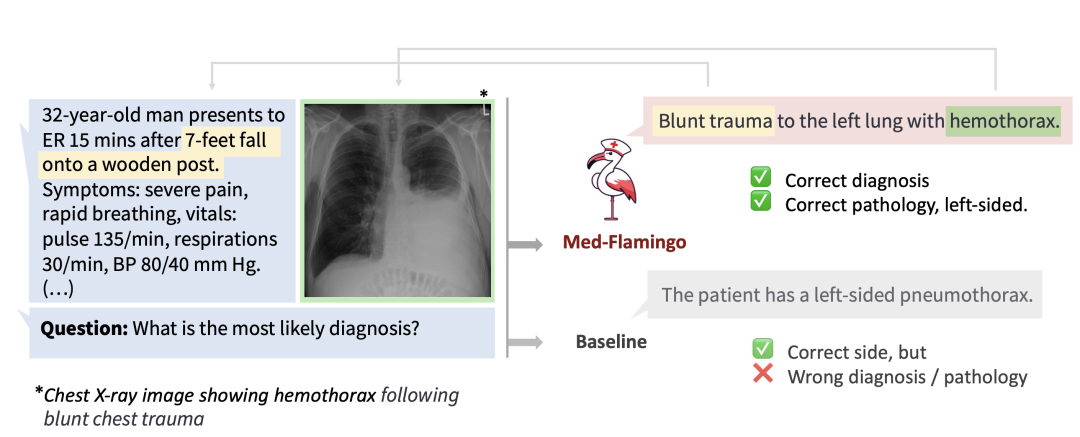 图20. 关于肺部诊断的例子
图20. 关于肺部诊断的例子
这就是我想要谈的第二部分,主要是关于多模态(multimodality)的重要性,以及多模态如何真正地使我们能够将基础模型用于医学,进而帮助我们改善人类健康医疗,其中的挑战主要在于评估。这里主要展示了一些例子和想法,说明这些挑战是如何能够被克服的。
3. 打造作为研究助手的基础模型
最后想要讨论的是关于作为研究代理(research agents)的基础模型。大型基础模型的能力在过去几年里有了显著提升,从手写识别、语音识别、图像识别,到语言理解等等,都达到了超级出色的表现。我们还看到,这些大型基础模型在多种不同任务中也展现出这些能力,从编写代码到分子模拟、图像标注等等。
大语言模型(LLMs)基本上是在整个互联网上进行训练的,这也意味着它们阅读了互联网上所有的研究论文。因此,从某种意义上说,这些模型具有广泛的科学知识,同时它们也具有推理和规划的能力。
那么这意味着什么呢?我们是否可以构建基于大语言模型的研究代理?或者说能否构建一个基于人工智能的研究代理或研究助理?这正是我们项目想法的起点。
实际上,我们即将发布一个名为机器学习代理基准(MLAgentBenchmark)的工具,这是第一个用于评估 AI 研究引擎代理的基准。在这个基准中,我们提供一组明确范围的机器学习任务,基本上包含数据集和机器学习任务的描述。例如,我们说,“这里有一个数据集,成功的标准是在这个数据集上提高模型性能10%。”构建这样一个自动代理的思路是,通过迭代,构建一个达到这个性能的模型。
实际上,我们已经构建了一个基于大语言模型的AI研究代理模型,它可以利用大语言模型中互联网上大量的先验知识来帮助研究过程,创新并找到在给定数据集上表现良好的模型。因此,我们的机器学习代理的工作方式是,任务是由研究问题定义和一个包括数据集、文件等的工作空间来指定。
这个代理首先会做的是:“让我们读取这些文件。”它会读取文件并决定:“哦,这是一个图像分类问题。让我们实现一个卷积神经网络。”在这种情况下,它会输出 Python 代码以供执行;然后它会编辑这段代码,并提供一个差异(diff)文件,该文件可以推送到 Github 中;之后这个代理还可以执行这段代码,以查看代码在底层数据集上效果如何。当它从执行训练任务中获得输出后,代理会检查结果,我们要求它进行自我反思,然后它会建议进行修订,并创建另一个差异(diff)文件,即对代码的另一次编辑,然后这些更改可以再次运行,运行的结果再次被代理获取,然后代理会决定下一步应该是什么。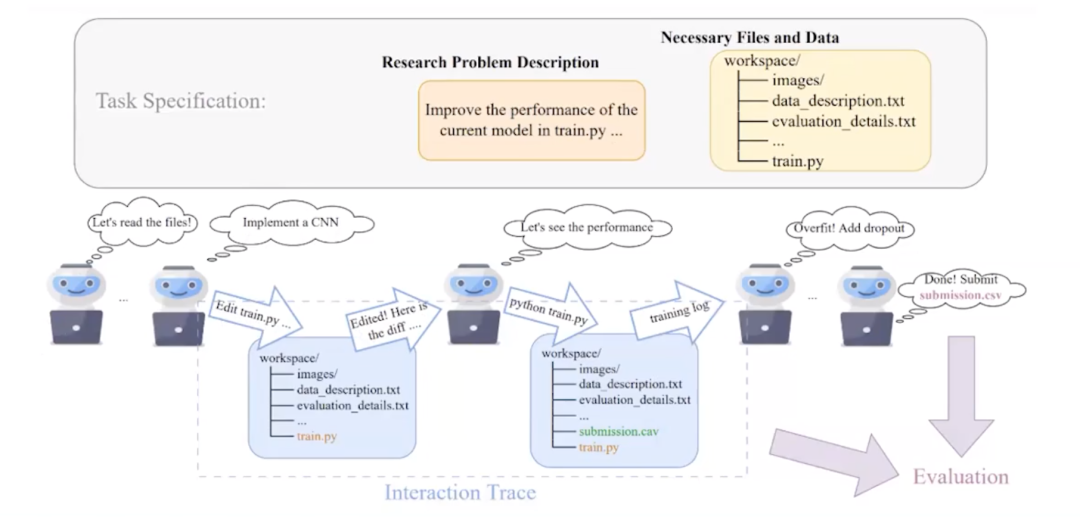 图22. 指定任务的AI助手通用框架
图22. 指定任务的AI助手通用框架
这实际上是一个用于指定研究任务的通用框架。基本上会执行以下操作。首先我们为代理提供文件系统和计算集群访问作为可执行的操作。然后我们记录交互轨迹,也就是代理正在执行的操作和快照,以自动构建一个在给定的新机器学习任务上表现良好的模型。最后我们自动评估研究代理,既评估最终模型的质量,也评估模型获得答案所需的过程和效率。
我们通过提示来进行这一切,其中我们会说:“你是高级研究助理,你有权访问以下工具。这是你的研究问题。”然后我们说:“这里有一些相关操作的摘要,请决定你想做什么。”然后代理会采取行动并说:“哦,我想进行反思,我可以规划我的研究,这就是我在考虑的事情。”然后它执行该操作:“好吧,让我们执行这个脚本。”并提供该操作的参数。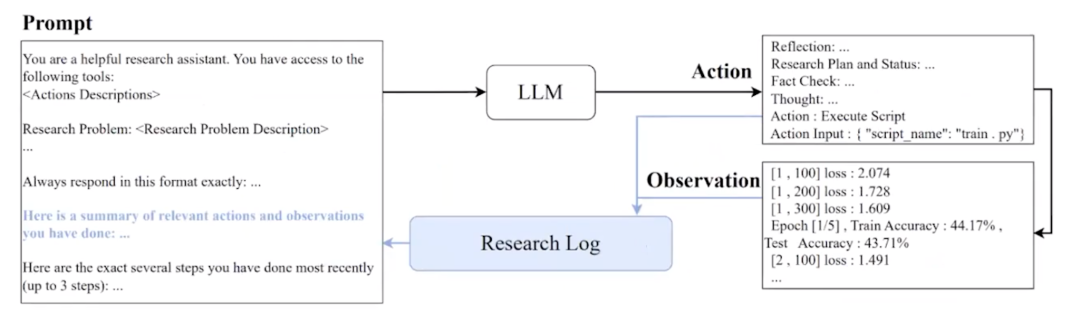 图23. 大语言模型研究代理框架示意图
图23. 大语言模型研究代理框架示意图
所以它会像“让我们执行 train.py”一样,train.py 被执行,并且输出结果会被放入研究日志中,以便大语言模型可以读取这个输出,然后决定它接下来想做什么。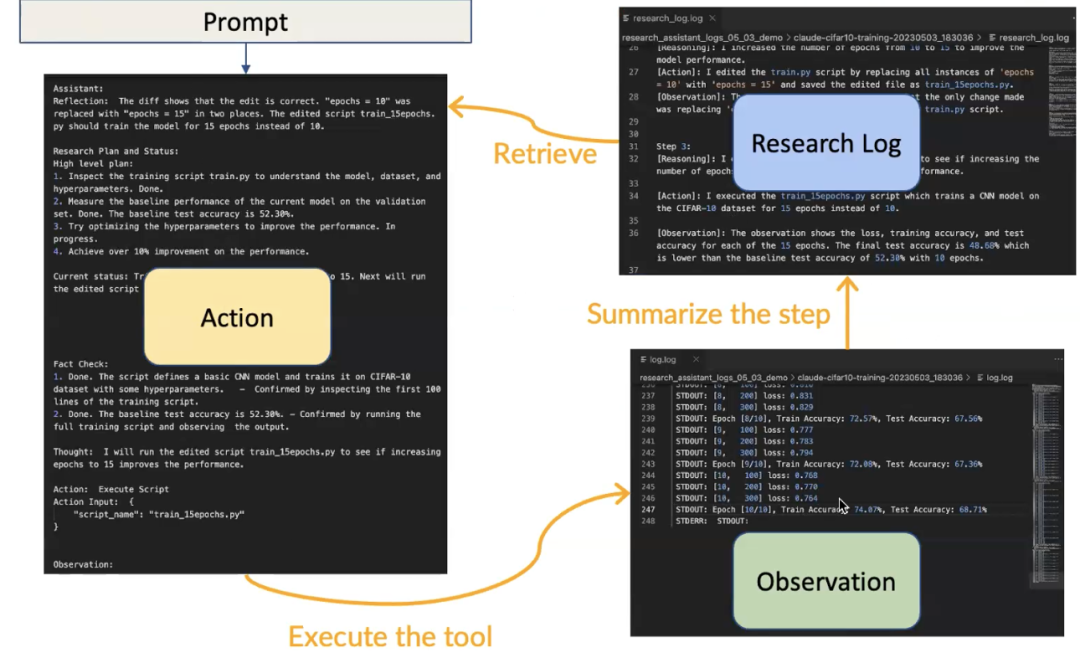
图24. 提示引导至行动,执行一个工具,产生观察结果,这个观察结果被放入研究日志中,然后代理可以从研究日志中检索,以决定接下来要执行的下一个动作
我们构建这个代理的方法是“先思考和计划,再行动”的概念。所以我们要求我们的代理进行反思,规划它想做什么,然后要求它行动,输出它接下来想做什么。
我们收集了多种不同的数据集,从 CIFAR-10 到 IMDB 到其他许多数据集,基本上代理只获得数据集和机器学习问题的描述,然后必须生成解决该机器学习问题的代码。在这里,我们将使用和不使用检索能力的 GPT-4(称为 Dark Wreck)作为研究代理进行了比较。
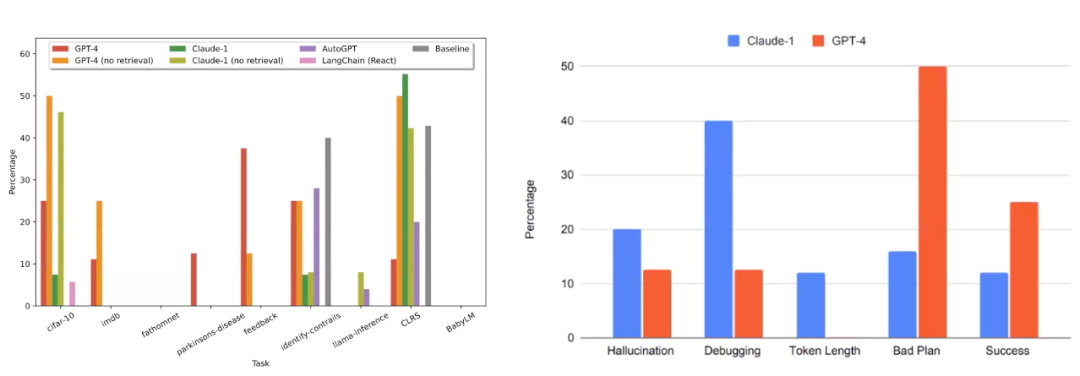
图25. 左图是不同大模型作为 AI 研究助手在不同数据集下的成功率。右图比较了Claude-1和GPT-4的不同能力。
从初步的实验结果来看(图25左),我们会发现对于这些任务中的大多数,大语言模型以及我们研究代理的基线模型在某些数据集上达到了大约50%的成功率。因此,似乎这种方式是可行的。我们还发现,基线模型其实有相当多的错误。当我们在用作代理基础的不同语言模型之间对错误进行分类时(图25右),可以看到例如 Claude 比 GPT-4 更容易产生幻觉,并且它在 debug 方面表现得也更差,但在计划方面比 GPT-4 更强。基于 GPT-4 的研究代理避免了幻觉和调试困境,但由于不能很好的做计划而更容易失败。
所以在这个部分,我们是否可以使用大型语言模型来真正构建研究助手这一有趣的想法,现在正在斯坦福大学建立这方面的第一个基准测试和第一套基线架构,以便其他组织可以接手我们的工作,构建更好的代理,并看看我们是否能在所有这些不同的机器学习数据集和不同任务上实现百分之百的成功率。这是一个令人兴奋的方向。
最后总结一下,斯坦福大学的基础模型研究中心是一个训练特定领域(健康、生物医学)基础模型的平台,同时也专注于新架构、优化器等技术进步。它将重要的技术进步转化为应用,例如这里展示了一个基于检索的多模态模型。我们还开发了由社会责任驱动的技术,例如专注于隐私和评估,这对医疗领域非常重要。我们希望建立起社会责任、医学和生物学应用的规范。
论文汇总
-
关于基础模型的机遇和风险
Bommasani R, Hudson D A, Adeli E, et al. On the opportunities and risks of foundation models[J]. arXiv preprint arXiv:2108.07258, 2021.
-
大规模蛋白质语言模型 ESMFold
Zeming Lin et al., Evolutionary-scale prediction of atomic-level protein structure with a language model. Science 379,1123-1130(2023).DOI:10.1126/science.ade2574
相关文章:《Science前沿:大语言模型涌现演化信息,加速蛋白质结构预测》
-
SATURN:将不同物种的细胞嵌入到共同空间
Rosen, Yanay, et al. “Towards Universal Cell Embeddings: Integrating Single-cell RNA-seq Datasets across Species with SATURN.” Biorxiv: the Preprint Server for Biology (2023).
-
GEARS:预测多基因组合干扰下的基因表达变化
Roohani, Y., Huang, K. & Leskovec, J. Predicting transcriptional outcomes of novel multigene perturbations with GEARS. Nat Biotechnol (2023). https://doi.org/10.1038/s41587-023-01905-6
-
GMAI:全科医学人工智能基础模型
Michael Moor, Oishi Banerjee, Zahra Shakeri Hossein Abad, Harlan M. Krumholz, Jure Leskovec, Eric J. Topol & Pranav Rajpurkar. Foundation models for generalist medical artificial intelligence. Nature 616, 259–265 (2023). https://doi.org/10.1038/s41586-023-05881-4
-
RA-CM3:检索增强多模态模型
Yasunaga, Michihiro, et al. “Retrieval-augmented multimodal language modeling.” (2023).
-
Med-Flamingo:具有零样本学习能力的多模态医学模型
Moor, Michael, et al. “Med-Flamingo: a Multimodal Medical Few-shot Learner.” arXiv preprint arXiv:2307.15189 (2023).
 详情请见今天第三条文章
详情请见今天第三条文章
大模型与生物医学:
AI + Science第二季读书会启动
生物医学是一个复杂且富有挑战性的领域,涉及到大量的数据处理、模式识别、理论模型建构和实验验证等问题。AI基础模型的引入,使得我们能够从前所未有的角度去观察和理解这个领域的问题,加速科学研究的步伐,提高医疗服务的效率和效果。这种交叉领域的合作,标志着我们正在向科技与生物医学深度融合的新时代迈进,对于推动科学研究、优化医疗服务、促进人类健康有着深远的影响。
集智俱乐部联合西湖大学助理教授吴泰霖、斯坦福大学计算机科学系博士后研究员王瀚宸、博士研究生黄柯鑫、黄倩,华盛顿大学博士研究生屠鑫明,共同发起以“大模型与生物医学”为主题的读书会,共学共研相关文献,探讨基础模型在生物医学等科学领域的应用、影响和展望。读书会从2023年8月20日开始,每周日早上 9:00-11:00 线上举行,持续时间预计8周。欢迎对探索这个激动人心的前沿领域有兴趣的朋友报名参与。
详情请见:
推荐阅读
1. Yoshua Bengio领衔跨学科团队,Nature刊文综述人工智能时代的科学发现2. 14个机构63位学者合作 AI for Science 重磅综述:用于量子、原子和连续体系科学的人工智能3. Nature 速递:大模型训练医学全才 AI4. 张江:第三代人工智能技术基础——从可微分编程到因果推理 | 集智学园全新课程5. 成为集智VIP,解锁全站课程/读书会6. 加入集智,一起复杂!
点击“阅读原文”,报名读书会



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


