由中国药学会支持,清华大学药学院、百度和临港实验室联合主办的首届全球AI药物研发大赛落幕。此次比赛共吸引来自全球的1105人和878个团队参加。微软研究院王童研究员带领的ViSNet-Drug团队,凭借其团队自主开发的AI2BMD[1]高精度动力学模拟系统、ViSNet[2]三维分子结构建模图神经网络和蛋白质全构象数据集AIMD-Chig[3]在三轮比赛中均排名第一并获得总冠军!
新冠爆发以来已经在全球造成巨大危害和经济损失,随着对新冠病毒的致病机理研究逐渐深入,如何设计特效药对新冠治疗已经成为研究的热点话题。在本次比赛中,主办方围绕新冠病毒的小分子药物设计展开命题,通过两轮预测比赛和决赛答辩评选出最具创新性的AI药物研发设计方案。其中,初赛围绕对新冠病毒的主蛋白酶展开。主蛋白酶(M pro)在新冠病毒感染人体过程中发挥关键作用,负责对病毒进行剪切产生感染前体并在病毒复制中发挥作用。作为潜在治疗靶点,抑制主蛋白酶的生物活性能够有效干扰病毒的复制过程,为新冠药物设计和治疗提供关键思路。复赛围绕小分子药物在Caco细胞上能够抑制新冠病毒的复制而展开。

决赛答辩现场
在此次大赛中,王童研究员团队提出全新的新冠小分子药物解决方案:通过AI2BMD高精度的分子动力学模拟有效区分正负样本小分子与主蛋白酶靶点之间的结合自由能,利用AIMD-Chig蛋白全构象数据集对ViSNet模型进行预训练,最终通过对预训练的ViSNet网络进行fine tuning,得到高准确度的预测结果。
AI2BMD模拟系统是一套由深度神经网络驱动的分子动力学模拟系统(https://www.biorxiv.org/content/10.1101/2023.07.12.548519v1 )。一方面,它实现了对从数百原子到上万个原子的各种蛋白质的能量和原子受力量子模拟精度的精确计算。另一方面,相比较于量子模拟,例如密度泛函理论DFT,AI2BMD节省了数个数量级的计算时间。AI2BMD通过对蛋白质进行数百万步的模拟,展现了对蛋白质构象空间探索、蛋白质动力学性质分析、自由能计算等各个方面的卓越能力。相比于利用传统分子对接无法很好地区分正负样本结合自由能的问题,AI2BMD的计算展现出明显优势。
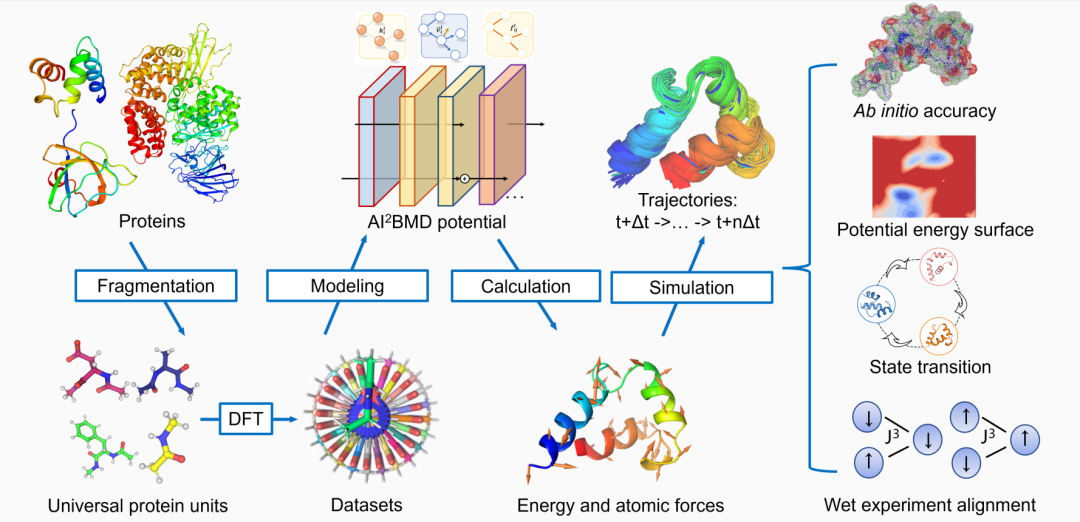
AI2BMD模拟系统流程图
ViSNet是王童研究员团队提出的通用分子三维结构建模网络(https://arxiv.org/abs/2210.16518 )。该网络通过RGC模块将分子三维结构中的距离、角度、二面角等信息转成模型中点和边之间的操作,并将计算复杂度降低到线性水平。ViSNet提出全新的向量-标量交互式信息传递过程ViS-MP,显著增强了对分子三维建构几何信息的提取和建模能力,并在MD17、MD22、QM9等多个分子性质预测数据集上取得最优的预测结果。在比赛中,ViSNet通过对药物小分子的建模,有效提取了分子的几何结构信息,取得了显著比其他分子网络更高的预测精度。
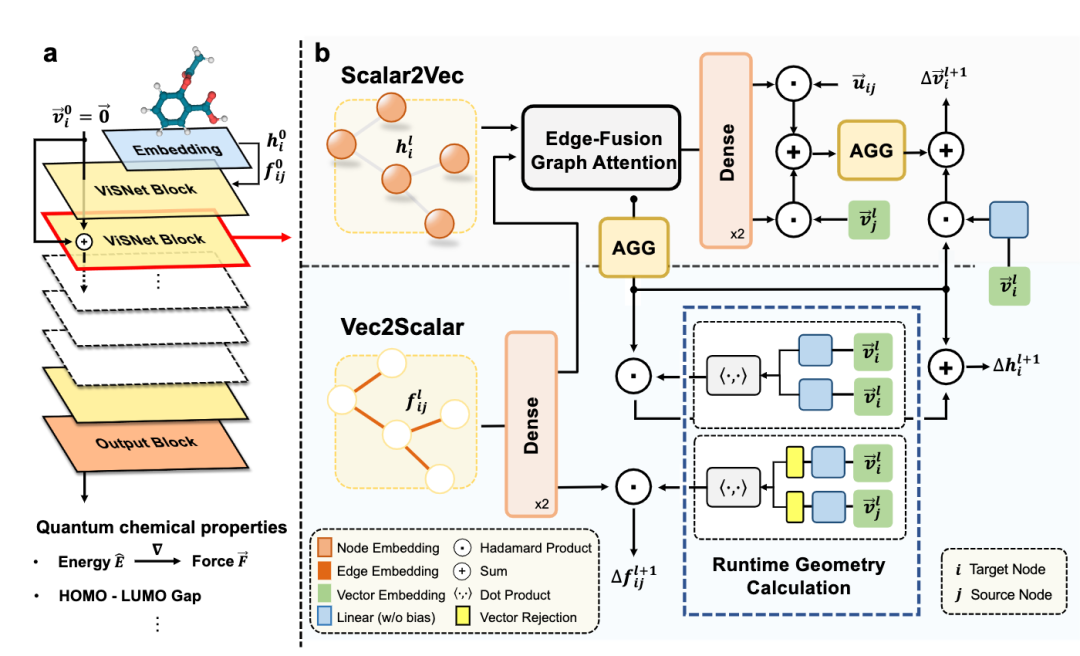
ViSNet模型架构图
此外,ViSNet-Drug团队还利用了其构建的蛋白质全构象高精度数据集AIMD-Chig(https://www.nature.com/articles/s41597-023-02465-9 ;https://bioengineeringcommunity.nature.com/posts/aimd-chig-exploring-the-conformational-space-of-proteins-at-dft-level )对ViSNet模型进行预训练,在比赛训练集十分有限的情况下,显著提高了ViSNet模型对分子结构的识别和建模能力。依靠AI2BMD模拟系统、ViSNet神经网络和AIMD-Chig数据集,王童研究员团队在对M pro和Caco细胞的预测准确率显著高于第二名团队,并以99.60分的成绩获得总冠军(亚军、季军分别为90.76分和85.31分)。

AIMD-Chig高精度蛋白质构象数据集
此次大赛的举办,为新冠小分子药物的研发提供了潜在的解决方案,也为AI如何更好地助力药物研发带来更多思考。仅通过单一模型预测往往效果不佳,结合分子动力学模拟、高质量的数据集、更为强大的深度学习模型以及对靶点和药物的理解,多管齐下,将为AI药物研发提供更多的可能。
参考资料
[1] Wang T, He X, Li M, et al. AI2BMD: efficient characterization of protein dynamics with ab initio accuracy. bioRxiv, 2023: 2023.07. 12.548519.
[2] Wang Y, Li S, Wang T, et al. ViSNet: a scalable and accurate geometric deep learning potential for molecular dynamics simulation. arXiv preprint arXiv:2210.16518, 2022.
[3] Wang T, He X, Li M, et al. AIMD-Chig: Exploring the conformational space of a 166-atom protein Chignolin with ab initio molecular dynamics. Sci Data 10, 549 (2023).



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


