表征蛋白质可以揭示疾病的机理,包括减缓或逆转疾病的方法,而创造蛋白质则可以开发出全新的药物和疗法。然而,从计算和人力资源的角度来看,目前在实验室设计蛋白质的过程成本高昂。它需要提出一种能在体内执行特定任务的蛋白质结构,然后找到一种可能“折叠”到该结构中的蛋白质序列。
日前,微软开源了一种通用的扩散框架 EvoDiff,它将进化尺度数据与扩散模型的独特调节能力相结合,可在序列空间中可控地生成蛋白质。EvoDiff 能生成高保真、多样化、结构上合理的蛋白质,涵盖自然序列和功能空间。重要的是,EvoDiff 可以生成基于结构的模型无法生成的蛋白质,如那些具有无序区域的蛋白质,同时还能保持设计功能结构主题支架的能力,这证明了基于序列公式的通用性。
Protein generation with evolutionary diffusion: sequence is all you need
文献来源:https://doi.org/10.1101/2023.09.11.556673
代码:https://github.com/microsoft/evodiff.
内容:深度生成模型是用于在计算机上设计新型蛋白质的越来越强大的工具。最近,一种名为扩散模型的生成模型家族已经证明了生成与自然界中任何实际蛋白质不同的生物学上可信蛋白质的能力,从而在从头开始蛋白质设计方面提供了前所未有的能力和控制。然而,目前最先进的模型生成的蛋白质结构,受限于训练数据,并使得生成局限于蛋白质设计空间的一个小而有偏的子集。在这里,作者介绍了一个通用的扩散框架,EvoDiff,它将进化规模的数据与扩散模型的独特调节能力相结合,用于序列空间中可控的蛋白质生成。EvoDiff生成高保真度、多样性和结构上合理的蛋白质,覆盖了天然序列和功能空间。至关重要的是,EvoDiff可以生成基于结构的模型无法获得的蛋白质,例如那些具有无序区域的蛋白质,同时保持设计功能结构基序支架的能力,这表明作者的序列为基础的方案的通用性。作者预计EvoDiff将扩展蛋白质工程的能力,超越结构功能范式,迈向可编程的序列优先设计。
1.背景介绍
进化已经产生了多种能够精确地调节细胞过程的功能蛋白。近年来深层生成模型的通过学习这种多样性用于生成既有效和新颖的蛋白质,然后通过编辑功能来解决一些真实的挑战,如有针对性的治疗的快速发展和疫苗或工程酶降解工业废物(图1)。扩散模型为新蛋白质的生成建模提供了一个特别强大的框架–它们可以生成高多样性的样本,并可以给定各种各样的输入或设计目标。事实上,今天在生物学上最可信的计算设计蛋白质的例子来自于蛋白质结构的扩散模型。
本文中,作者将进化规模大小的数据集与扩散模型相结合,开发了一个强大的新的生成建模框架,并称之为EvoDiff,用于仅从序列数据进行可控的蛋白质设计(图1)。鉴于蛋白质的自然框架是在一种氨基酸语言上的离散标记序列,本文使用了一个离散扩散框架:在一个正向过程通过改变蛋白质序列的氨基酸来迭代地破坏蛋白质序列,然后在一个学习到的由神经网络参数化的反向过程,预测每次迭代中发生的变化 (图1B)。相反的过程可以从随机噪声开始生成新的蛋白质序列(图1C)。重要的是,EvoDiff的离散扩散公式在数学上不同于以前用于蛋白质结构设计的连续扩散公式。除了单一蛋白质序列的进化规模数据集外,多序列比对(MSAs)通过揭示相关蛋白质组的氨基酸序列的保守和变异模式,内在地捕获了进化关系。因此,作者还建立了在MSA上训练的离散扩散模型,以利用这一额外的进化信息层来生成新的单个序列(图1C-D)。
作者评估了序列和MSA模型—EvoDiff-Seq和EvoDiff-MSA—在一系列生成任务中,以证明它们在可控蛋白质设计方面的能力(图1D)。作者首先展示了EvoDiff-Seq无条件地生成高质量、多样化的蛋白质,从而捕获蛋白质序列、结构和功能空间的自然分布。利用EvoDiff-MSA,作者实现了进化引导的新序列设计,基于进化相关但不同的蛋白质对齐。最后,通过利用基于扩散的建模框架的调节能力及其接地在一个通用的设计空间,作者证明EvoDiff可以可靠地生成蛋白质,直接克服基于结构的关键限制生成模型,生成支架功能结构图案没有任何明确的结构信息。
EvoDiff是第一个基于进化规模的蛋白质序列数据进行训练的蛋白质设计的生成扩散模型。在本文,作者研究了两种在离散数据模式上扩散的正向过程,以确定哪一种是最有效的(图1B)。其中,EvoDiff–OADM为顺序不可知的自回归扩散模型,EvoDiff-D3PM为离散去噪扩散概率模型。
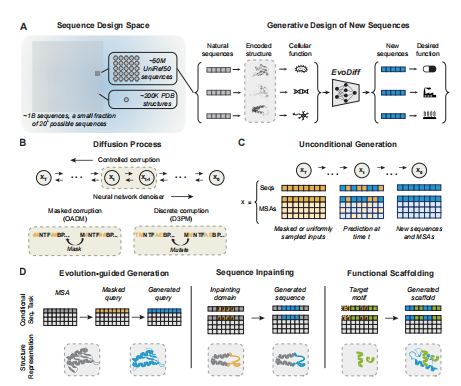
图1 具有进化扩散特性的蛋白质序列生成。(A)(左)进化法已经取样了一小部分可能的蛋白质序列。更少的蛋白质的实验结构。(右)EvoDiff是一种基于自然蛋白质序列训练的生成式离散扩散模型。从EvoDiff取样产生新的蛋白质序列,可能执行所需的功能。(B)离散扩散模型由受控的破坏和学习过的去噪过程组成。在隐藏损坏过程中,输入令牌以顺序无关的方式屏蔽(左下)。在离散的破坏过程中,输入通过一个由捕获氨基酸突变频率的转移矩阵控制的马尔可夫过程被破坏(右下)。(C) EvoDiff可以无条件地生成蛋白质序列或MSA。从屏蔽或均匀采样的输入xT开始,EvoDiff通过反向破坏过程,迭代去噪xt到真实的序列或MSA,生成新的序列或MSA x0。D)可控蛋白设计与EvoDiff,通过调节MSAs编码的进化信息(左);从序列的掩蔽部分绘制功能域(中间);或没有明确结构信息的支架结构基序(右)。
2.结果
2.1生成的序列的结构合理性
接下来,作者研究了EvoDiff是否能够产生新的单独有效和结构合理的蛋白质序列。为了评估这一点,他们开发了一个工作流,评估由EvoDiff生成的序列的可折叠性和自一致性(图2A)。他们从每个EvoDiff序列模型中生成了1000个序列,其长度来自于训练集中长度的经验分布。他们将EvoDiff的生成与由从左到右的自回归语言模型(LRAR)生成的序列进行了比较,该序列具有与EvoDiff相同的架构和训练集,以及由蛋白质掩蔽语言模型如ESM-2生成的序列。作者通过使用OmegaFold预测其相应的结构,并计算整个结构的平均预测局部距离差检验(pLDDT)来评估单个序列的可折叠性(图2B)。除了整个蛋白质的平均pLDDT外,我们观察到pLDDT评分在一个蛋白质序列中可能存在显著差异。值得注意的是,虽然pLDDT得分超过70通常被认为表明较高的预测置信度,但较低的pLDDT得分可能与蛋白质的内在无序区域(IDRs)相一致,这在许多天然蛋白质中都有存在。作为结构合理性的另一个度量,作者通过使用逆折叠算法ESM-IF重新设计每个预测结构,并对原始生成的序列计算困惑,计算了自一致性复杂度(图2A,C)。考虑到ESM-IF和EvoDiff都是在UniRef50数据上训练的,来自EvoDiff验证集的序列可能与ESM-IF训练集中的序列重叠;因此,我们使用蛋白质MPNN进行了相同的自一致性评估,它没有在UniRef50上进行逆折叠。虽然没有生成模型接近于可折叠性和自一致性的测试集值,但EvoDiff-OADM优于EvoDiff-D3PM,并且随着模型大小的增加而有所改进(图2B-D)。因此,作者选择了640m参数的EvoDiff-OADM模型进行下游分析,并随后将其称为EvoDiff-Seq。对从EvoDiff-Seq中采样的4个不同序列长度的结构可信序列的代表性例子的分析说明了它们来自训练集中序列的结构合理性和新颖性,表明EvoDiff生成的蛋白质序列是单独有效的(图2E)
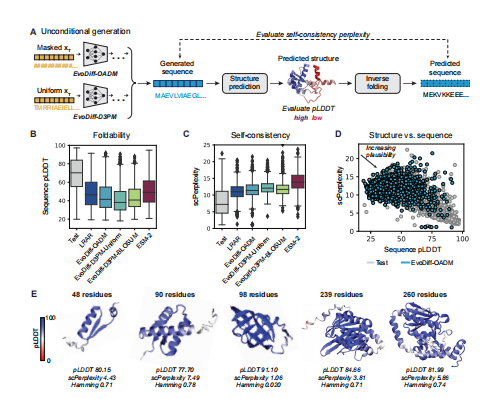
图2 EvoDiff生成了真实的和结构上可信的蛋白质序列。(A)用于评估由EvoDiff序列模型生成的序列的可折叠性和自一致性的工作流。B-C)可折叠性分布,通过预测结构(B)的序列pLDDT测量,自一致性,来自测试集、EvoDiff模型和基线(每个模型n=1000个序列;盒图显示中位数和四分位数范围)。(D)序列测试集(灰色pLDDT与n=1000)和640m参数OADM模型EvoDiff-Seq(蓝色,n=1000)的序列。(E)来自640m参数OADM模型EvoDiff-Seq的代表性结构可信代的预测结构和指标。
2.2所生成的序列分布的生物学特性
在证明了EvoDiff的可进行设计之后,作者接下来评估了设计的蛋白质序列的分布对自然蛋白质空间的影响。理想情况下,生成的序列应该捕获序列、结构和功能属性的自然分布,同时仍然具有彼此之间和与自然序列之间的差异。无论是定性还是定量,EvoDiff-Seq生成的蛋白质比从最先进的蛋白质屏蔽语言模型(ESM-2)采样或从最先进的结构扩散模型(RF扩散)生成的结构预测序列更好地再现自然序列和功能多样性(图3A)。为了评估生成序列中结构属性的分布,作者计算了生成序列和自然序列中每个残基的三态二级结构,并定量地比较了结构属性的结果分布与测试集的分布(图 3B)。EvoDiff-Seq产生的链和无序区域的比例与自然序列更相似,而ESM-2和RF扩散都产生了在螺旋中富集的蛋白质(图3B)。
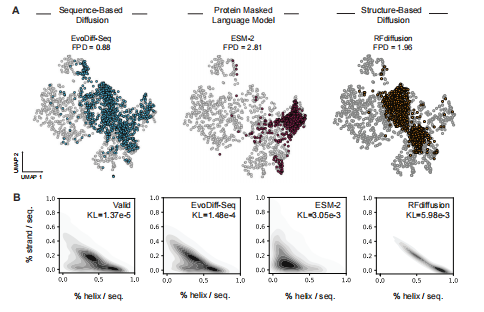
图3 生成的蛋白质序列捕获了蛋白质功能和结构特征的自然分布。ProtT5嵌入的(A) UMAP,用FPD注释,测试集的自然序列(灰色,n=1000)和EvoDiff-Seq(蓝色,n=1000)和ESM-2(红色,n=1000)生成的序列,以及射频扩散结构的(A)UMAP(橙色,n=1000)推断序列。(B)基于DSSP 3预测(每个模型或验证集的n=1000个样本),并标注了相对于测试集的螺旋和链结构特征的多元分布。
2.3条件设计
EvoDiff的OADM扩散框架通过固定一些子序列和替换剩余的子序列,引入了一种条件序列生成的自然方法。因为该模型被训练成生成具有任意解码顺序的蛋白质,所以这很容易通过简单地屏蔽和解码所需的部分来完成。作者将EvoDiff的能力应用于三种情况下的条件蛋白质设计:调节msa编码的进化信息、绘制功能域和支架功能结构基序(图1D)。具体见图4,图5。总得来说,该方法表现不错。
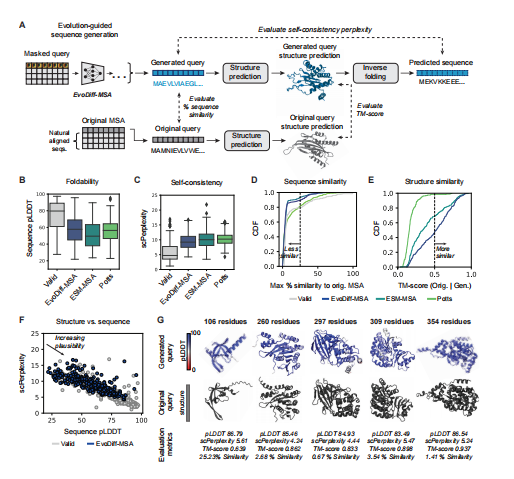
图4 EvoDiff-MSA支持进化引导的序列生成。(A)EvoDiff-MSA通过查询组件的扩散生成一个新的序列。评估世代的多样性和自一致性,以及其预测结构的质量和一致性。(B-E)来自验证集、EvoDiff-MSA、ESM-MSA和Potts模型(每个模型n=250个序列)的pLDDT (B)、精确度(C)、序列相似性(25%)和=分数(E;0.5)的分布;盒图显示中位数和四分位数范围)。(F)验证集(灰色,=250)和EvoDiff-MSA(蓝色,=250)序列的pLDDTvssc=复杂性。(G)从EvoDiff-MSA中获得的结构上可信的世代的预测结构和指标。
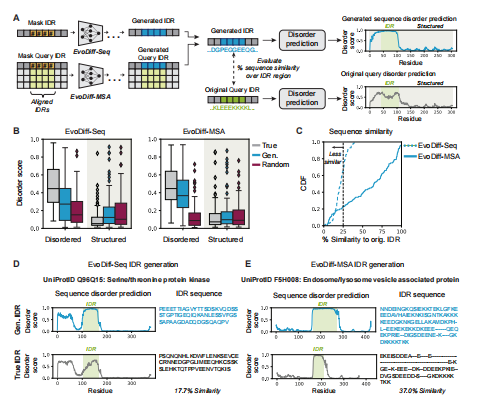
图5 EvoDiff生成了本质上无序的区域。(A)通过在查询序列中添加无序残基,从EvoDiff-Seq或EvoDiff-MSA中生成一个新的IDR序列。然后使用DR-BERT来预测原始序列和再生序列的无序评分。(B)真(灰色)、插图(蓝色)和随机采样(红色)idr(每个条件n=100个序列在无序和结构区域上的无序得分分布;箱形图显示中位数和四分位数范围)。(C)从EvoDiff-Seq(蓝色,虚线)和EvoDiff-MSA(蓝色,固体)生成的序列相似性相对于原始IDR的分布(n=100;虚线为25%)。(D-E)从EvoDiff-Seq (D)和EvoDiff-MSA (E)中生成的具有代表性的(上行)和真(下行)IDRs的预测障碍得分和相应的序列
3.总结
作者提出了EvoDiff,一种扩散建模框架,能够生成高保真度、多样化和新的蛋白质,并可选择根据序列约束进行条件调节。由于EvoDiff在通用的蛋白质设计空间中运行,它可以无条件地取样不同结构可信的蛋白质,产生本质上无序的区域,并仅使用序列信息进行支架结构基序,挑战基于结构的蛋白质设计的范式。
EvoDiff是第一个证明扩散生成建模在进化尺度的蛋白质序列空间上的能力的深度学习框架。与之前在蛋白质结构或序列上训练扩散模型的尝试不同,EvoDiff是在所有自然序列的大样本上训练的,而不是在较小的蛋白质结构数据集或来自特定蛋白质家族的序列数据上训练的。以往在全局序列空间上训练的蛋白质生成模型要么是从左到右的自回归(LRAR)模型,要么是掩蔽语言模型(MLMs)。EvoDiff的OADM训练任务概括了LRAR和MLM的训练任务。具体来说,OADM设置通过考虑所有可能的解码顺序来推广LRAR,而MLM训练任务则相当于对OADM扩散过程的一个步骤进行训练。
因为EvoDiff只需要序列数据,所以它可以很容易地扩展到不同的下游应用程序,包括那些从传统的基于结构的范例中无法访问的应用程序。作为第一个例子,作者已经演示了EvoDiff生成的能力——克服基于结构的预测和生成模型的原型失败模式—通过没有微调的插入绘制。对特定于应用程序的数据集进行微调EvoDiff,例如来自显示库或大规模屏幕的数据集,可能会解锁新的生物、治疗或科学设计机会,否则由于获取大型序列数据集结构的成本是无法获得的。与序列相比,结构的实验数据要稀疏得多,虽然许多序列的结构可以使用AlphaFold和类似的算法进行预测,但这些方法在点突变体上不能很好地工作,而且可能对假蛋白质过于自信。
总之,作者提出了一套开源的离散扩散模型,为基于序列的蛋白质工程和设计提供了基础。EvoDiff模型可以直接部署用于蛋白质序列的无条件、进化引导和有条件的生成,并可以扩展到基于结构或功能的引导设计。我们设想EvoDiff将通过直接在蛋白质语言中的读写功能,实现可控蛋白质设计的新能力。



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


