
研究摘要
本文提出了RTLogD模型,通过使用色谱保留时间数据集进行预训练,并且将pKa特征纳入分子图的原子特征,并同时对LogD7.4和LogP进行多任务学习。该文章强调了使用保留时间数据集进行预训练的重要性和pKa特征的重要性
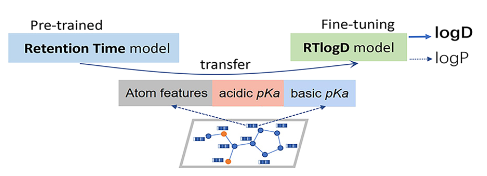
模型架构
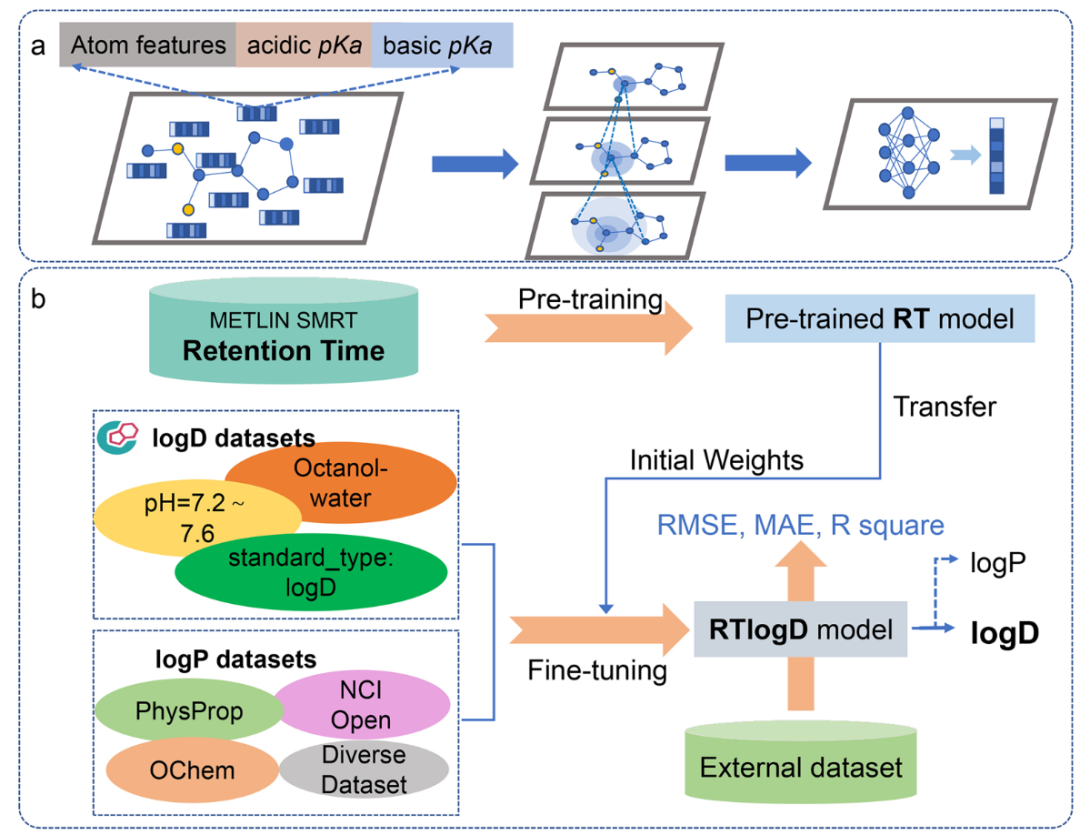
使用其团队在另一篇文章中开发的的AttentiveFP模型,这是一种基于图注意力的模型,添加了一个拥有所有原子特征的超级节点来获取图级特征,在原子中添加pKa特征也就是本文模型与其他模型的不同之处。训练分为2步:
预训练
使用SMRT数据集,一个含有79,957个分子的色谱保留时间数据集(去除无法被RDKit处理的分子后)。使用SmoothL1Loss作为损失函数进行训练,并使用网格搜索进行超参数优化,最终结果与色谱保留时间预测模型GNN-RT相当.
LogD与LogP预测
在通过预训练获得初始权重后,更改模型最后一层的输出,使其从单目标值输出更改为多目标输出值,一个输出用于logP,一个输出用于LogD,除了学习率降为原来的,其余超参数与原模型相同。使用Scaffold分割训练集、验证集、测试集。通过SmoothL1Loss计算logP和logD的loss,如果有缺失值就忽略,在两者都有的情况下,计算折两个任务的平均损失。
主要结果
与baseline(作者自己实现)相比
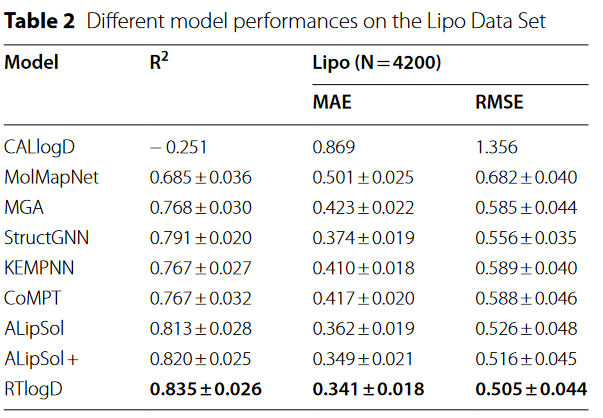
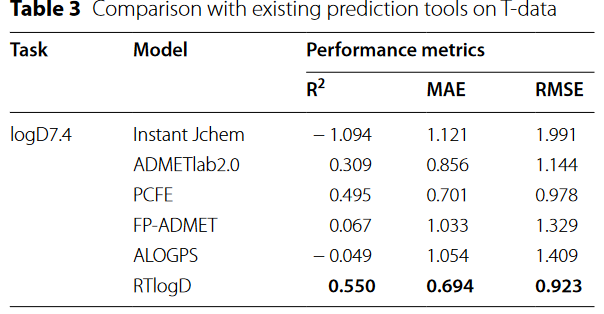
与其他文章的模型相比
消融实验
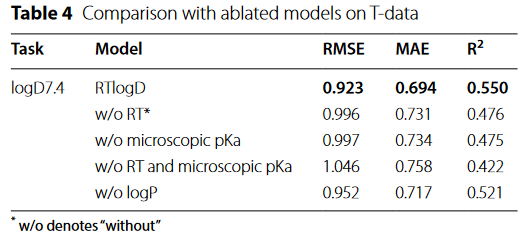
分析保留时间数据集预训练的重要性
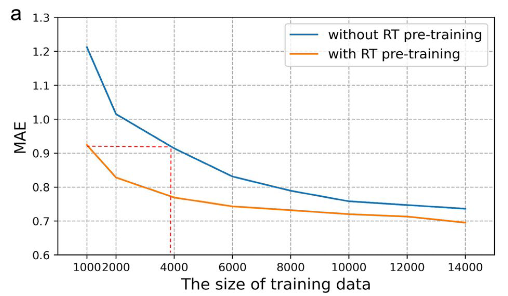 训练数据大小对T数据预测性能的影响。
训练数据大小对T数据预测性能的影响。
通过4个示例分子分析pKa的重要性
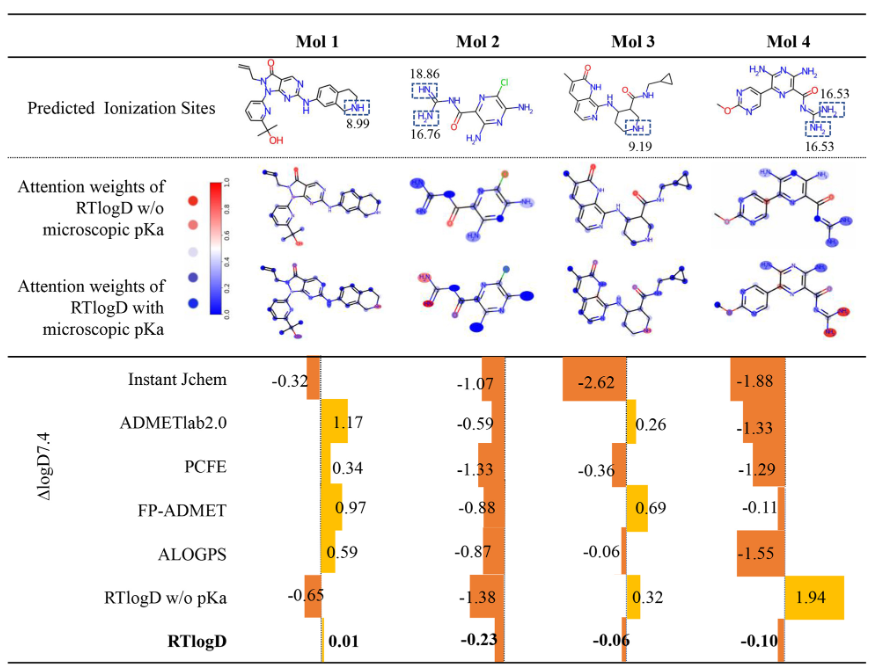 注意力权重可视化,蓝色代表注意力权重小于0.5,红色达标大于0.5,误差值为logD7.4。
注意力权重可视化,蓝色代表注意力权重小于0.5,红色达标大于0.5,误差值为logD7.4。
文章链接:https://doi.org/10.1186/s13321-023-00754-4
代码链接:https://github.com/WangYitian123/RTlogD



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


