关键词:蛋白质结合界面,机器学习,Transformer 网络,对比学习


论文题目:Evaluating protein binding interfaces with transformer networks期刊名称:Nature Machine Intelligence斑图地址:https://pattern.swarma.org/paper/a66db7e6-4dd3-11ee-b3ff-0242ac17000d论文地址:https://www.nature.com/articles/s42256-023-00715-4
计算蛋白质结合研究被广泛用于探究基本生物过程,促进现代药物、疫苗和治疗方法的发展。评分函数的目标是对预测蛋白质复合物的结合强度进行评估和排序。然而,对蛋白质结合界面进行准确评分仍然是一个挑战。
这项研究展示了评估蛋白质结合界面的 Transformer 网络(PIsToN)方法,该方法能够区分类原生的蛋白质复合物和不正确的构象。蛋白质界面被转化成一系列二维图像(界面图),每个图像对应于一种几何或生化特性。像素强度代表特征值。研究从流行的视觉 transformer 模型中改进了一个神经网络,并进行了几项增强:一个混合部分用于接受基于实验的能量项,一个多注意力模块用于突出重要特征和结合位点,并使用对比学习(contrastive learning)来提高排序性能。由此产生的 PIsToN 模型在已有知名数据集上明显优于最先进的评分函数。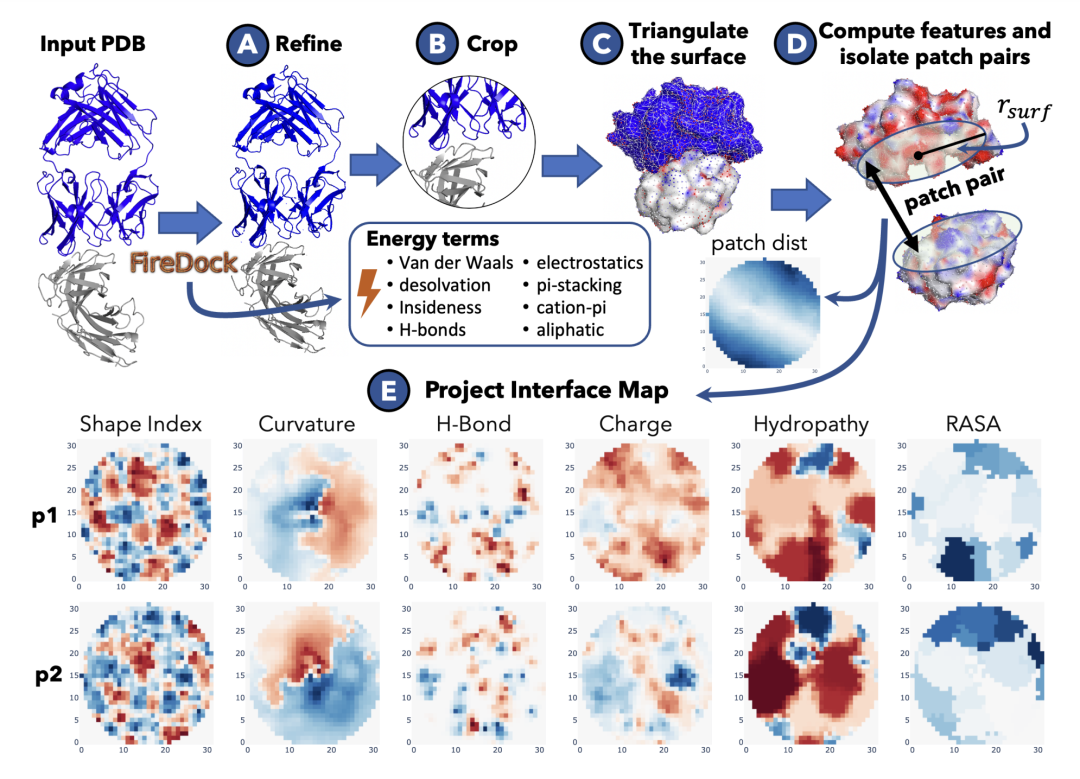 图1. PIsToN 方法对特征计算过程。
图1. PIsToN 方法对特征计算过程。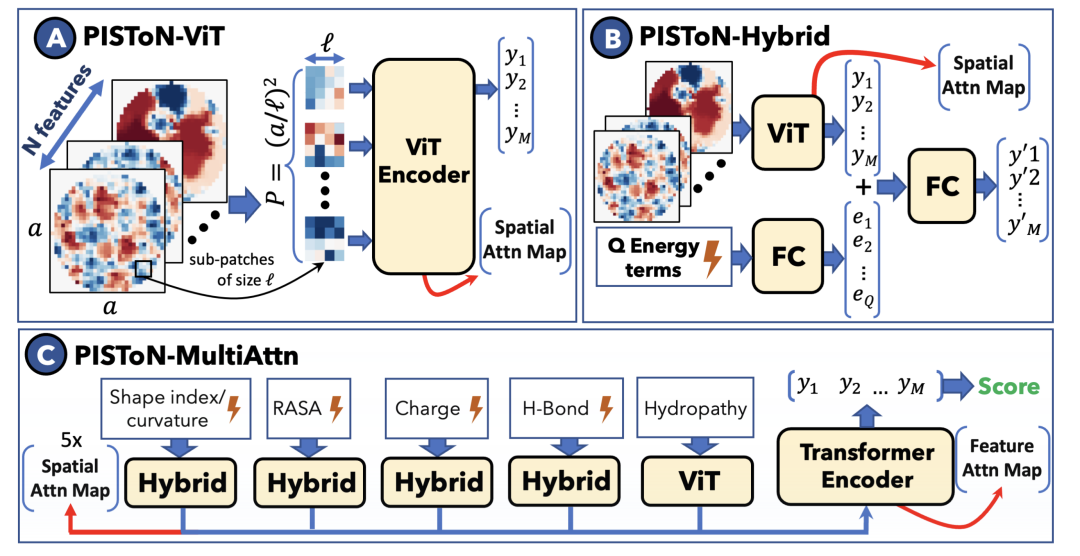 图2. PIsToN 方法的训练过程
图2. PIsToN 方法的训练过程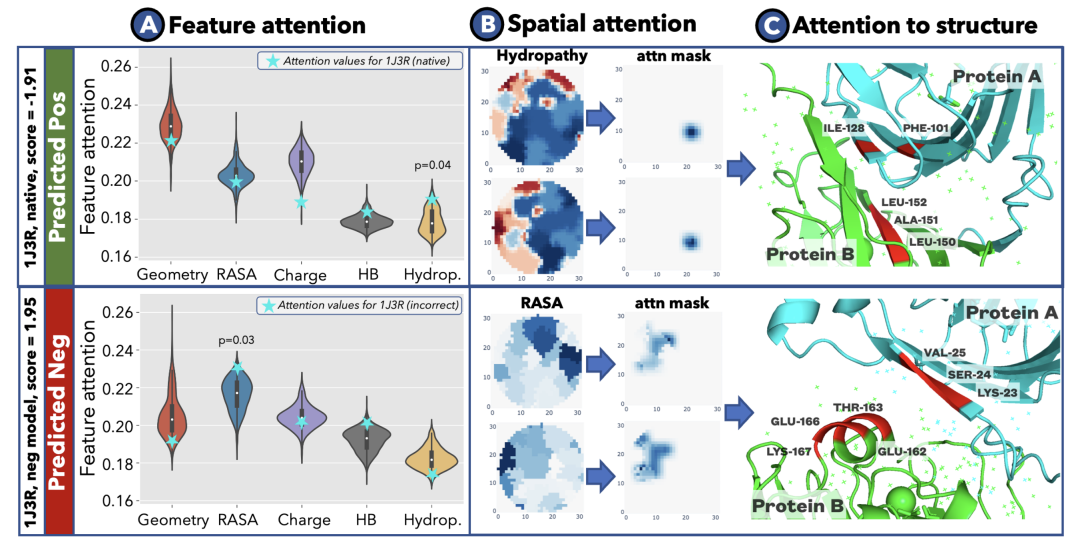 图3. PIsToN 方法的可解释性
图3. PIsToN 方法的可解释性
编译|梁金
大模型与生物医学:
AI + Science第二季读书会启动
详情请见:
推荐阅读
1. PNAS 速递:蛋白质结构预测中 transformer 的变换能力2. 演化能力本身也能演化吗?RNA 和蛋白质适应性景观中增强演化能力的突变 | Nat.Commun. 速递3. Nat. Commun. 速递:无参数几何深度学习精准预测蛋白质结合界面4. 张江:第三代人工智能技术基础——从可微分编程到因果推理 | 集智学园全新课程5. 成为集智VIP,解锁全站课程/读书会6. 加入集智,一起复杂!
点击“阅读原文”,报名读书会



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


