 今天给大家分享一篇由北京大学、中国科学技术大学及微软联合发表在ACL2023上的一篇文章“MolXPT: Wrapping Molecules with Text for Generative Pre-training”。在这项工作中,作者提出了MolXPT,其是一种支持文本和分子的统一语言模型,核心在于使用文本包裹分子的SMILES序列来进行预训练。简单来说,检测每个文本序列中所包含的分子名称,并将其替换为相应的SMILES字符串表示。通过这种方式,分子的SMILES便可以利用周围文本中的信息,反之亦然。最终,将包裹分子SMILES序列的文本、来自PubMed数据集的文本序列以及来自PubChem数据集的SMILES序列输入到语言模型中进行预训练。实验结果表明,MolXPT要优于MoleculeNet上分子性质预测任务的强基线模型;在文本-分子翻译任务中的性能与最佳模型相当,但使用的参数更少,并且无需微调即可实现零样本分子生成。
今天给大家分享一篇由北京大学、中国科学技术大学及微软联合发表在ACL2023上的一篇文章“MolXPT: Wrapping Molecules with Text for Generative Pre-training”。在这项工作中,作者提出了MolXPT,其是一种支持文本和分子的统一语言模型,核心在于使用文本包裹分子的SMILES序列来进行预训练。简单来说,检测每个文本序列中所包含的分子名称,并将其替换为相应的SMILES字符串表示。通过这种方式,分子的SMILES便可以利用周围文本中的信息,反之亦然。最终,将包裹分子SMILES序列的文本、来自PubMed数据集的文本序列以及来自PubChem数据集的SMILES序列输入到语言模型中进行预训练。实验结果表明,MolXPT要优于MoleculeNet上分子性质预测任务的强基线模型;在文本-分子翻译任务中的性能与最佳模型相当,但使用的参数更少,并且无需微调即可实现零样本分子生成。
1 引文
生成预训练Transformer(GPT)已经在自然语言处理方面取得了巨大成功,其相关技术如今也被广泛地应用于分子建模。例如,小分子药物可以用SMILES序列(使用深度优先搜索和其它若干规则通过遍历分子图而获得)来表示。在序列化分子后,便可输入到语言模型中进行预训练,最终实现分子生成任务。文本是分子科学乃至整个科学发现中最重要的记录,其中描述了分子的详细性质,如分子是否有毒,如何合成分子等。在这方面,已有许多工作基于生物医学文献来训练语言模型。
近年来,一个新的趋势是将分子的SMILES序列和文本序列联合建模,以获得两种模态的共享表征。虽然这方面的相关工作在分子预测和生成方面取得了进展,但它们并没有显式地利用分子和文本之间的关系。从直觉上来说,在科学文献中,当一个分子的名字出现在一个句子中,周围的上下文可能是对该分子的描述。所以在联合训练阶段应充分考虑这种上下文信息,但这在以往的研究工作中是被忽略的。
在这项工作中,为了显式地建模分子和文本之间的关系,作者提出了一种分子-文本语言模型(MolXPT),该模型在“包裹”序列上进行了预训练。具体来说,给定一个句子,用命名实体识别工具检测句中所包含分子的名称,如果有,则将它们替换为相应的SMILES序列,从而获得SMILES和文本之间的“包裹”序列。作者预训练了一个24层的MolXPT(有350M参数),其中训练数据包括分子-文本的“包裹”序列(8M)、来自PubChem上的分子SMILES表示(30M)、来自生物医学文献搜索引擎PubMed上的标题和摘要文本(30M)。预训练之后,作者基于提示的方法在MoleculeNet(一个分子属性预测的benchmark)和分子-文本翻译两种不同的下游任务上对模型进行微调。在分子性质预测任务上,MolXPT的表现超过其它强基线模型;在分子-文本翻译任务上,MolXPT的性能和目前的SOTA模型MolT5-large相当,但MolXPT仅使用了其 44% 的参数,且具有零样本文本-分子生成能力。
2 方法
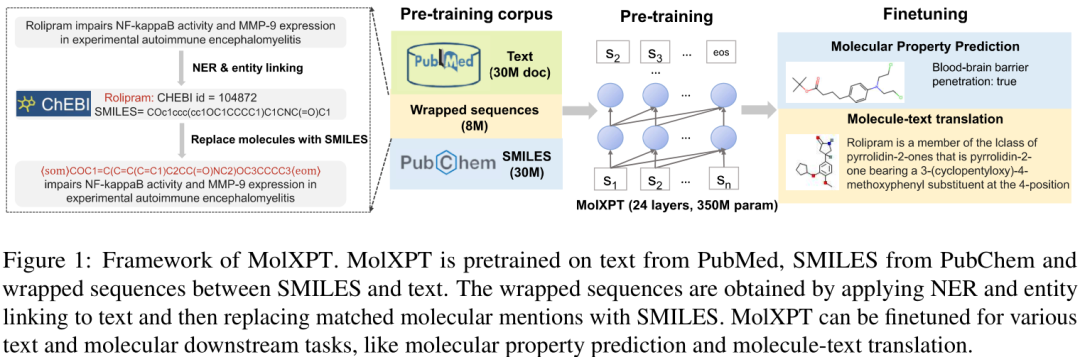
MolXPT模型框架如上,其在科学文本、分子SMILES序列、文本与分子“包裹”的序列这种异构数据上进行预训练。因其灵活的输入,模型可以针对不同的分子和文本下游任务进行微调。
2.1 预训练语料库
科学文本来源于PubMed中论文的标题和摘要(共30M数据),分子SMILES序列来源于PubChem中的分子(随机选择30M的数据)。
由分子和文本构成的“包裹”序列则通过检测加替换的流程来构建。具体来说,首先使用BERN2(一种广泛使用的生物医学命名实体识别工具)来检测文本中可能包含的分子名称,将它们链接到公共知识库(如ChEBI等)中的实体,之后便可以在数据库中检索匹配到的分子实体的SMILES。需要强调的是,这种“包裹”序列至少需要包含一个分子SMILES。最终共得到8M的”包裹“序列数据。
作者分别对文本和SMILES进行了分词。对于文本数据,使用字节对编码算法(byte-pair encoding ,BPE)将其拆分为子词,其中并集操作的数量为40k。对于SMILES序列数据(包括由分子和文本组成的“包裹”序列中的数据),使用正则表达式对它们进行分词。对于每个SMILES序列,在其开头添加一个表示分子序列开始的token,即⟨som⟩,在其末尾添加一个表示分子序列结束的token,即⟨eom⟩。
2.2 模型和训练
- 模型架构
MolXPT与GPT模型的架构相同,但受限于计算资源,在这项工作中作者按照GPT-2 medium版本中的配置将模型层数设置为24层、隐藏层维度设置为1024、注意力头16个。可处理的最大输入长度为2048,词汇表大小为44536,模型拥有350M参数。
- 预训练
MolXPT的预训练目标函数为负对数似然。具体来说,定义为文本、分子SMILES、“包裹”文本这三种类型数据的序列集合,其中表示含有个tokens的第个序列。训练目标函数如下:
- 基于提示的微调
MolXPT可以通过微调来应对不同的分子和文本下游任务。以往研究表明,在预训练好的骨干模型中增加分类或回归的输出层会加大预训练阶段和微调阶段之间的差距。因此,为了和预训练阶段的目标保持一致,采用基于提示的微调方法将不同的下游任务统一为序列生成任务。简单来说,给定一个任务,将输入和输出转换为文本和/或SMILES序列,再为序列配备上针对特定任务的提示,并使用语言模型损失进行微调。
- 讨论
以往的一些研究也试图联合建模文本和分子。例如KV-PLM模型,其在分子名称之后附加SMILES序列进行预训练;MoMu模型使用对比学习联合建模文本和分子图两种模态。MolXPT不同于上述两个基于BERT的模型,其是一个生成模型。MolXPT模型是对以往研究工作的一个补充。
3 实验
作者在两个不同的下游任务上评估MolXPT模型。
(1)在MoleculeNet基准上进行分子性质预测,即预测给定的分子是否具有特定性质。
(2)文本描述和分子之间的生成,即给定文本描述生成相应分子或给定分子生成相应文本描述。
3.1 在MoleculeNet基准上的结果
MoleculeNet是一个广泛使用的分子建模基准,它有超过70万种具有不同性质的化合物数据。作者选择BBBP、Tox21、ClinTox、HIV、BACE、SIDER六个分子分类任务进行评估。遵循之前的研究工作GEM,根据分子骨架将数据集划分为训练集/验证集/测试集。对于上述6个任务,模型输入为SMILES,输出为二值标签。
- 微调策略
先前针对分子性质预测的模型主要使用SMILES序列或者分子图作为输入。本文则使用由文本和分子SMILES组成的”包裹“序列作为模型输入。例如,预测分子血脑屏障穿透(blood-brain barrier penetration,BBBP),提示可以设置为“We can conclude that the BBB penetration of ⟨som⟩ ⟨SMILES⟩ ⟨eom⟩ is ⟨tag⟩”。其中⟨SMILES⟩表示分子的SMILES,⟨tag⟩表示分类标签。所以对于BBBP任务,设计⟨tag⟩为“true”或者“false”来表示化合物是否可以或不能跨越血脑屏障。不同的任务有不同的提示,但是作者将标签(tags)放在所有任务的提示的最后一个token上。令表示某一下游任务中第个具有个tokens的“包裹”序列,其中为序列的标签。假设使用个样本进行有监督的微调。微调策略有两种,一种是仅微调标签,另一种是微调所有提示,公式定义如下:
经过实验分析,上述等式(1)可以取得更好的效果,即仅微调标签。所以在所有任务上均使用等式(1)。给定一段提示文本**”We can conclude that the BBB penetration of ⟨som⟩ ⟨SMILES⟩ ⟨eom⟩ is”**,编码该前缀文本后,最终预测标签“true”和“false”的概率分别为和。分子⟨SMILES⟩能够或不能跨越血脑屏障的概率分别归一化为和。
- 对比baseline及性能
MolXPT和两种类型的baseline进行了对比。一类是预训练语言模型,包括KV-PLM、Galactica、MoMu;一类是预训练GNN,包括GContextual、G-Motif、GROVERbase、GROVERlarge、GraphMVP、MGSSL、GEM。评估指标使用ROC-AUC分数,结果如下表:
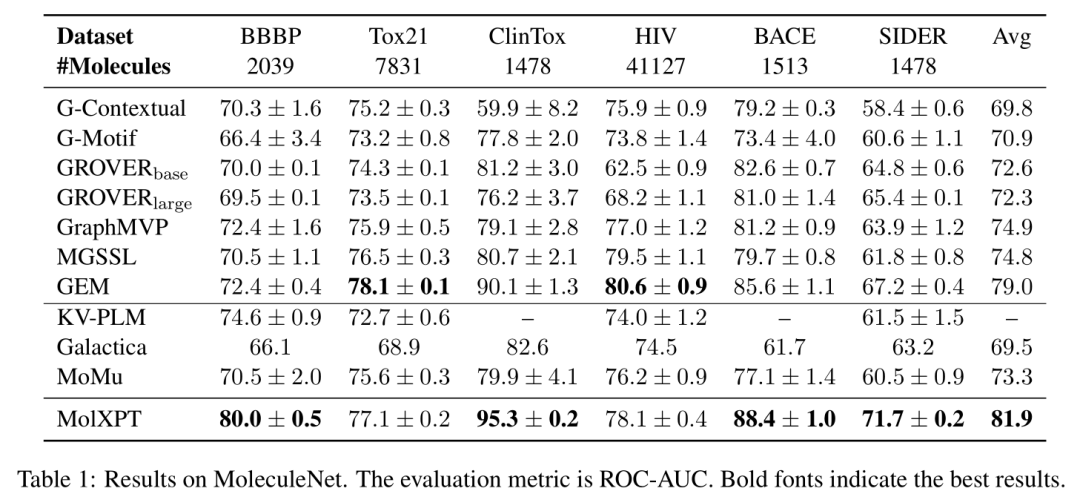
Obs1:MolXPT的性能要优于仅在分子数据上进行预训练的GNN基线模型,这表明使用科学文本语料库进行预训练是有效的。
Obs2:Galactica也是一种使用SMILES和文本进行预训练的类似GPT的模型。相比之下,MolXPT的表现更好。这是因为Galactica没有构建和本文一样的由文本和分子组成的“包裹”序列,并使用它进行预训练。
对于MolXPT模型优越性能的一种可能的解释是分子SMILES描述了分子的组成和结构信息,文本则描述了分子的一般属性。科学文本和分子SMILES相互补充,联合训练它们可以得到更有效的表示。
3.2 在文本-分子翻译任务上的结果
CheBI-20是双向的文本-分子翻译数据集(文本到分子翻译和分子到文本翻译),由33,010个分子-描述对组成。作者在该数据集上评估MolXPT的性能。在数据划分上,作者使用MolT5文章中提供的切分方式,将训练集、验证集以及测试集的比例设置为8:1:1。
- 微调策略
对于分子到文本的生成任务,给定一个分子SMILES序列S,提示设置为”The description of ⟨som⟩S⟨eom⟩ is: The molecule is”,提示中的末尾是针对序列S的文本描述。对于文本到分子生成,给定文本描述,提示设置为“T. The compound is ⟨som⟩”,模型将生成以⟨eom⟩为结尾的分子SMILES序列。
- 对比baseline及性能
主要和MolT5模型进行了对比。对于分子到文本的生成任务,使用NLP中BLEU、Rouge、METEOR三种常用于机器翻译的指标来进行评估。Text2mol是一种基于深度学习的用于衡量文本-分子对相似度的指标。对于文本到分子的生成任务,使用如下评估指标:1)模型生成的SMILES与标准答案SMILES完全匹配的比例(记为“Exact”);2)分子指纹三种谷本系数,分别是MACCS、RDK 、Morgan;3)FCD评分,即通过一个预训练模型测量分子距离;4)生成有效的分子SMILES所占的百分比。评估结果如下表。
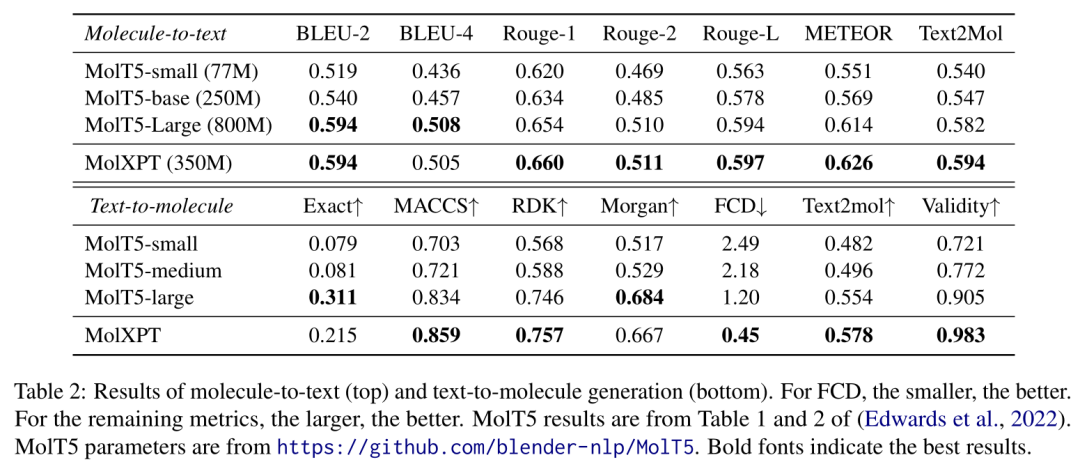
从上述表2中可以发现如下观察:
Obs1:MolXPT的性能明显优于MolT5-small和MolT5-base,并且与MolT5-large的性能相当。MolT5-large具有800M的参数,而MolXPT的参数仅为其44%。
Obs2:对于分子到文本生成和文本到分子生成这两项任务,MolXPT在Text2Mol度量指标上表现最好,这表明“包裹”序列可以显式的学习分子和文本之间的关系,从而更准确地对齐文本和分子。
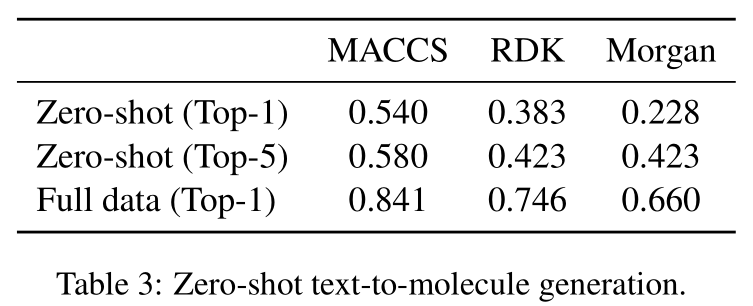
作者进一步验证了MolXPT的零样本文本到分子的生成能力。使用预训练好的MolXPT,将文本作为输入,无需微调即可直接生成分子。top-1和top-5的指纹相似度如下表3。此外,零样本MolXPT模型基于输入文本成功地生成了与标准答案中的分子完全匹配的33个分子。
4 总结
本文提出了一种基于科学文本、分子SMILES及其包裹序列进行预训练的生成式模型MolXPT。该模型共24层,有350M的参数。通过基于提示的微调方法,MolXPT的表现要优于MoleculeNet基准上的强基线baseline,并在分子-文本翻译任务上获得了与最佳模型相当的结果,但使用的参数要少的多。
对于未来的工作,首先,将训练更大的MolXPT,以进一步验证在不同任务上的性能以及零样本/上下文学习的能力。其次,应该研究如何进一步增强分子与文本之间的相互作用(例如,使用对比学习来增强一致性)。最后,如何有效地将MolXPT应用于其他分子和文本任务,如文本引导的分子优化。
5 更广泛的影响
作者提供了一个关于分子和文本的生成预训练模型。一方面,该模型可加快科学发现,如分子设计、药物优化等。另一方面,一旦根据临床数据(其中也描述了药物分子的使用细节)对模型进行训练,可能会导致个人信息泄露。故需要加强数据过滤,匿名化所有个人信息,并设计新的模型来保护信息。文章地址:https://arxiv.org/abs/2305.10688



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


