来自 Google DeepMind 的研究团队基于 AlphaFold 方法论创建了 AlphaMissense——通过利用蛋白质序列数据库和变异结构背景,可以识别致病的错义突变和未知致病基因。与许多现有的类似工具(变异效应预测器或 VEPs)相比,AlphaMissense 表现出了更优越的能力。
Science:https://www.science.org/doi/10.1126/science.adg7492
数据集:https://github.com/deepmind/alphamissense
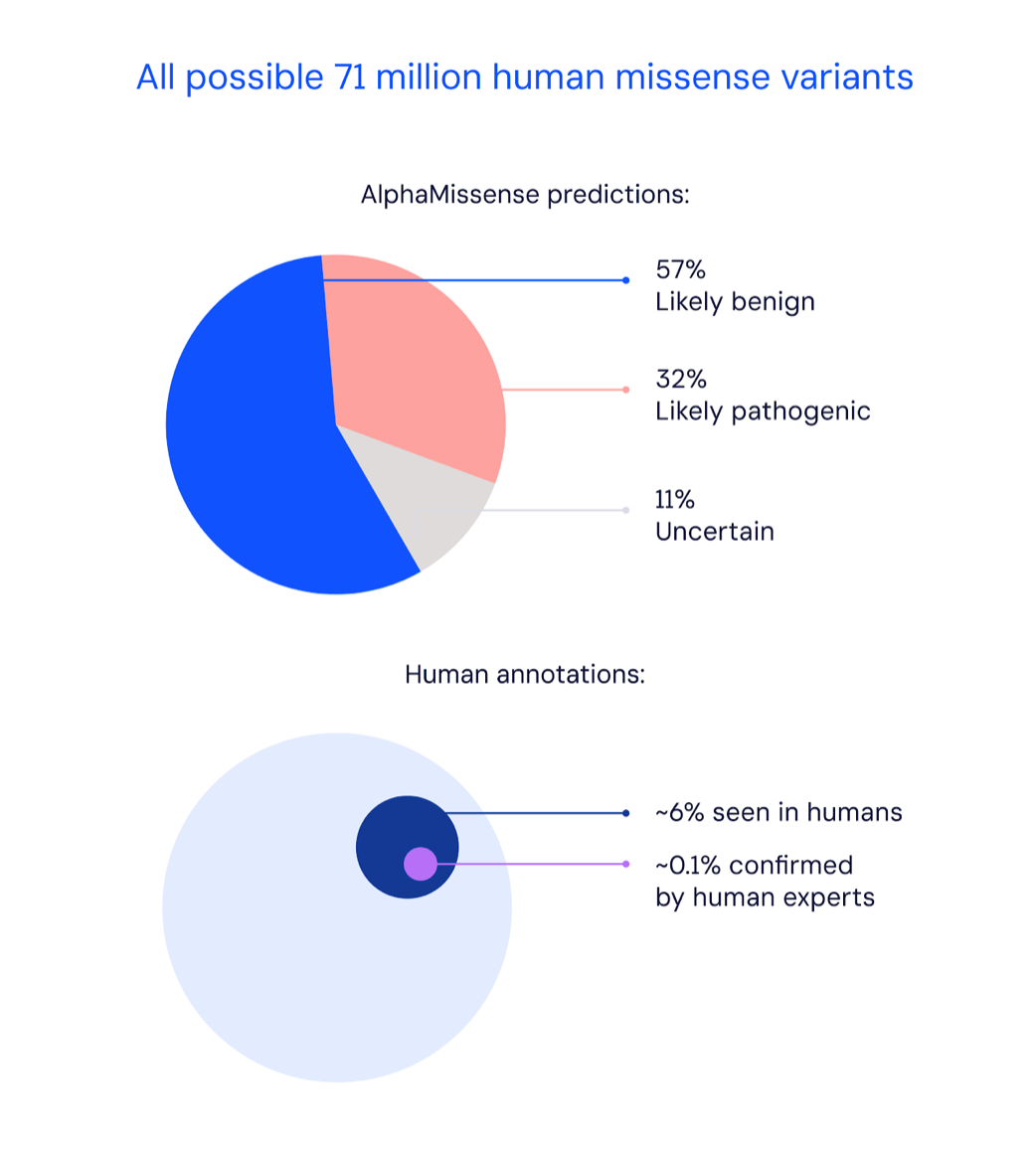
AlphaMissense 成功预测了 19233 个标准人类蛋白质的 216 百万种可能的单一氨基酸变化的致病性,得到了 7100 万个错义突变的预测。随后,AlphaMissense 更是成功预测出 89% 的错义突变,其中 57% 可能是良性的,32% 可能是致病的。
相关研究论文以“Accurate proteome-wide missense variant effect prediction with AlphaMissense”为题,已发表在权威科学期刊 Science 上。

错义变异是指一种可以改变蛋白质氨基酸序列的遗传变异。致病性错义变异会严重破坏蛋白质功能,降低生物体适应性,而良性错义变异的影响有限。
在超过 400 万个观察到的错义变异中,仅有约 2% 被临床分类为致病性或良性,对剩余未知的变异进行分类是人类遗传学中的一个重要挑战。缺乏准确的错义变异功能预测限制了罕见疾病的诊断率以及针对潜在遗传原因的临床治疗的开发和应用。
虽然多重分析变异效应(MAVEs)系统地测量蛋白质变异的效应并可以准确预测变异的临床结果,但 MAVEs 实验需要高昂的费用和劳动力,因此蛋白质组范围内的变异致病性调查仍然不完整。
借助 AlphaFold 的方法论,AlphaMissense 结合了现有策略的三个元素:
1)基于人口频率数据的弱标签训练,避免使用人工注释,从而避免了循环性;
2)通过使用无监督的蛋白质语言建模任务来学习在序列上下文中条件化的氨基酸分布;
3)通过使用 AlphaFold 派生的系统来整合上下文。
AlphaMissense 的训练分为两个阶段:结构预训练和变异微调。其中,预训练阶段与 AlphaFold 中描述的相同,但在蒙版多序列比对重建损失上增加了更高的权重;在微调期间,模型被优化,可以同时预测变异的致病性和参考序列的结构。



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


