编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Shiwei Sun, Peter Pak-Hang Cheung和 Xin Gao团队的一篇与SARS-CoV-2相关的论文。SARS-CoV-2的持续演变对公共卫生构成了重大威胁。由于庞大的序列空间,了解潜在的抗原变化具有重要意义,但也具有挑战性。在这里,作者引入了机器学习引导的抗原进化预测(MLAEP)方法,它结合了结构建模、多任务学习和遗传算法,通过体外定向进化模拟来预测病毒的适应性景观并探索抗原进化。通过分析现有的SARS-CoV-2变异,MLAEP准确地推断了抗原进化轨迹上的变异顺序,与相应的采样时间相关联。作者的方法在免疫功能受损的COVID-19患者和新出现的变异(如XBB1.5)中识别出了新的突变。

随着感染病例数量的增加和SARS-CoV-2在全球范围内传播,病毒基因组中出现了新的突变。截至2022年4月,已经在全球禽流感数据库(GISAID)中识别并上传了超过一百万个病毒基因组变异。这些突变通常涉及对SARS-CoV-2特性的改变。虽然大多数突变会降低病毒的毒力和传播能力,但一些单个或组合突变可以显著提高病毒的传播能力,增强细胞进入效率,或者消除感染或疫苗接种引发的中和抗体反应,从而产生高风险的变异。大部分中和抗体,包括单克隆抗体和疫苗诱导的抗体,都会针对刺突蛋白受体结合域(RBD)。针对RBD的抗体根据它们的结合表位被分为四类。第一类和第二类抗体结合于受体结合基序(RBM)的表面,从而与ACE2竞争RBD的结合。第三类抗体结合于RBM的相反侧面,与ACE2结合的重叠较少,与第一类和第二类抗体相结合时,提供截断ACE2结合的协同效应的潜力。第四类抗体靶向Sarbecoviruses中高度保守的区域,因此通常对变异更具抵抗性。然而,新出现的病毒系列如Omicron和BA.2仍然可能导致中和作用的丧失。
因此,理解这些突变的作用以及它们与传播能力和免疫逃逸之间的联系非常重要。在这项工作中,作者提出了基于现有数据和方法构建的MLAEP,用于预测整个RBD区域中的组合突变,该区域具有高抗原演化潜力。作者假设在高免疫压力下,病毒在短期时间尺度内会趋于逃避抗体的中和作用,因此预测问题转变为搜索问题:从初始序列开始,搜索在某个编辑距离范围内具有改进抗体逃逸潜力而不会失去太多ACE2结合能力的变异序列。通过直接测量RBD变异与ACE2和八种抗体的结合亲和力的DMS数据集,作者建立了一个多任务深度学习模型,可以同时预测变异对ACE2和抗体的结合特异性。
MLAEP模型
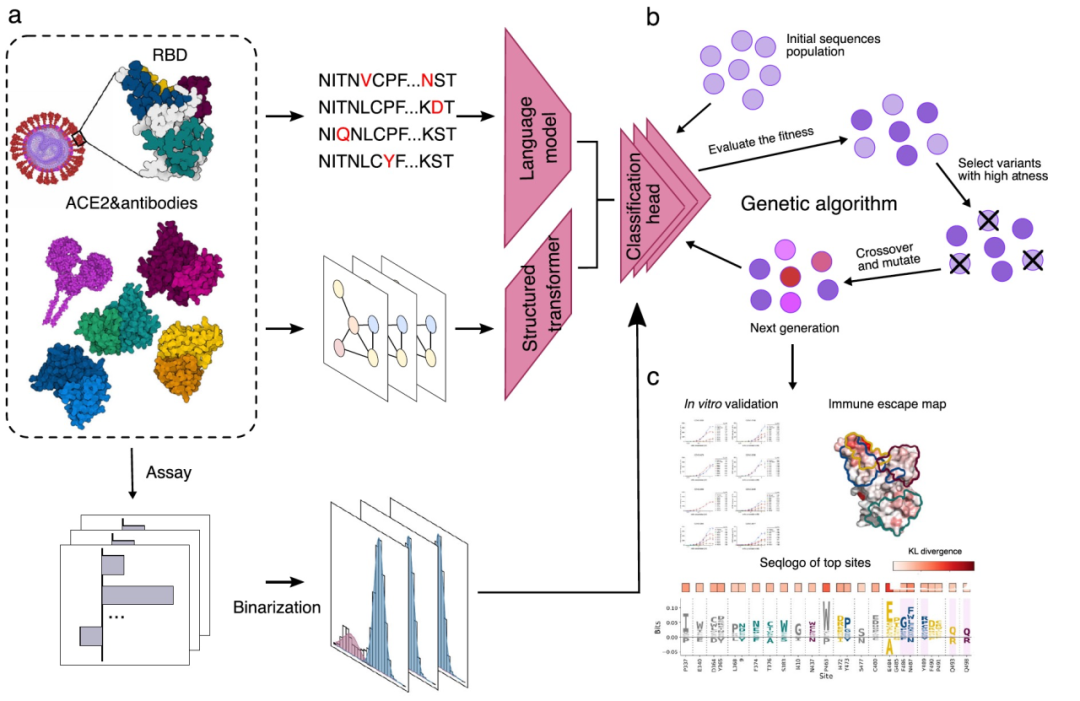
图 1
作者首先开发并训练了一个多任务深度神经网络模型,能够预测变异的RBD结合特异性,包括对ACE2和四种抗体的结合特异性,如图1所示。该模型接收两个输入:变异的RBD序列和ACE2/抗体的三维结构,并输出这两个输入的结合特异性。然后,该模型使用多任务目标函数进行训练,同时预测变异序列对所有目标的结合特异性。序列特征从微调了ESM-1b语言模型提取。使用ESM-1b模型,氨基酸序列被转换为密集的向量表示。对于ACE2/抗体结构,作者首先根据它们的接触图和生物物理性质将三维结构转换为图,然后使用结构化变换器进行结构特征提取。使用这两个模型作为特征提取模块,作者添加了九个并行的线性分类层,以在绑定目标结构的条件下学习序列到功能的映射(图1a)。由于对于变异体有多个结合目标,作者采用硬参数共享方案进行多任务学习,其中所有模块在所有九个任务中共享相同的参数。然后,以端到端的方式训练整个框架。最终,模型学习如何预测ACE2和八种抗体的结合特异性。给定一个输入的RBD变异序列,作者的模型输出九个分数,对应于ACE2和八种抗体,将八种抗体分数的平均值定义为预测的抗体逃逸潜力。
作者的假设基于抗原演化:不会在失去太多ACE2结合能力的情况下,未来的病毒变种在高免疫压力下往往会具有更高的抗体逃逸潜力。因此,模型学习的抗体/ACE2结合特异性可以用于为可能引起未来关注的新变异体的搜索提供有意义的方向。受到机器学习引导的蛋白质工程领域的进展的启发,作者将训练的多任务模型用作评分函数(图1a),将所有九个任务的平均预测分数作为适应度分数,并使用修改后的遗传算法搜索具有改进适应性的新变异体(图1b)。遗传算法受到自然选择过程的启发,它以迭代方式将一组候选序列演化到更好的适应性。每次迭代的群体被称为一个代。在每一代中,使用训练好的模型评估候选序列的适应性。然后,通过选择具有更高适应性的候选序列,以更高的概率培育下一代(图1c)。
多目标学习的有效性
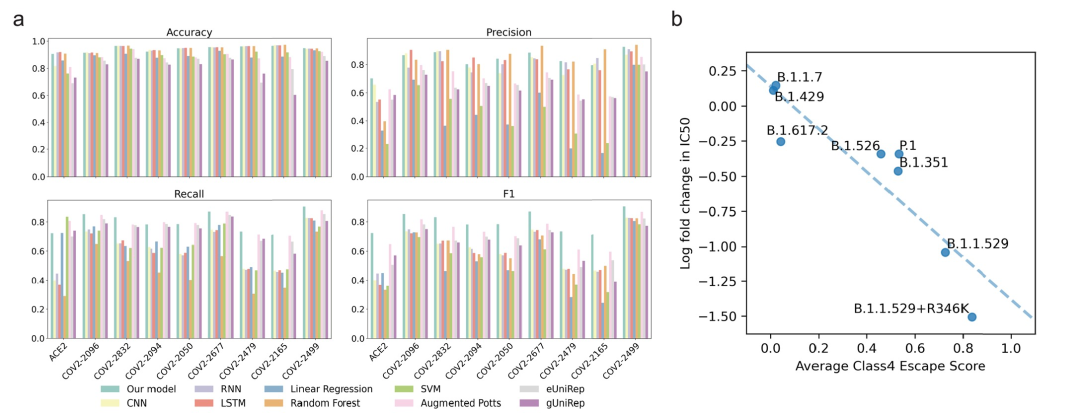
图 2
MLAEP遵循机器学习引导的定向进化范例,而生成序列的质量在很大程度上取决于序列到功能模型。首先,作者通过五折交叉验证验证了模型对新观察到的变异的泛化能力。作者收集和清理了九个包含19132个变异序列及其对ACE2和来自四个功能类别的八种抗体的结合特异性的深度突变数据集。然后比较了一系列专门为蛋白质工程设计的模型,并评估了它们在从变异序列中分类绑定物和非绑定物的性能。由于所有九个任务的正样本和负样本数量不平衡,因此作者报告了宏观精确率、宏观召回率和宏观F1分数,以增加对较小类别的权重。结合结构特征,作者的模型在预测所有九个任务的突变效应方面优于其他先进方法(图2a)。因此,作者在下游分析中专注于此模型。
图 3
Evo-velocity能够使用深度学习模型推断蛋白质的进化动力学。它是基于全局进化通过局部氨基酸变化发生,并利用蛋白质语言模型来建模进化的局部规则的前提建立的。作者评估了模型使用Evo-velocity推断现有RBD序列的进化轨迹的能力。作者使用了来自GISAID数据库的现有SARS-COV-2 RBD序列。现有的GISAID变异序列首先通过多任务模型转换为嵌入向量。在嵌入向量之上,根据模型预测的平均分数的变化为它们分配了方向,从而形成了进化的“矢量场”。作者使用Uniform Manifold Approximation和Projection (UMAP)将嵌入向量在二维空间中可视化。关注变异体,包括Alpha、Beta、Delta和Omicron,被映射到不同的聚类中,这些变异体之间的速度与已知的进化轨迹相吻合(图3a)。尽管模型仅使用RBD序列进行训练,但模型推断出的伪时间与已知的变异抽样时间有0.55的Spearman相关性。使用ESM-1b模型,得分迅速下降至-0.38。可以注意到,大量的突变发生在RBD区域之外,这可能解释了ESM-1b模型伪时间与采样时间之间的弱相关性。较长的序列长度会使ESM-1b模型的性能更好。
体外定向进化作为预测工具
图 4
利用模型作为评分函数,作者使用遗传算法搜索具有高抗原性进化潜力的新的RBD变异序列。搜索过程包括从GISAID数据库中选择初始序列,使用遗传算法生成并选择“优于初始”的序列,以在初始序列的15个突变“信任半径”内产生38,870个可能的高风险变异体。作者对GISAID数据库中的序列进行了搜索,时间范围从2022年1月1日到2022年3月8日,共得到了971个不同的序列。然后使用保持距离的多维缩放图(图4a)将生成的序列与现有序列一起可视化。尽管来自深度突变扫描实验的序列仅占据野生型序列周围的一个小区域,但盛行的变异体(例如,Omicron)位于不同的区域,远离野生型。与种子序列相比,作者的模型生成的合成序列包括了用于ACE2结合和抗体逃避的关键突变。为了可视化差异并进一步探索生成的突变模式,作者为两个序列集构建了位置频率矩阵(PFM),并基于两个PFM计算了每个位置的Kullback-Leibler距离(KL距离)。图4b提供了基于结构的可视化,并将每个位点的Kullback-Leibler距离投射到RBD的晶体结构上(PDB id: 6m0j)。作为替代表示,图4c为前50个最大差异位点提供了一个概率加权的Kullback-Leibler logo图,其中字母的总高度表示位点的KL距离,字母的大小与相对对数几率得分和观察概率成比例。
结论
作者提出了一种机器学习引导的抗原进化预测范式,用于预测SARS-CoV-2的抗原进化。作者训练了一个多任务深度学习模型,使用变异序列和结合目标结构来预测ACE2/抗体结合特异性。预测ACE2结合特异性是一个相对简单的任务,因为可以使用无监督学习的模型来捕捉结合特异性。然而,预测抗体结合特异性要困难得多。通过各种验证实验,模型可以预测由高免疫压力导致的抗原进化潜力。生成的合成序列显示出很高的免疫逃避潜力,作者使用体外计算工具和体外中和抗体结合实验进一步验证了这一点。
参考资料
Han, W., Chen, N., Xu, X. et al. Predicting the antigenic evolution of SARS-COV-2 with deep learning. Nat Commun 14, 3478 (2023).
https://doi.org/10.1038/s41467-023-39199-6





 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


