2023年9月7日,意大利比萨大学Miriana Di Stefano等人在Journal of Chemical Information and Modeling上发表文章VenomPred 2.0: A Novel In Silico Platform for an Extended and Human Interpretable Toxicological Profiling of Small Molecules。
 作者开发了VenomPred 2.0,一个强大的在线平台,可以通过基于多个机器学习模型的全面共识预测策略进行化合物毒性评估。此外,还开发了一个基于Shapley加性解释(SHAP)方法的新实用程序,进行可解释的小分子毒理学分析,能够突出显示对毒理学预测有重要贡献的特征,以获得毒理学结构基团。
作者开发了VenomPred 2.0,一个强大的在线平台,可以通过基于多个机器学习模型的全面共识预测策略进行化合物毒性评估。此外,还开发了一个基于Shapley加性解释(SHAP)方法的新实用程序,进行可解释的小分子毒理学分析,能够突出显示对毒理学预测有重要贡献的特征,以获得毒理学结构基团。
VenomPred 2.0 见
http://www.mmvsl.it/wp/venompred2/
背景
机器学习在毒理学中的应用,旨在开发新的基于人工智能的计算模型,能够预测化合物的毒性特征,目前正在取得进展。计算机毒理学的机器学习模型有助于降低体外和体内研究所需的高成本和长时间,并可以处理大量数据。目前,研究者们为收集和评估有关化学物质的多种特性和危害的数据进行了许多努力。
方法
VenomPred2.0中,用于生成雄激素模型的数据集来自ToxCast/Tox21和ChEMBL数据库,如果一种化合物在至少一种竞争性结合试验中显示出结合活性,并且在至少一种报告基因试验中检测到这种活性,则该化合物被标记为有毒。如果结合试验和所有报告的基因试验结果均为阴性,则化合物被标记为无毒。
皮肤刺激、眼睛刺激和急性口服毒性终点的数据集从文献和公开可用的REACH数据库中收集,如果化合物表现出刺激或腐蚀性,则将其归类为有毒。在刺激性方面,根据全球化学品统一分类和标签系统(GHS)的报告,如果化合物相应的平均红斑/水肿评分高于2.3分,则被认为是有毒的。在腐蚀性方面,如果报告了不可逆的皮肤刺激和腐蚀作用,则将化合物归类为有毒化合物。眼睛刺激数据从REACH数据库中收集,并根据单剂量化学品应用后获得的结果进行分类。如果一种化合物对结膜、角膜和虹膜有破坏性影响,则被认为是有毒的。对于急性口服毒性终点,数据集来自替代方法验证机构间协调委员会(ICCVAM)。根据相应的半致死剂量(LD50)对数据集中的化合物进行分类。具体来说,LD50值低于2000 mg/kg的化合物被认为是有毒的。
对于致突变性、致癌性、肝毒性和雌激素性终点,使用了与VenomPred相同的训练和测试集。对初始数据集进行处理,以去除不一致和重复的实例;然后,用标准化的SMILES表示化合物的结构。最后,采用随机分割策略,将每个端点得到的精细化数据集分为训练集和测试集,分别占原始数据集的80%和20%。每个端点获得的训练集和测试集使用t分布随机邻居嵌入(t-SNE)算法进行降维。
对于每个数据集,计算了化合物的分子表示。具体来说,基于VenomPred的结果,计算了Morgan、RDKit和PubChem的化学指纹(FPs)。Morgan和RDKit FPs使用RDKit python库生成,PubChem FPs使用PyBioMed python模块计算。
Morgan FPs根据周围原子和原子之间一定距离内的键来表示化合物的结构 (本文设置为2),并赋予它们一个唯一的标识符。这些标识符通常被散列成一个固定长度的位向量,以便比较不同的表示。在这项工作中,使用RDKit的Morgan FPs实现设置了1024位的矢量长度。RDKit FPs是RDKit特有的指纹,指纹识别算法识别分子中特定大小范围内的所有子图,对每个子图进行哈希以生成原始比特ID,调整原始比特ID以适应指定的指纹大小,然后设置相应的比特。PubChem FPs由基于子结构的FPs组成,用881位向量表示,其中每个位编码元素或子结构、特定环系统、原子对和原子最近邻居的存在。
特征重要性分析是根据Shapley范式进行的,Shapley范式是一种广泛使用的方法,用于评估单个组件对最终结果的影响,该方法源自博弈论。该方法应用于模型,目的是评估每个单独特征对最终预测的权重。在这项工作中,Shapley方法被应用于确定单个FP位在预测生成中的影响程度,如图1所示。
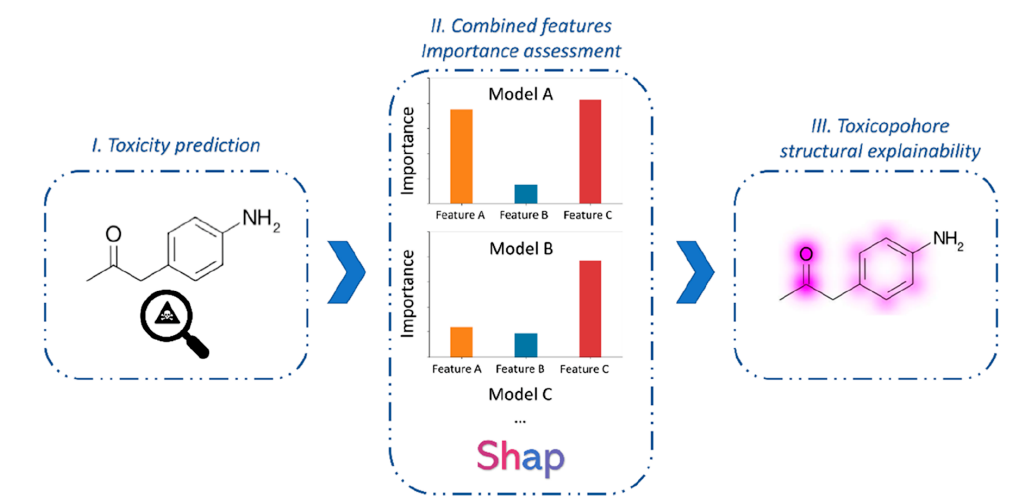 图1 VenomPred2.0与SHAP评估
图1 VenomPred2.0与SHAP评估
通过遵循Shapley值方法计算特征对模型预测的贡献。Shapley值是一种公正合理地评价每个个体重要性的稳健方法,它得到了一个独特的结果,其特点是:局部准确性、一致性和零效应。使用Shapley加法解释(SHAP)值解释机器学习模型背后的思想是基于识别与模型结果直接相关的重要特征。对于二元分类器,如本文开发的模型,特征的重要性提供了一个与贡献幅度方向相对应的符号。具体来说,正号表示对毒性预测的贡献,而负号表示对无毒性预测的贡献。考虑到SHAP方法的模型依赖特性,采用由SHAP python库提供的内核SHAP模型无关方法。由于与所有Shapley值的精确计算相关的计算成本相当大,因此这种局部近似方法代表了获得机器学习模型可靠解释的有效替代方法。
VenomPred2.0使用四种不同的分类算法 (随机森林、支持向量机、k近邻和多层感知器) 开发了用于预测不同毒性终点的模型。使用python Scikit-learn库的专用函数来生成模型。
随机森林(RF)算法由大量的决策树组成,这些决策树作为一个整体工作。每个单独的树提供一个类预测。获得多数选票的类代表模型的最终预测。在模型构建过程中优化的主要超参数是max_features和n_estimators, max_features表示在单个树中可以考虑的最大特征数量,n_estimators表示在预测之前构建的树数量。
支持向量机(SVM)根据数据的共同模式对数据进行映射,目标是在两个类之间进行最优划分,每个类都完全位于分离超平面的相对两侧。目标是通过最大化最近的训练数据点(所谓的支持向量)和超平面之间的距离来实现的。模型构建过程中优化的超参数有:(1)核函数,它表示将数据映射到高维特征空间以使它们可分离的函数;(2)惩罚项系数,表示对错误分类数据的重视程度,从而有助于超平面的优化。
K近邻(KNN)算法根据其邻居的类别对实例进行分类。通过考虑其k个最近邻居中最具代表性的类来预测实例的类。因此,最终的预测是通过最接近输入数据的近邻特征的最频繁输出来获得的。模型生成过程中优化的超参数是那些减少由于周围邻居投票而产生的误差的参数,即n_neighbors和weight。N_neighbors表示用于分类的邻居的数量,而weight定义了不同的周围元素对最终预测的影响程度。
多层感知器(MLP)是一种由多层节点组成的前馈人工神经网络,它使用反向传播的监督学习策略。为了最小化输出预测的误差,调整了四个超参数:(1) hidden_layer_size,表示神经元的数量和隐藏层的数量;(2)求解器,它是通过不同层优化每个决策步骤的预测的基本参数;(3)激活函数,定义了输入的加权和如何被每一网络层的一个或多个节点转化为输出;(4) learning_rate_init,它控制更新权重的步长。
通过使用3种不同的化学FPs和4种不同的机器学习算法,为每个考虑的数据点生成了12种不同的毒性模型。在Scikit-learn中实现了基于网格搜索交叉验证的超参数优化过程。
共识方法包括从多个模型提供的预测结果的组合中生成特定终点的分子最终毒性预测 (称为共识预测)。具体来说,当机器学习模型返回给定化合物的毒性预测时,概率评分(PS)在0到1的范围内。通过对共识预测中包含的每个模型产生的PS进行平均来生成共识评分(CS);如果获得的CS小于0.5,则根据一致预测将化合物标记为无毒,如果CS等于或大于0.5,则标记为有毒。
结果
作者将VenomPred 2.0与一些具有代表性的方法进行了比较,对比马修斯相关系数结果如表1所示。VenomPred 2.0在8个指标(end point)中的6个中的马修斯相关系数均最高。结果表明,模型在预测化合物毒性方面优于其他方法。(表格注释:(a) n.d.表示not determined即未定值 (b) 该值通过评估皮肤对化合物的敏感度来计算)
表1 与其他方法对比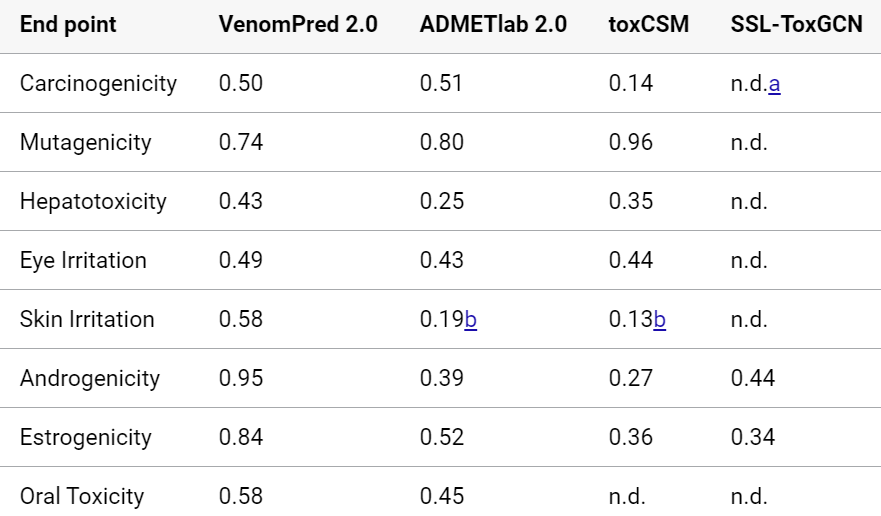
作者设计了模型消融实验,对比了使用共识策略和仅使用单个模型的MCC值的变化,如图2所示,以诱变性为例,使用共识策略后,马修斯相关系数由0.69(红线)提升到了0.72(黑线),表明共识策略的有效性。图2中,纵坐标和蓝色区域表示采用共识策略时,各个分子的马修斯相关系数分布及其频数值。
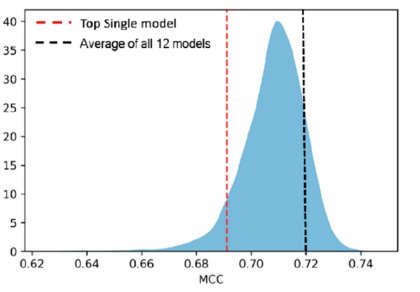
图2 消融实验
作者还进行了案例分析。以急性口服毒性为例,使用乙基对硫磷作为测试结构。该化合物是一种已知的有机磷杀虫剂,含有有机硫代磷酸盐基团,通常通过抑制乙酰胆碱酯酶来破坏神经系统细胞。事实上,在摄入对硫磷后,一种氧化酶用氧取代了双键硫,从而产生了对硫磷,它在生物体中比磷硫酯更具活性。该衍生物作为乙酰胆碱酯酶抑制剂,可引起恶心、呕吐、腹痛、腹泻、流涎等典型症状。VenomPred 2.0正确预测了其急性口服毒性,基于SHAP分析,硫磷键被确定为该化合物毒性预测的主要因素(图3),这与文献报道的有机硫磷化合物的作用机制一致。
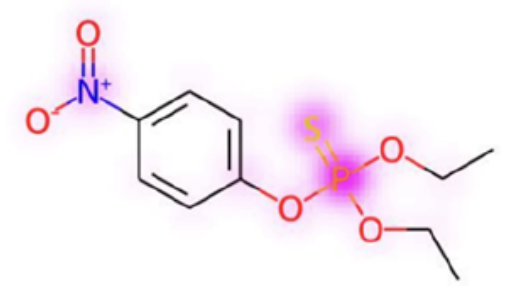
图3 案例分析
总结
本文介绍了VenomPred 2.0,一个强大的基于网络的平台,用于多方面和可解释的化合物毒性预测。VenomPred 2.0保持了免费和用户友好的特性,提供了多重毒性终点,即致癌性、诱变性、肝毒性、雌激素性、雄激素性、皮肤刺激、眼睛刺激和急性口服毒性。基于多种机器学习模型的组合使用,采用了一种创新的详尽共识策略,可以最大限度地提高毒性预测性能及其可靠性。
VenomPred 2.0实现了一个基于Shapley加法解释(SHAP)方法的新实用程序,该方法赋予VenomPred 2.0预测的可解释性,从而能够探索与预测的分子毒理学效应相关的特定结构部分。这样,只需在平台上加载所需化合物的SMILES字符串,或绘制相应的分子结构,用户就可以轻松快速地获得有关所需分子的毒理学潜力以及可能导致其毒性的毒性基团和结构片段的信息。
参考资料:
[1] Stefano et al. VenomPred 2.0: A Novel In Silico Platform for an Extended and Human Interpretable Toxicological Profiling of Small Molecules. J Chem Inf Model. 2023
[2] Galati et al. VenomPred: A Machine Learning Based Platform for Molecular Toxicity Predictions. Int J Mol Sci. 2022
——— End ———





 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง





