编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Yasushi Okuno团队的一篇论文。识别化合物-蛋白质相互作用(CPI)对于药物发现至关重要。由于实验验证CPI通常耗时且昂贵,因此期望计算方法能够促进这一过程。可用的CPI数据库迅速增长加速了许多机器学习方法用于CPI预测的发展。然而,它们的性能,特别是它们在外部数据上的泛化能力,往往受到数据不平衡的影响,这归因于缺乏经验证的非活性(负面)样本。在这项研究中,作者开发了一种自我训练方法,用于增加可信和信息丰富的负样本,以改善由数据不平衡导致的模型性能下降问题。构建的模型表现出比使用其他传统方法解决数据不平衡时更高的性能,且在外部数据集上改进明显。

识别化合物-蛋白质相互作用(CPI)对于药物发现至关重要。特别是在早期药物开发阶段,许多CPI通过各种生物学测定技术进行实验评估,以寻找命中化合物并避免不希望的非靶效应。这些实验方法往往耗时且昂贵。因此,期望计算方法能够加速在药物发现中识别CPI。目前计算方法大致分为两类:基于结构和基于结构无关(基于化学基因组学)的方法。基于结构的方法通过从分子对接模拟生成的三维复合物模型中计算基于物理化学的分数来评估CPI。当目标蛋白质的可靠三维结构可用时,可以在没有已知相互作用的先验信息的情况下应用对接方法。它们的性能在很大程度上取决于评分函数的准确性和预测的对接构象。基于结构无关的方法主要是化学信息学方法,利用已知CPI的先验信息来预测未知的相互作用。当先验信息涵盖足够的药理空间时,它们可以以相对较低的计算成本准确预测CPI。
近年来,诸如ChEMBL、BindingDB、PubChem、DrugBank和PDBbind等公开可用的CPI数据库迅速增长,从而加速了使用机器学习(ML)算法开发各种基于结构无关方法。这些ML方法在统一的框架中考虑化合物信息、蛋白质信息以及它们之间的相互作用。最近,使用深度学习(DL)技术的CPI预测模型,如卷积神经网络(CNN)、图卷积网络(GCN)和Transformer算法,显著提高了预测性能和可解释性。这些模型可以在学习相互作用的端到端学习过程中提取化合物和蛋白质的特征表示。使用机器学习(ML)技术的基于结构无关方法的性能常常受到训练数据质量的影响,这些数据来自于CPI数据库中已知的相互作用。在许多情况下,公共数据库中缺乏经验证的非活性(负面)样本,这导致了可用CPI数据中的类别不平衡。这种不足导致了ML模型在域外样本上的表现不佳,并在CPI预测中由多数类引起的过度估计。为了弥补负样本的不足,先前的研究采用了随机配对方法(以下简称随机负样本)。该方法从尚未经实验证实为相互作用的化合物-蛋白质对中随机生成负样本。然而,提取的样本可能包含潜在的活性(正面)样本,从而降低了可信度。
为了克服这些限制,作者提出了一种新的自我训练方法,有效增加负样本。这种方法被应用于基于图的CPI预测模型,并成功地提高了模型的性能,包括泛化能力。
自我训练方法的工作流程
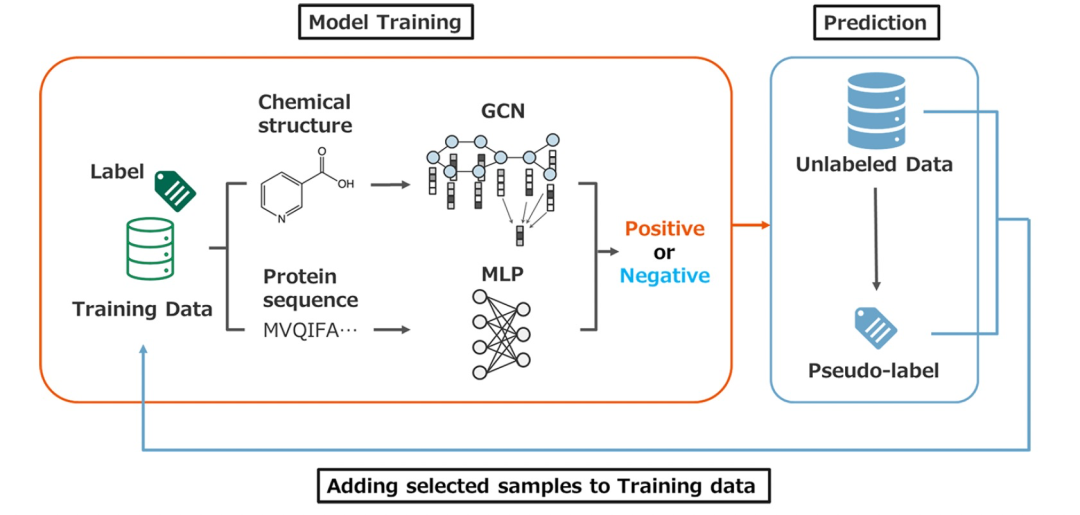
图 1
自我训练始于使用标记数据训练一个教师模型。教师模型用于通过对未标记数据进行预测来生成伪标签。在将选定的伪标记数据添加到训练数据后,使用更新后的数据训练一个学生模型。这个过程是通过使用学生模型作为下一个教师模型来迭代的。图1说明了作者提出的自我训练方法的工作流程,并且实施了以下步骤。第一步,使用最小化二元交叉熵损失进行训练,得到教师模型。第二步,利用教师模型f为未标记的数据集生成伪负标签。如果对于某个数据样本,满足ϕ < f(x) < 0.5,那么该样本被认为是一个伪负样本。在这里,ϕ ∈ [0, 0.5) 是一个阈值参数。第三步,将伪标记的负样本被添加到标记数据中。第四步利用新数据训练学生模型。第五步,将学生模型视为新的教师模型,重复如上过程。
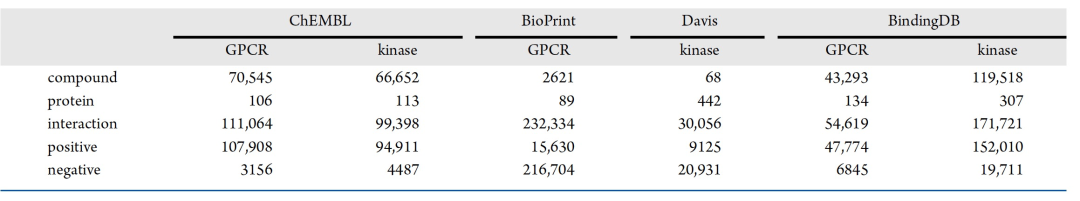
表 1
实验结果
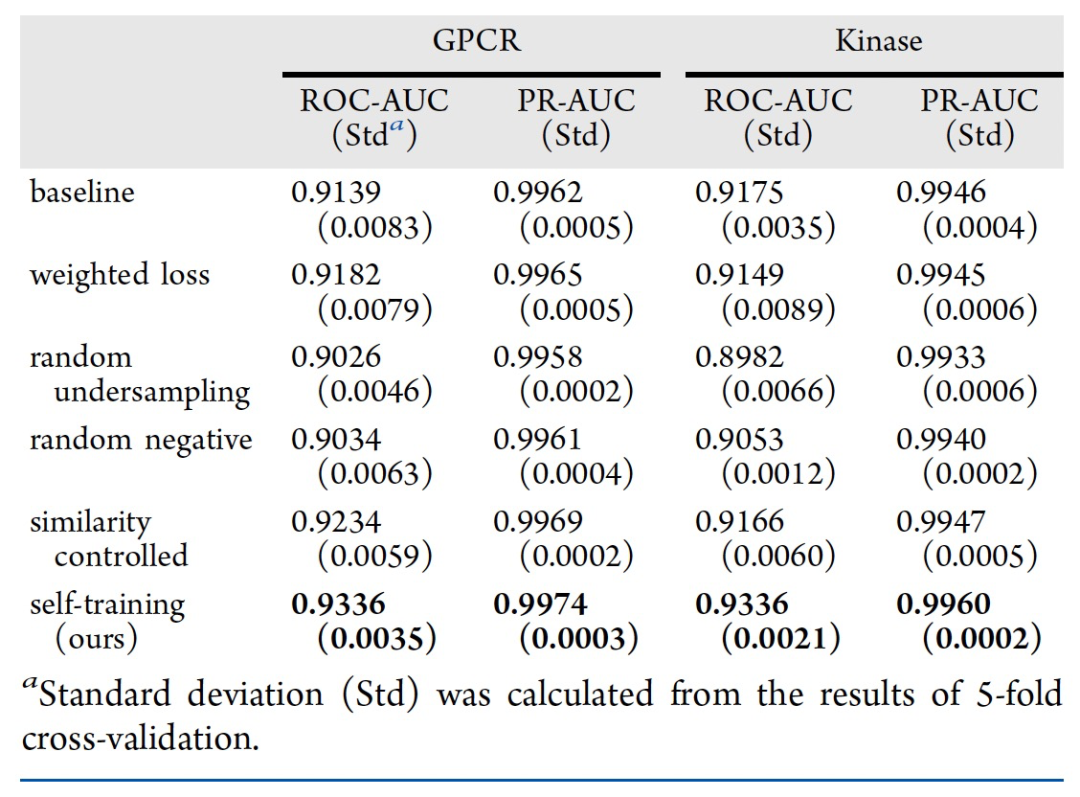
表 2
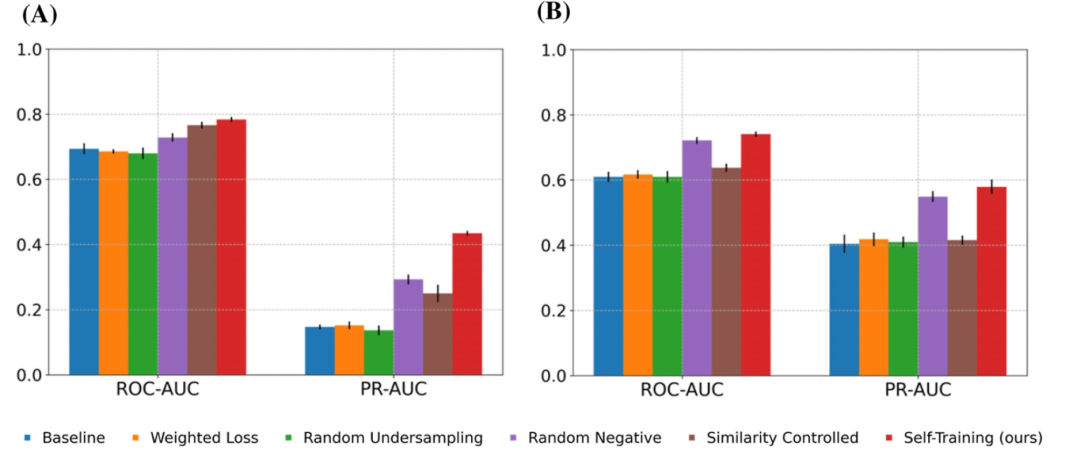
图 2
在ChEMBL上的性能:为了验证自我训练在CPI预测中的有效性,作者对ChEMBL数据集进行了5折交叉验证。如表2所示,在GPCR和激酶家族的内部数据集上,基线模型的平均ROC-AUC分数分别为0.9139和0.9175。通过将自我训练方法应用于不平衡数据,正负样本的数量逐步逼近,并且每个蛋白质中正样本的比例在最终迭代时收敛到0.5。使用更新的数据训练的模型在GPCR(0.9336)和激酶(0.9336)的ROC-AUC分数上表现稍好。这种改进可以归因于数据分布的扩展和数据不平衡的解决。此方法的性能优于随机负样本模型(GPCR:0.9034,激酶:0.9053)。这种改进是可以预期的,因为基于预测分数生成的样本会比随机配对的负样本更可信。模型性能也优于其他方法(表2)。
在外部数据集BioPrint、Davis和BindingDB上的性能:为了评估目前构建模型的泛化能力,作者比较了模型在外部数据集上的性能,其中BioPrint适用于GPCR家族,Davis适用于激酶家族。在这里采用了PR-AUC分数作为主要指标,该指标更适用于评估在由负样本主导的不平衡数据集上的模型性能。模型在BioPrint和Davis数据集上表现出更好的性能,其PR-AUC分数分别为0.4344和0.5792,比基线模型分别提高了28.7%和17.5%(图2)。作者的方法在解决数据不平衡方面也优于其他方法。第二好的随机负样本模型的PR-AUC分数分别为0.2927和0.5491。这些结果表明,自我训练方法可以显著提高模型在外部数据集上的鲁棒性。
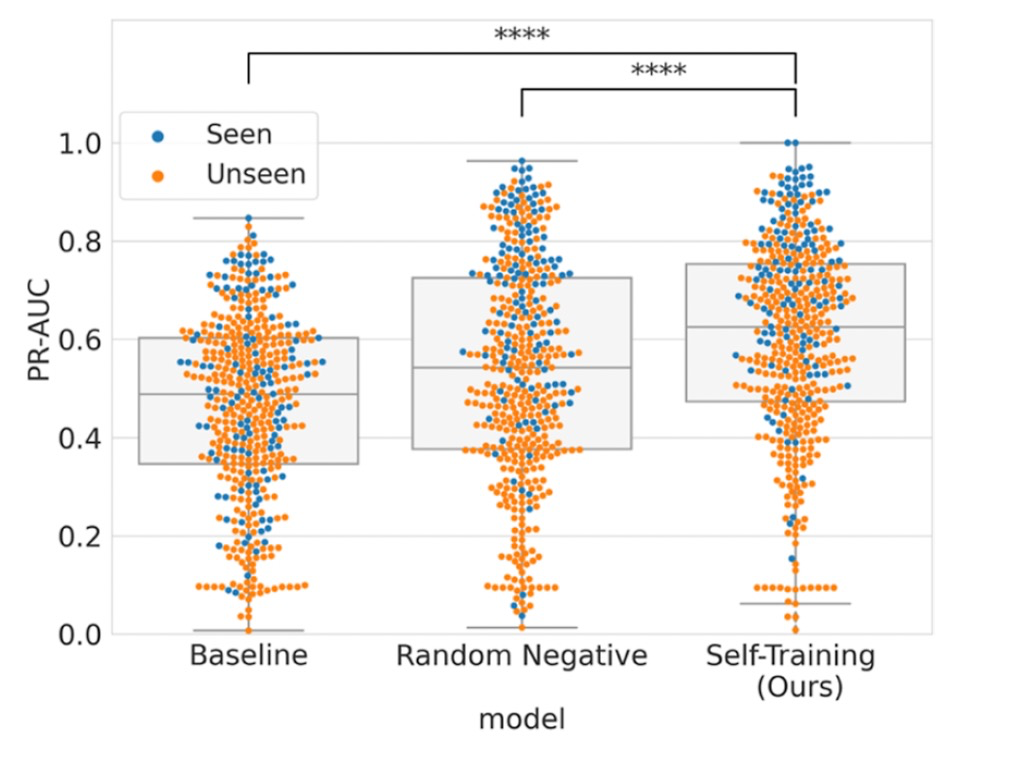
图 3
随后作者使用Davis数据集分析了模型的泛化能力,以评估其在蛋白质水平的预测性能。如图3所示,模型的PR-AUC分数分布(中位数为0.6253)显著高于基线模型和随机负样本模型。分别评估了对已见和未见蛋白质的预测性能,这些蛋白质根据其是否在训练数据集(ChEMBL)中出现进行了分层。对于未见蛋白质,基线模型、随机负样本和作者的模型的平均PR-AUC分数分别为0.4504、0.4877和0.5573,而对于已见蛋白质,这些分数分别为0.4975、0.6658和0.7144。虽然在已见和未见蛋白质之间观察到性能差距,但提出模型在两种情况下均优于其他模型。这一观察进一步支持了方法在提高其泛化能力方面的有效性。对于BioPrint数据集,作者的模型(中位数:0.1695)在蛋白质水平上优于基线模型,而与随机负样本相比并没有发现统计显著性。无法从随机负样本中观察到显著差异可能是因为可用蛋白质数量较少所致。
结论
在这项研究中,作者构建了一种自我训练方法,以改善因CPI预测中的数据不平衡而导致的模型性能和泛化能力问题。模型评估表明,作者方法在处理类别不平衡方面在内部和外部数据集上都优于其他方法。对伪标记样本确定的分数阈值进行分析表明,通过使用经验证的、基于预测值的可信样本,并优先添加靠近边界的信息丰富样本,可以实现改进的泛化能力。此外,作者还验证了自我训练在其他CPI预测模型中的有效性,方法和所得的见解在解决其他基于结构无关方法中的数据不平衡问题方面具有广泛的帮助。研究将为改进CPI预测提供指导,促进在真实世界药物发现中识别新的相互作用。
参考资料
Koyama, T., Matsumoto, S., Iwata, H., Kojima, R., & Okuno, Y. (2023). Improving Compound-Protein Interaction Prediction by Self-Training with Augmenting Negative Samples.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


