作者提出了AttenSyn,一个基于注意力的深度图神经网络,用于准确预测抗癌药物联合用药组合。采用图神经网络模块仅基于分子图提取特征,利用基于注意力的池化模块学习药物对之间的交互信息,加强药物对的表征。结果表明,AttenSyn在预测抗癌协同药物组合方面比最先进的方法表现得更好。
背景
确定协同药物组合对于治疗癌症等各种复杂疾病、同时避免严重的不良药物-药物相互作用至关重要。目前的计算方法高度依赖人工 (handcrafted) 的特征工程,无法更好地学习药物对之间的交互信息,性能相对较低。最近,深度学习方法,特别是图神经网络,在这一领域得到了广泛的发展,并证明了它们解决复杂生物化学问题的能力。
方法
为了将模型的性能与最先进的方法进行比较,作者从O’Neil数据集中收集了药物组合数据集,包含13243个三元组,其中每个三元组包含两种药物和一种癌细胞系。数据集中有31种癌细胞系和38种独特药物,这些药物由24种FDA批准的药物和14种实验药物组成。采用Combenefit工具计算各药物对的协同作用评分。选择10作为阈值对药物对细胞系三元组进行分类。协同得分大于0的组合被认为是积极的(协同),小于0的组合被认为是消极的(拮抗)。药物的SMILES从DrugBank获得。癌细胞系的基因表达数据来自CCLE(Cancer Cell Line Encyclopedia)。基于全基因组读取计数矩阵,通过TPM (Transcripts Per Million)对表达数据进行规范化。
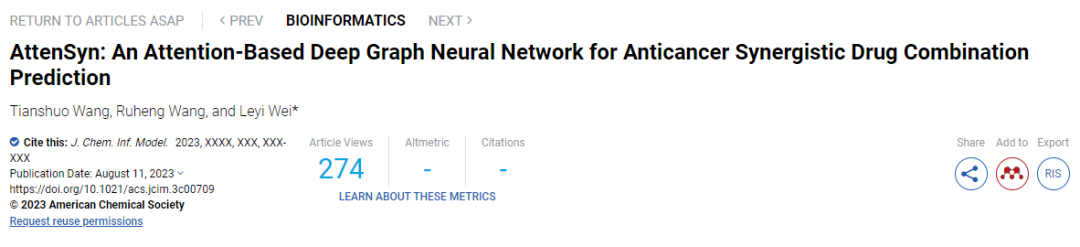
图1显示了AttenSyn的总体架构。该网络架构主要包括三个部分:(1)基于图的药物嵌入模块,(2)基于注意力的池化模块,(3)预测模块。在基于图的药物嵌入模块中,首先将药物SMILES字符串转化为分子图,同时将CCLE得到的细胞系特征加入到药物分子的特征矩阵中。然后,采用图卷积网络(GCN)模型和LSTM模型提取分子图的多分辨率特征。使用基于关注的池化模块学习药物对之间的交互信息,加强药物对的表征。最后,在预测模块中,将药物对的表示与细胞系的特征连接起来,并将其输入到一个全连接的神经网络中,以预测药物对在某些细胞系中的协同作用。
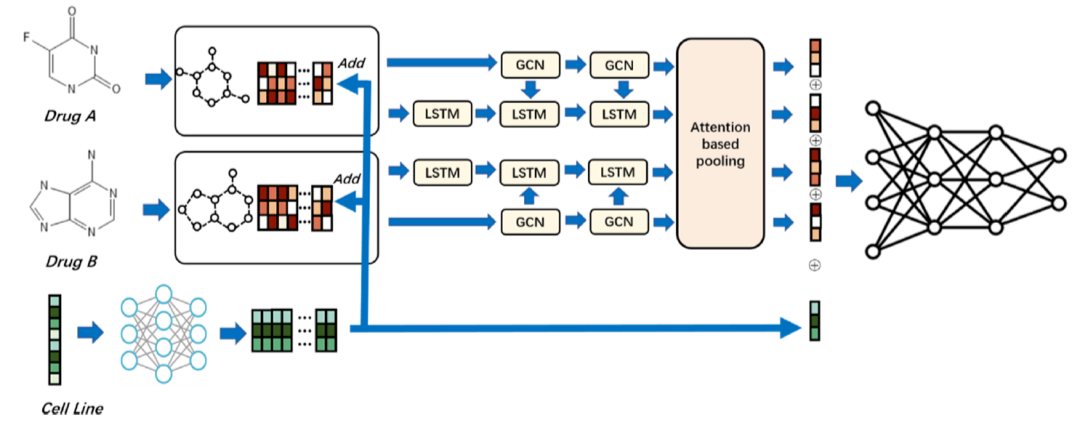 图1 AttenSyn结构
图1 AttenSyn结构
通过使用开源python包RDKit,可以将SMILES字符串转换为一个分子图,其中节点是原子,边缘是化学键。因此,可以用图G = (V, E)来表示一个药物分子,其中V和E分别是节点集和边集。为了聚合具有细胞系信息的分子图,只需将细胞系向量添加到节点的特征中。
为了获得化学子结构的表示,使用了一个GNN模块,该模块使用化学图结构作为输入,并从相邻原子更新每个原子的向量嵌入。因此,每个原子的更新特征向量可以表示化学子结构。
为了获得分子图的多分辨率信息,受MR-GNN的启发,使用LSTM提取图的多分辨率局部特征。使用多个LSTM模型对多个GCN层的特征进行聚合。具体来说,LSTM以从小到大的接受场作为输入,依次接收每个GNN层的输出。
为了提高模型的性能,AttenSyn设计了一个基于注意机制的池,以更好地学习药物对的交互信息,并加强药物的表征。使用基于注意力的池有助于提出的模型考虑化学物质中哪些子结构对协同药物组合的预测更重要。使用基于注意力的池化模块为药物的每个子结构打分,并对所有节点的嵌入进行加权求和,得到图级表示。不仅可以获得药物对之间的交互信息,还可以识别药物的重要化学子结构。
在预测模块中,首先将两种药物的所有特征与细胞系的特征向量进行整合,然后使用多层感知器(MLP)进行预测。模型通过最小化交叉熵损失函数来优化。
结果
作者将AttenSyn与一些具有代表性的方法进行了比较,对比了AUROC, AUPR, 准确率(ACC),平衡准确率(BACC),精确率(PREC),真正例率(TPR)(也就是召回率),KAPPA系数等多个指标,结果如表1所示。AttenSyn在七个指标中的六个中均最高。结果表明,模型在预测药物联合作用方面优于其他方法。
表1 与其他方法对比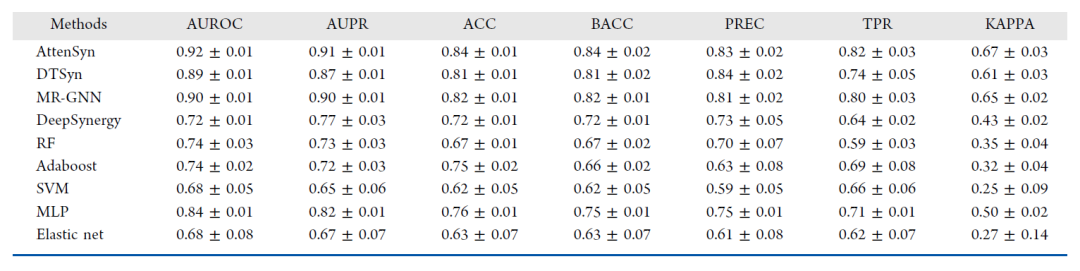
作者设计了模型消融实验,研究基于注意力的池化模块和基于图的药物嵌入模块对模型性能的影响。考虑了AttenSyn的以下变体:(1)提出的AttenSyn;(2) AttenSyn (add);(3) AttenSyn(mean);(4) AttenSyn (SAG);(5) AttenSyn(无图)。具体来说,AttenSyn (add)使用全局添加池方法,而不是基于注意力的池化。AttenSyn (mean)使用全局平均池化方法,而不是基于注意力的池化。而AttenSyn (SAG)则采用SAGPooling引入的方法计算自我注意分数,以更新其节点的嵌入,然后添加它们以获得图级表示。AttenSyn(无图)去除了基于图的药物嵌入模块。
从图2中可以看出,与其他版本的AttenSyn相比,原始AttenSyn的性能更好。此外,AttenSyn (add)和AttenSyn (SAG)的结果略优于AttenSyn (mean)的结果。这可能是因为,对于分子图,全局平均池化方法将每个子结构视为同等重要,并且简单地平均所有节点的嵌入。具体来说,SAGPooling使用自注意机制计算每个子结构的分数,并根据注意分数对所有节点的嵌入进行加权和,得到最终的图级表示。然而,在所有7个指标中,AttenSyn (add)和AttenSyn (SAG)之间没有显著差异。原因可能是SAGPooling方法仅使用单个药物的分子图来获得每个子结构的关注分数,而不是使用药物对的交互信息来计算每个子结构的重要性。同时,提出的基于注意力的池化模块使用药物对的交互信息,因此可以获得比其他池化策略更好的性能。此外,提出的AttenSyn优于无图的AttenSyn,证明了基于图的药物嵌入模块的有效性。 图2 消融实验
图2 消融实验
作者还进行了案例分析,探索药物对中哪些子结构对协同药物组合预测贡献最大。AttenSyn通过模型的注意机制可视化了药物对中最重要的子结构,并以不同的颜色将分数可视化。图3a-c为随机选取的三对药物对在细胞系作用中的可视化结果,颜色越深表示子结构越重要。模型检测到的化学结构之一是酰胺基团,它在生物分子的组成中起着关键作用,包括许多临床批准的药物。酰胺普遍存在于重要的医药化合物中,不仅因为它们特别稳定,而且因为它们是极性的,这使得含酰胺的药物能与生物受体和酶相互作用。这一结果表明模型具有良好的可解释性。
为了进一步探索训练过程中子结构注意分数的变化,作者还可视化了模型训练前药物对的注意分数分布。如图3d-f所示,模型训练前的注意分数分布更加均匀,说明模型不能注意到重要的结构。然而,随着训练的进行,模型认为某些特定结构比其他结构更重要。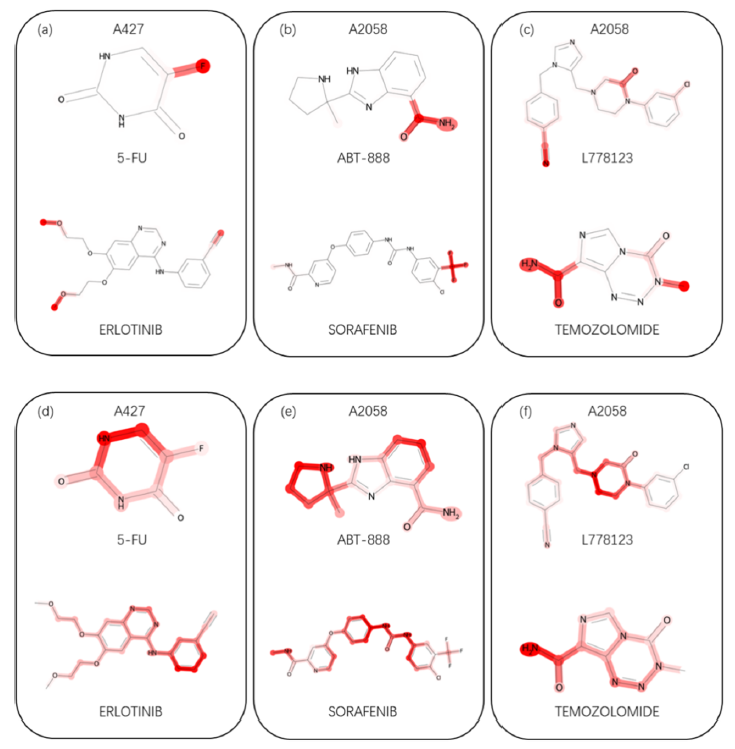 图3 案例分析为了从特征分析中进一步解释深度学习在训练过程中的工作原理,并直观地展示所提出的AttenSyn的特征学习能力,作者可视化了两种细胞系(即A375和HT29)中药物组合的嵌入。具体而言,分别使用t-SNE和UMAP将模型中提取的药物对的嵌入空间降至二维空间,如图4所示。在图4的每个子图中,每个点代表一个药物对,并用不同的颜色来区分协同药物组合和拮抗药物组合类别。不同类别下的点的可分辨性越强,分类效果越好。如图4a所示,通过对t-SNE进行降维处理,两类样本在训练模型的特征空间中的分布比未经过训练的模型更加清晰,说明模型可以从不同的类样本中捕捉到判别性和高质量的特征。通过UMAP降维得到的模型也有类似的结果。从图4b中可以看出,与没有训练过程的模型相比,经过训练过程的模型学习并获得了更多可区分的特征。
图3 案例分析为了从特征分析中进一步解释深度学习在训练过程中的工作原理,并直观地展示所提出的AttenSyn的特征学习能力,作者可视化了两种细胞系(即A375和HT29)中药物组合的嵌入。具体而言,分别使用t-SNE和UMAP将模型中提取的药物对的嵌入空间降至二维空间,如图4所示。在图4的每个子图中,每个点代表一个药物对,并用不同的颜色来区分协同药物组合和拮抗药物组合类别。不同类别下的点的可分辨性越强,分类效果越好。如图4a所示,通过对t-SNE进行降维处理,两类样本在训练模型的特征空间中的分布比未经过训练的模型更加清晰,说明模型可以从不同的类样本中捕捉到判别性和高质量的特征。通过UMAP降维得到的模型也有类似的结果。从图4b中可以看出,与没有训练过程的模型相比,经过训练过程的模型学习并获得了更多可区分的特征。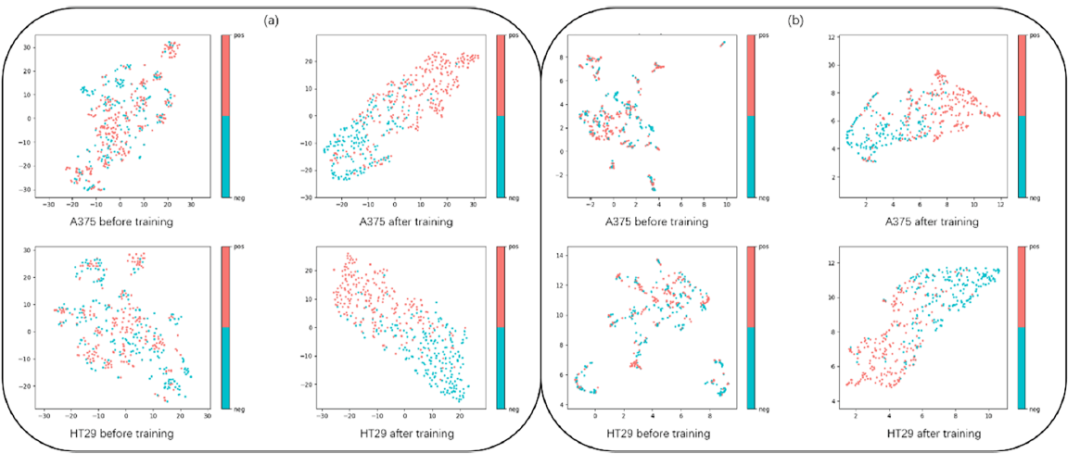 图4 在两种细胞系上的可视化
图4 在两种细胞系上的可视化
总结
在这项研究中,作者开发了一种新的基于注意力的深度图神经网络AttenSyn来预测抗癌药物联合用药的协同作用,这是快速虚拟药物筛选和药物开发的关键一步。
具体而言,首先生成药物的分子图,并利用基于图的药物嵌入模块分别提取药物对的结构信息。然后设计基于注意力的池化模块,学习更好的交互信息,加强药物对的表征。在基准数据集上进行的综合实验表明,该方法比对比方法具有更好的预测性能。为了克服基于深度学习的模型中“黑箱”的局限性,用注意力机制探索了模型在训练过程中在发现药物的关键子结构和进行特征分析方面所学习到的内容,这为模型提供了良好的可解释性,并为理解药物协同作用机制提供了生物学见解。
然而,模型仍然存在一些缺点。例如,生物网络已经证明了其在药物协同作用预测中的有效性,而本文只使用分子结构信息和细胞系特征,而不使用生物网络信息等额外信息进行预测。未来可以考虑引入生物网络来提高抗癌药物联合用药预测的性能。
参考文献[1]Wang et al. AttenSyn: An Attention-Based Deep Graph Neural Network for Anticancer Synergistic Drug Combination Prediction. J Chem Inf Model. 2023
——— End ———



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


