编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Leo Klarner团队的一篇论文。加速发现新型和更有效的治疗方法是一个重要的药物学问题,深度学习在其中扮演着日益重要的角色。然而,现实世界的药物发现任务通常具有标记数据的稀缺性和显著的协变量转移,这对标准的深度学习方法构成了挑战。作者提出了Q-SAVI,一种概率模型,能够通过将数据生成过程的显式先验知识编码为函数的先验分布,为研究人员提供一种透明且基于概率原理的方式,以编码数据驱动的建模偏好。

发现能够安全有效地治疗被忽视疾病或对抗多药耐药病原体的新型药物候选化合物,是一个具有重要科学和社会意义的挑战性生物医学研究问题。利用现代深度学习算法准确预测临床相关的分子性质,从而减少时间和资源密集型实验的需求,有潜力显著加速有希望的和创新的药物发现中的化学前导化合物的开发。
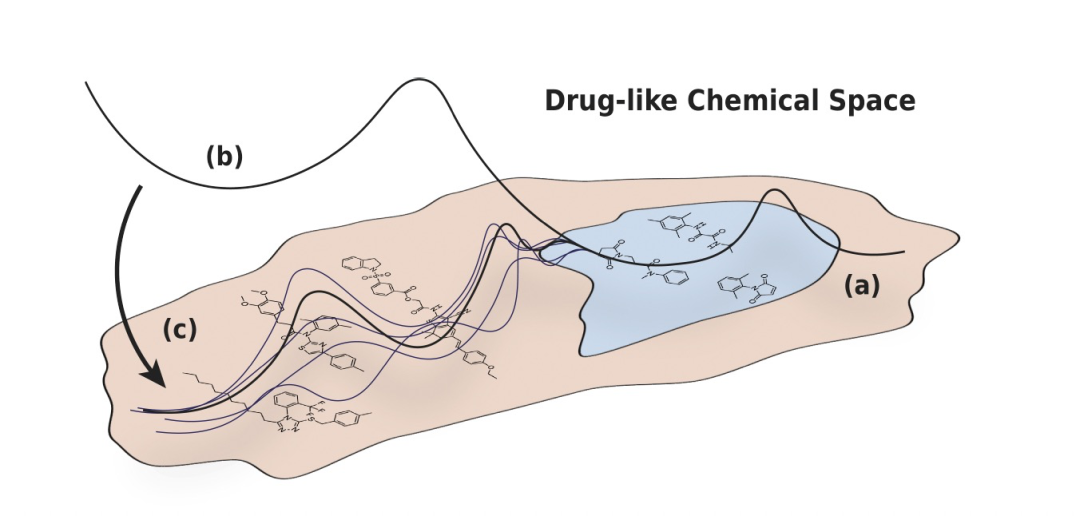
图 1
实际早期药物发现研究的一个关键特点是将预测模型应用于结构或功能与已经研究过的分子不同的新化合物(见图1)。在这样的外推环境中,机器学习系统的实际实用性取决于它们在以下方面的能力:(a)对化学空间未知领域进行稳健的泛化;(b)通过生成良好校准的预测不确定性估计,当它们无法做到时可靠地指示。然而,标准的深度学习算法在协变量转移下往往表现不佳,会生成不正确且高度不校准的预测。这在早期药物发现的背景下尤其成问题,因为实验标签的获取成本昂贵,因此只适用于一小部分通常高度偏倚的化合物。
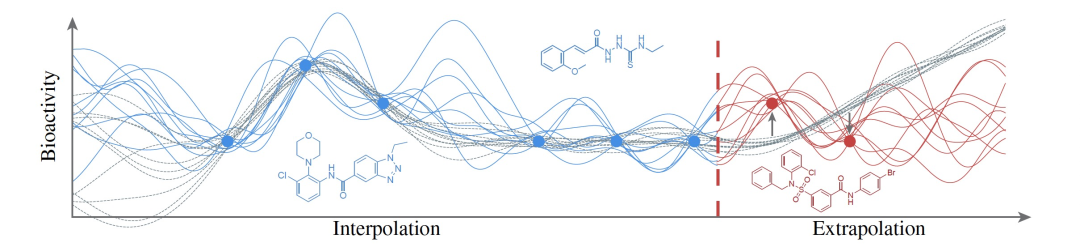
图 2
为了在这种资源受限、数据稀缺的情况下改进深度学习算法的预测性能,作者希望利用有关问题领域的相关先验知识,以指定归纳偏见,使某些预测函数比其他函数更有可能。赋予神经网络有用的归纳偏见的常见方法包括:(a)在更大的、可能是无标签的数据集上对其进行预训练,以及(b)调整其架构,以反映其输入域的适当不变性。然而,这些方法只是将显式建模偏好转化为神经网络假设空间上的约束的间接方法,通常不够精确。作者提出了一种替代方法。为了将数据生成过程的领域信息先验知识编码到神经网络训练中,作者在定量结构活性映射空间中的一组精心选择的上下文点上指定了一个先验分布,并在生成的概率模型中执行变分推断(见图2)。
预测性质以发现药物
小分子药物发现的总体目标是识别调节感兴趣的生物靶点并引发治疗有益反应的化合物。不幸的是,发现一个有希望的候选化合物以进入临床试验的过程是困难且往往不成功的,因为可行的药物样式分子的搜索空间。这又受到药物化学固有实验限制的影响,这意味着只能为化合物的一个微小子集获得标签。自然地,这引发了对在可用数据训练监督式机器学习算法的广泛兴趣,以预测广阔的化学空间中化合物的性质。在实践中,用于训练的子空间的组成在很大程度上由实证考虑因素确定,如化合物的可用性以及药物化学家的偏好和直觉,从而导致高度偏倚的子样本。这意味着,为了可靠地预测新颖且在科学上有趣的化合物的性质,机器学习算法在外推区域内表现良好是至关重要的。由于这个要求与分布内泛化不同,标准的正则化方法可能不会有效。
Q-SAVI
监督学习设置目标是在从化学空间的偏倚子集中抽取的N个独立同分布样本的实验标签上训练机器学习模型,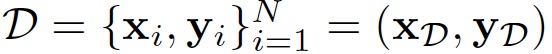 。X为输入数据,Y为标签。与将后验推断问题表述为找到随机参数
。X为输入数据,Y为标签。与将后验推断问题表述为找到随机参数 的后验分布不同,作者遵循先前研究人员Rudner提出的方法,将随机神经网络中的变分推断重新构造为在潜在随机函数
的后验分布不同,作者遵循先前研究人员Rudner提出的方法,将随机神经网络中的变分推断重新构造为在潜在随机函数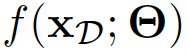 上找到后验分布。具体而言,参数空间贝叶斯推断问题可以表示为:
上找到后验分布。具体而言,参数空间贝叶斯推断问题可以表示为:
基于潜在随机函数的推断为:
现在将扩展这个函数空间的贝叶斯推断形式,以定义一个概率模型,该模型能够在训练点的有偏子集之外,集成完整输入空间的先验知识。具体而言,作者将上面的概率模型扩展到随机变量 ,从而得到后验分布:
,从而得到后验分布:
在上述公式中指定的推断问题在计算上是难以处理的。因此可以将其变分地表述为:
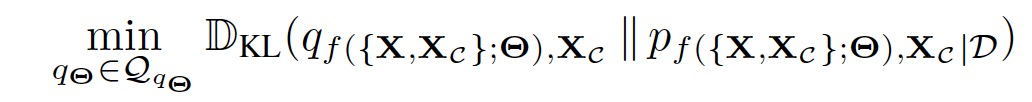
随后经过简化,可以变为:
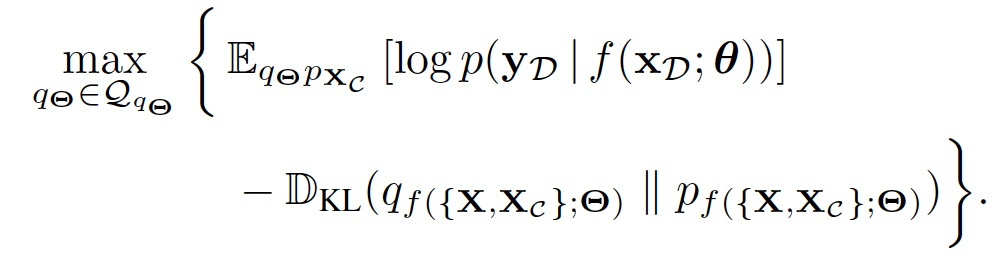
实验部分
训练和评估QSAR模型面临的一个根本障碍是缺乏具有高质量标签的足够大的数据集。尽管存在公开可用的生物活性数据集合,但它们通常直接来自高通量筛选(HTS)数据库,例如PUBCHEM、CHEMBL或TOXCAST,而没有经过显著的过滤或预处理。虽然这种方法最大限度地增加了可用数据点的数量,但它可能会降低模型性能比较的判别力。例如,上述库中的主要测量结果通常是确认性剂量响应筛选的已知问题,它们通常包含大量可复制的假阳性读数,这是由分子亚结构引起的,这些亚结构干扰了测定的读数系统。在没有进一步处理的情况下使用这些数据会导致算法只能测试其记忆这些亚结构的能力,而无法评估有意义的外推性能。为了筛选出质量足够高的数据集,以便进行有信息量的预测模型比较,作者使用了生物活性和毒性筛选的测量元数据来优先考虑一些数据点进行进一步的检查。在调查与最有前途的数据集相关的出版物之后,作者选择了一个高质量的筛选任务,该任务旨在抑制肝期疟原虫的发育,并对其进行了进一步处理。具体来说,作者检索并重新处理了原始测量数据,以去除可能的假阳性,从而得到一个二进制分类数据集,其中包括7,301个不活性分子和849个活性分子,每个分子均进行了生物重复测量,并通过一组质量保证的对照筛选来确认为真正的阳性或阴性。
为了评估不同的训练-测试数据集划分在引起协变量移位和标签移位方面的影响,作者确定了一组适合的两样本检验统计量,并使用它来量化相应训练和测试数据集 Dtr = (Xtr; ytr) 和 Dte = (Xte; yte) 的边际协变量和标签分布的不相似性。由于 ytr 和 yte 都由抗疟活性的二进制指示器组成,因此可以使用像 Fisher 的精确检验这样的成熟分类统计方法。在接下来的分析中,作者使用其负对数 p 值作为标签移位的标量指标。定义用于量化两组分子之间协变量移位的相应统计量更具挑战性,因为它们构成了不重叠的离散对象集合。为此,作者使用最大平均偏差(MMD)指标来量化两组分子样本之间的差异。
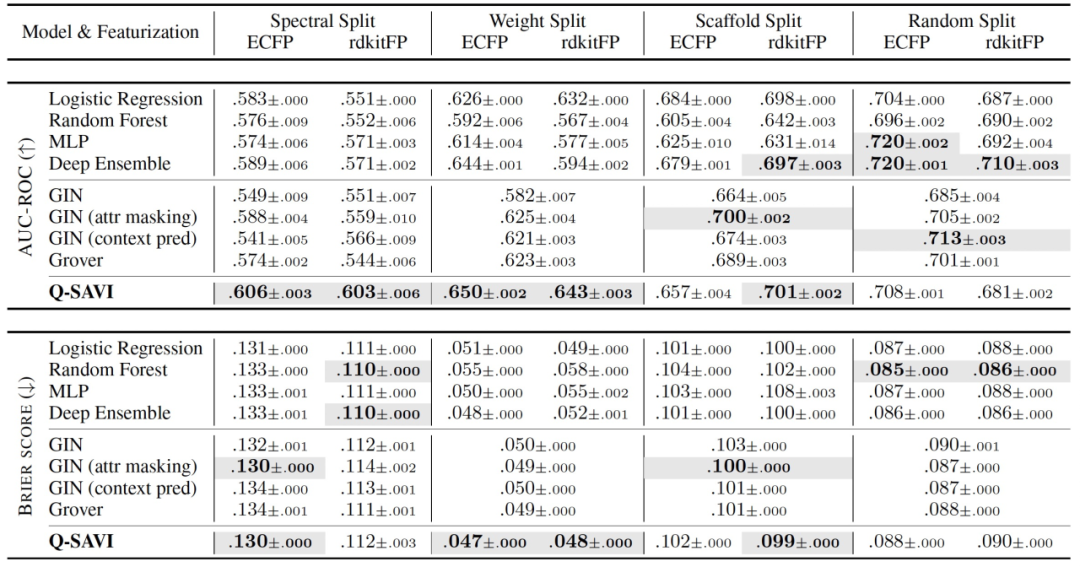
表 1
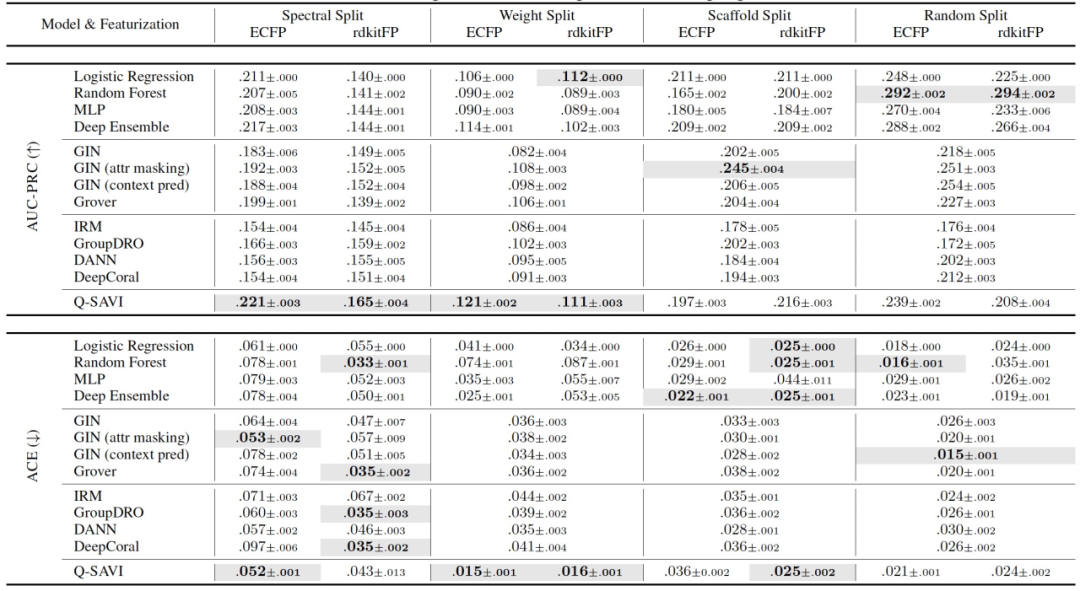
表 2
在表1和表2中呈现的预测准确性和校准度指标表明,在分布外情况下,Q-SAVI实现了显著的性能提升。在具有最强协变量和标签移位的光谱和分子量分割设置中,Q-SAVI在预测准确性方面明显优于所有其他算法,并且在统计上显著。类似地,在分子量分割上,其预测不确定性估计显著优于所有其他算法,并且在基于ECFP的分割上,它的预测不确定性估计也显著优于大多数其他算法。在较少数据移位的骨架结构和随机分割中,相对简单的机器学习算法(例如,随机森林和深度集成)以及更复杂的基于自监督预训练的方法始终实现了最佳的预测性能。与先前研究人员的经验观察一致,IRM、GroupDRO、DANN和DeepCoral等最初用于图像的领域适应和泛化技术在大多数分割和特征化中表现较差。
结论
为了构建一个稳健的实验环境,并在提议的方法中进行实际有意义的评估,作者精心预处理了高质量的生物活性数据集,并探索了不同领域特定的统计量,以量化此设置中的分布变化。利用这些统计数据来突显常用的随机和骨架划分能够引发有意义的协变量和标签变化的有限范围,作者在两种基于分子权重和谱聚类的替代方法的基础上构建了具有挑战性的训练-测试划分。借助这个外推评估设置,作者证明了使用Q-SAVI为神经网络提供与药物样化学空间有关且情境化的信息,显著提高了神经网络模型的预测准确性,优于一系列最先进的自监督预训练、集成和领域适应技术。
参考资料
Klarner, L., Rudner, T. G., Reutlinger, M., Schindler, T., Morris, G. M., Deane, C., & Teh, Y. W. (2023). Drug Discovery under Covariate Shift with Domain-Informed Prior Distributions over Functions.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


