编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Arijit Roy团队的一篇论文。生成人工智能算法已经成功地在探索庞大的化学空间和设计新颖多样的分子方面发挥作用。人工智能用于药物样性质的预测模型的发展引起了业界相当大的兴趣,这些模型有潜力减少药物候选物的后期淘汰或预测新型AI设计的分子的性质。同时,理解功能基团对这些性质的贡献,并对其进行修改以获得性能优化的前导化合物至关重要。因此,越来越多的人对可解释性性质预测模型的开发产生了兴趣。然而,目前的可解释方法大多是基于原子的,通常只显示了一个片段的一部分是显著的。为了解决上述挑战,作者开发了一种新颖的领域感知分子分解方法,称为后处理的BRICS(pBRICS),它可以将小分子分解为它们的功能基团。作者基于此开发了多任务模型来预测各种分子性质。

基于人工智能(AI)算法的出现已经彻底改变了完整的药物设计过程。在生成式AI领域取得了显著进展,通过有效地探索化学空间来设计新颖的药物样分子。一些研究已经展示了这些模型在真正加速早期药物发现过程和药物样分子设计方面的潜力。虽然在生成针对感兴趣的靶蛋白的新颖药物样小分子时,有必要探索潜在的广阔化学空间,但也必须优化各种物理化学性质和后期药物样性质,包括吸收、分布、代谢、排泄和毒性(ADMET),以减少新药分子的后期淘汰率。
除了衡量相似分子的性能之外,模型预测的可解释性还可以在引导优化方面发挥作用。每个亚结构或分子片段对所预测性质的贡献能力的量化能够引导药物化学家优化引导分子。分子的输入表示的选择可以影响模型学习的内容,从而影响模型预测的解释。用于开发预测模型的几种广泛使用的输入表示包括SMILES、基于原子级分子图的指纹、基于功能团频率的指纹以及基于描述符的特征向量。使用上述每种输入表示开发的模型,还可以使用基于梯度的解释方法以及基于非梯度的解释方法提取相应的模型解释。分子图表示的解释通常涉及着色方案,以突显梯度变化的大小。然而,这些方法通常为功能团产生部分解释。例如,只有少数环系统和功能团的原子被突出显示为重要。为了解决这个问题,功能团水平的分子图表示可能是一个解决方案,其中功能团是节点,断裂的键是边。然而,大多数现有的分子片段化方法存在一些局限性,例如BRICS、RECAP和SynDiR,这些方法导致在分子片段化时产生单独的原子,不能处理非常小的分子,并且无法对分子中核心骨架的所有取代基进行片段化。
在这项研究中,作者通过一种基于梯度的解释方法和一种新颖的基于片段的分子图表示,解决了量化分子片段级别对性质预测的贡献的挑战。作者提出了一种新颖的分子片段化方法,称为pBRICS,用于对任何新型小分子进行精细的片段化。基于pBRICS的片段获得的分子图被用来训练图卷积网络(GCN)模型,用于预测超过40种不同的ADMET性质。
使用后处理BRICS(pBRICS)方法对分子进行片段化
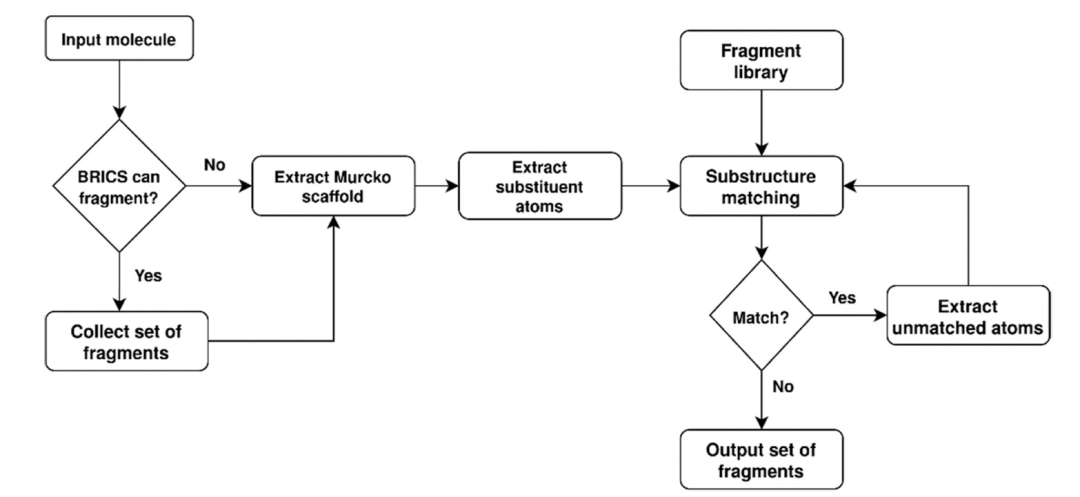
图 1
在这项工作中,作者引入了一种名为pBRICS的新型分子片段化方法。pBRICS方法对常用的BRICS方法得到的结果进行进一步的分子片段化。BRICS方法考虑了16种化学环境及其对应的分子片段原型,用于分解分子中的每个键。由于分子可能存在更多的断裂规则,BRICS方法能够比RECAP分子片段化方法分解更多的分子。pBRICS方法试图迭代地分解分子,以便对骨架的最小可能取代基进行枚举。如果分子可以通过BRICS方法进行分解,则所得到的分子片段主要被分类为骨架和取代基。分子或片段的骨架是基于Bemis−Murcko框架的定义。与取代基相对应的原子进一步与手动策划的片段库进行匹配,以获得最大可能的片段匹配,并且该过程会迭代,直到不能再进行更多的片段匹配为止。如果无法通过BRICS方法分解分子,则会对应于分子的骨架和取代基的原子进行枚举,并且将迭代分子片段化过程应用于来自BRICS方法的片段之外的取代基原子(图1)。
从pBRICS方法获得的片段构建分子图
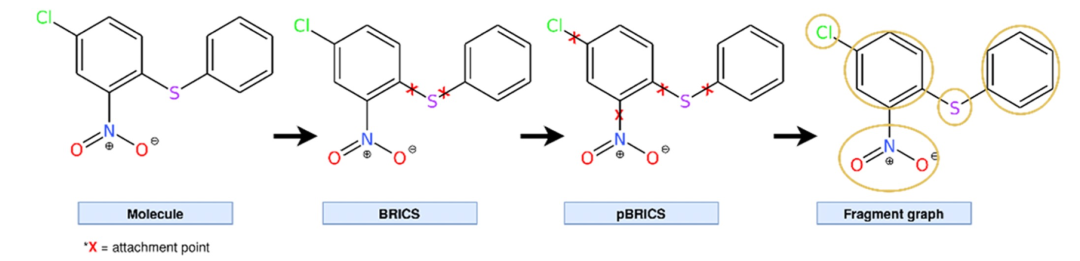
图 2
从pBRICS获得的片段被用于构建一个领域感知的片段级图表示。为了生成这个图,片段被视为节点,并且如果两个片段在分子中由至少一条键连接,则在它们之间定义一条边(参见图2)。一旦图构建完成,下一步是为图中的每个节点定义特征向量。对于每个节点,MACCS的167种表示被视为特征向量。连接片段的键的类型被编码为一个值,用于在聚合邻居信息时作为边权重使用。
图网络模型构建
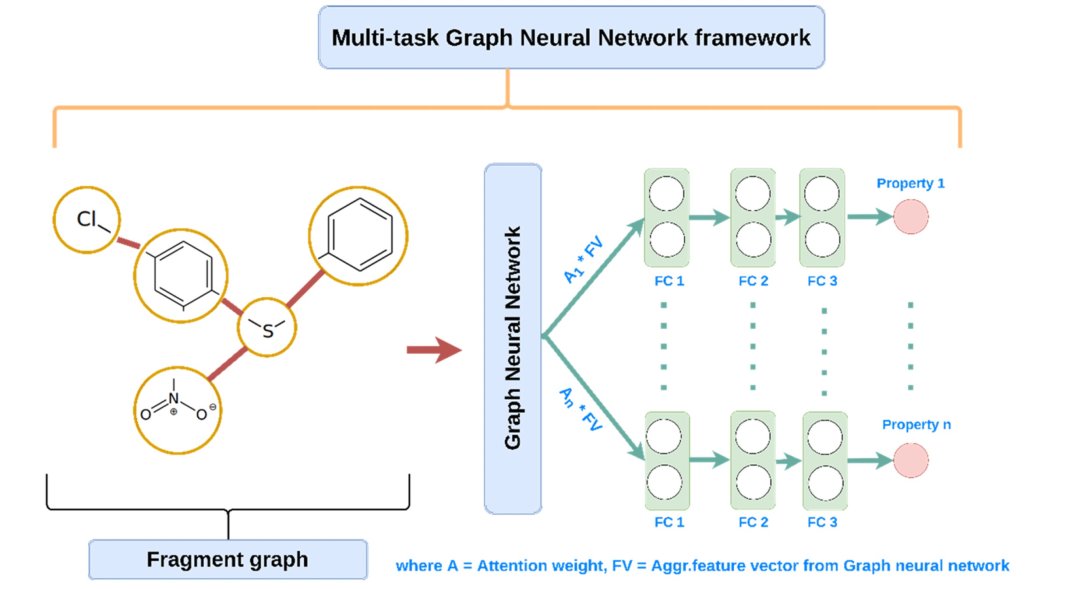
图 3
多任务图神经网络模型(图3)被应用,使得模型能够从相关任务中受益,并提高对于数据较少任务的预测能力。与单任务预测模型不同,多任务模型可以同时预测分类和回归属性,不需要为感兴趣的每个任务(属性)独立构建单独的模型。在多任务模型中,一批片段图首先通过两层GCN,每层大小为64。每个图的最终GCN层的聚合特征向量(FV)与其相应的注意权重相乘,结果向量被送入一个独立的全连接(FC)层堆栈(每个属性一个FC堆栈)。在这里,所有的FC堆栈共享最终GCN层的聚合特征向量,使得模型能够学习涉及的所有属性组合之间的共同特征。
实验部分
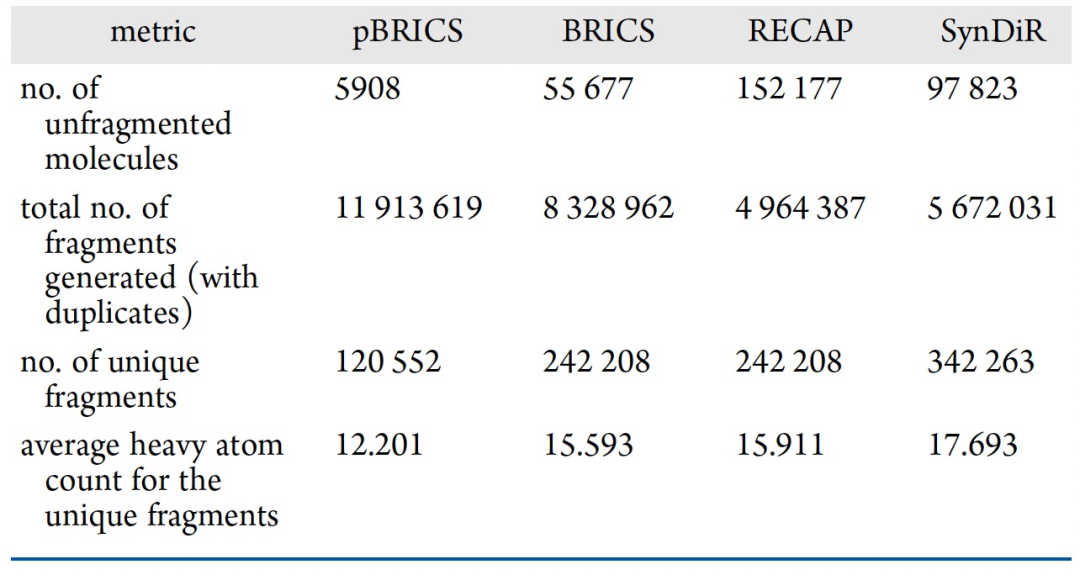
表 1
本研究提出的pBRICS分子片段化方法与现有的分子片段化方法,包括BRICS、RECAP和SynDiR,在基准数据集ChEMBL上进行了比较,比较采用了前文定义的四个指标(表格1)。从比较结果中可以看出,pBRICS相比于RECAP、BRICS和SynDiR方法,可以分解出16倍、5倍和10倍的分子片段。然而,pBRICS生成的独特分子片段数量最少,重原子数最小,这是由于其细粒度的分子片段化过程所导致的,从而产生了更加通用的分子片段库。
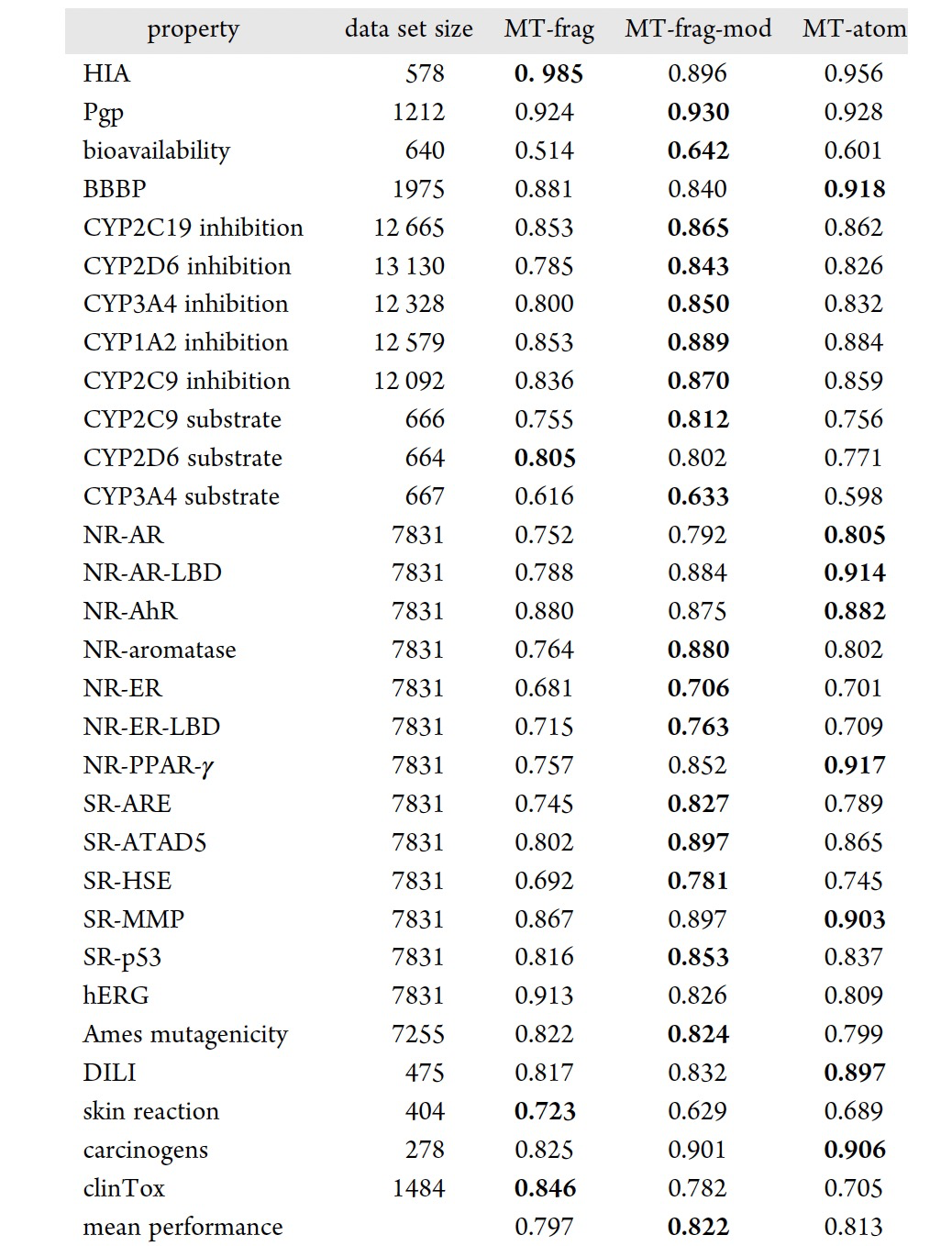
表 2
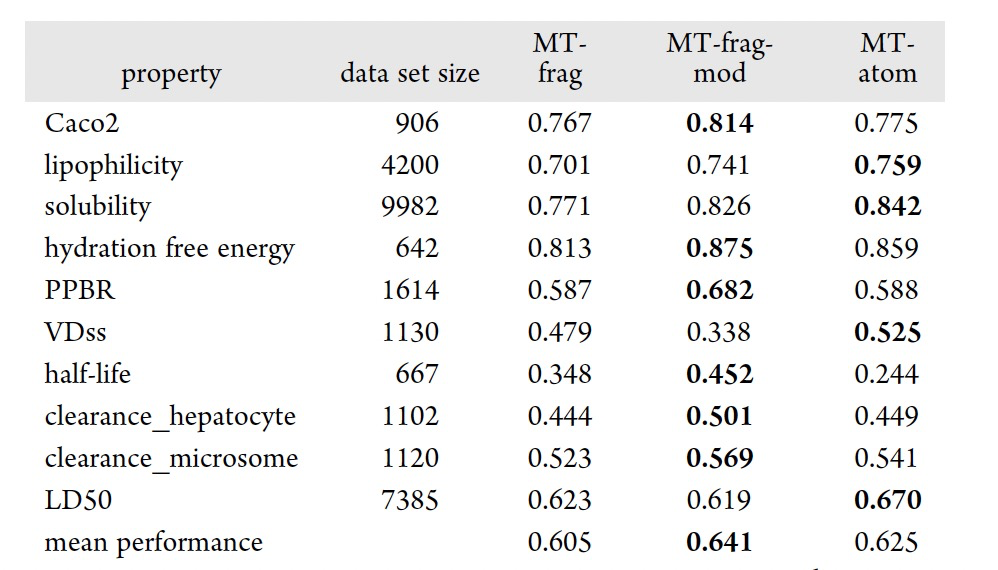
表 3
如上文所述,使用基于pBRICS的领域感知分子图表示来训练多任务模型,用于预测40个对于小分子被视为药物分子至关重要的性质。在这40个性质中,有30个基于分类任务,而另外10个则基于回归任务。一个单独的基于GCN的多任务模型,被称为MTFrag,被用于所有40个性质的训练。接下来,为了检查它们是否能够提高性能,将分别训练两个独立的多任务模型,来处理分类任务和回归任务,模型被标记为MT-Frag-Mod。MT-Frag和MT-Frag-Mod之间的主要区别在于前者同时训练了分类和回归任务。在表格2和3中,MT-Frag和MT-Frag-Mod分别与基于原子的多任务图注意框架进行了比较,该模型在表格2和3中分别被标记为MT-Atom。ADMETLab2.0中的MT-Atom被重新训练,使用了与MT-Frag和MT-Frag-Mod相同的训练和测试数据集,以便进行公平比较。在分类性质方面,经过超参数调优,MT-Frag-Mod在平均性能上达到了82.2%,比MT-Atom模型(81.3%)高出1%,比MT-Frag模型(79.7%)高出2.4%。在回归性质方面,MT-Frag-Mod的平均性能达到了64.1%,比MT-Atom模型(62.5%)高出1.5%,比MT-Frag模型(60.5%)高出4%。MT-Frag-Mod性能的提升可以归因于对分类和回归性质进行分别训练。对于研究考虑的40个性质,观察到MT-Frag-Mod在24个性质中取得了最佳表现,而MT-Frag仅在4个性质中取得了最佳表现。另一方面,MT-Atom模型在40个性质中有12个取得了最佳表现。为了确定这三个模型(MT-Frag、MT-Frag-Mod和MT-Atom)之间是否存在显著差异,以0.05的显著性水平进行了t检验。MT-Frag-Mod和MT-Atom的平均性能相当,并且与MT-Frag不同(见表2和表3)。
预测模型的一个主要挑战是准确地预测来自MMP数据集(仅通过单个功能团的微小变化但具有对比性质的配对)的小分子,以使其解释与模型预测良好地相关。BBBP性质的MMP数据集包括102个独特的分子对,以及它们相应的真实标签。为了检查模型在MMP数据集上的性能,从所有MMP条目的分子中组合形成一个独特的110个分子的集合。所识别的MMP被用于比较两个经过训练的模型,即MT-Frag-Mod和MT-Atom(基于ADMETLab2.0),以及一个外部模型,SwissADME。该提出的方法被发现可以准确地预测MMP数据集中37.3%的分子,比ADMETLab2.0的预测率高出7.3%,比swissADME的预测率高出5.5%。
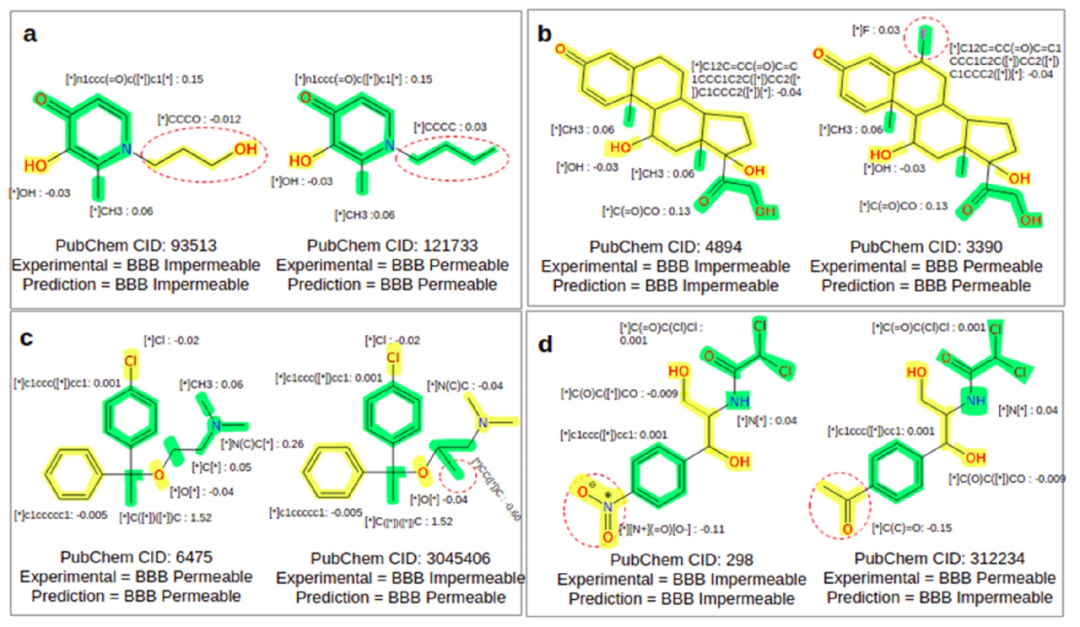
图 4
为了获得解释,Grad-CAM算法被应用于MMP数据集中的所有分子,以获取与每个节点相对应的梯度权重。首先,根据模型预测使用Grad-CAM方法计算所有分子的每个片段的贡献。接下来,从所有正确分类为正样本的分子中收集所有独特片段的Grad-CAM值,并取平均。平均化解决了单个Grad-CAM值周围的变异性问题,并有助于在MMP对中得出适当的决策。分子根据每个独特片段的平均Grad-CAM值进行着色(图4)。在BBB通透性的情况下,BBB通透性和非通透性片段分别属于正类和负类。正类和负类的片段分别以绿色和黄色显示(图4)。图4展示了数据集中两个正确预测的MMP对的示例,其Grad-CAM值与其标签(经实验验证的BBB通透性或非通透性)相匹配。在图4a中的MMP对中,将甲基基团替换为羟基基团将分子从BBB通透性转变为BBB不通透。在图4b中的情况下,将卤素(氟)替换为氢原子将分子从BBB通透性转变为BBB不通透。值得注意的是,这些转变在实验上也被证明会破坏分子的此项性质。
结论
作者提出了一种名为pBRICS的新型分子片段化方法,以捕捉化学家的直觉。使用pBRICS片段训练的模型性能在与基于原子表示的模型相比时更高。同时,针对分类和回归任务分别训练的多任务性质预测模型在所有40个ADMET性质上显示出了显著的改进。从训练模型获得的解释对药物化学家来说易于理解,可以在药物类性质优化方面发挥特别的作用。
参考资料
Vangala, S. R., Krishnan, S. R., Bung, N., Srinivasan, R., & Roy, A. (2023). pBRICS: A Novel Fragmentation Method for Explainable Property Prediction of Drug-Like Small Molecules. Journal of Chemical Information and Modeling.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


