今天为大家介绍的是来自广州国家实验室陈红明团队发在Briefings in Bioinformatics上的一篇论文“3D based generative PROTAC linker design with reinforcement learning”[1]。PROTAC 全称为 proteolysis-targeting chimeras (蛋白水解靶向嵌合分子),是一种杂合双功能小分子化合物,由三部分组成:靶蛋白配体(warhead)、连接子 Linker、和 E3 连接酶配体(E3-ligand),结构中两个配体之间通过 linker 相连,从而形成“三体”复合物(PTS):warhead-Linker-E3-ligand。它通过将靶蛋白和细胞内的 E3 泛素连接酶的距离拉近,利用泛素-蛋白酶体途径特异性地降解靶蛋白。由于PROTAC相对较大的分子量,以及维持其在蛋白口袋中结合模式的复杂性,合理的设计多样性的linker极具挑战性。已有的PROATC的linker生成方法只能生成1D或2D的linker,并没有考虑该linker对三元复合物PTS的影响,无法衡量其在PTS内的合理性。本文提出了一种新的3D的Linker生成模型PROTAC-INVENT,该模型不仅可以生成PROTAC的2D结构,还可以生成PROTAC与靶蛋白和E3连接酶的三维推定结合构象PTS。该模型在强化学习(RL)框架下训练,使PROTAC结构的生成偏向于预定义的2D和3D属性。并通过实例验证了该模型在生成合理的PROTAC三维构象方面的实用性。另一方面,该算法的工作流也可以作为专门针对PROTAC的对接协议。

模型概览
PROTAC-INVENT以一对SMILES片段(warhead和E3-ligand)以及一个参照的PTS作为输入,返回生成的linker和所形成的PROTAC在PTS口袋的结合构象(如图1所示)。PROTAC-invent集成了两个模块(生成模型和打分)。首先,预训练的生成模型产生Linker的SMILES形式,与warhead和E3-ligand一起形成完整的PROTAC的SMILES。然后利用RL搜索化学空间,优化分子性质。在其中实现了保持warhead和E3-ligand构象不变(与参照PTS相比)的前提下,将PROTAC由2D转成3D,并给出docking score。然后利用基于2D和3D的综合评分来驱动RL(如图2)。
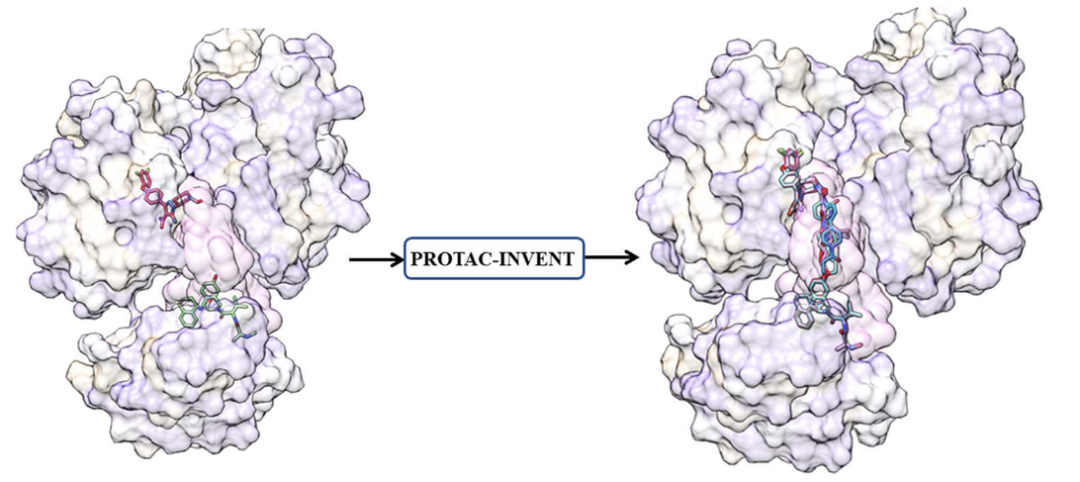
图1. 模型概览
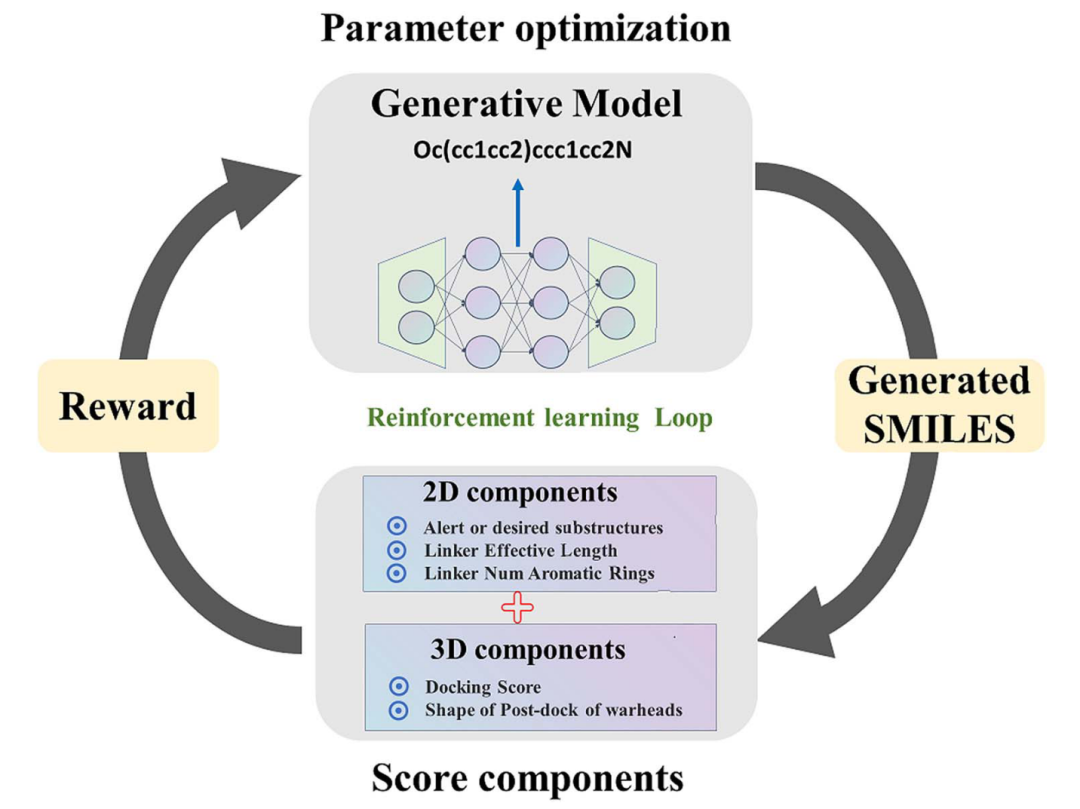
图2:PROTAC-INVENT的工作流
PROTAC的3D构象生成
在参考配体附近生成PROTAC的初始构象
首先,由预训练生成模型去生成Linker的SMILES形式,与输入的warhead和E3-ligand合并,得到完整PROTAC的SMILES。然后使用Omega[2]将其转换为初始3D构象。然后使用ROCS程序将PROATC的3D构象与参考PROTAC构象叠加在一起。ComboScore[3]用来衡量参考构象和PROTAC构象之间的相似度。由于PROTAC的分子量过大,这种叠加通常不能产生良好的对齐效果,但可以达到将生成PROTAC的linker部分带到参考分子的linker附近的目的。
拼接生成的PROTAC的linker部分与参考配体warhead和E3-ligand
叠合完成后,生成的PROTAC分子将会去除两端的结构,只保留linker部分,然后将linker与参照PROATC的两端结构拼接起来,形成重组的PROTAC构象(RPC)。经过该操作后,RPC中两端片段(warhead和E3-ligand)的坐标与参考配体完全一致,但linker与两端结构连接处的键长、二面角需要进行修正。
优化RPC的构象
为了修正RPC连接处的构象,同时使两端结构尽可能的接近参考PROTAC构象,作者使用了Schrödinger下的MacroModel模块进行了带约束的分子构象优化,得到了优化后的构象(OPC)。实现了既保持两端构象为晶体结构的构象一致,又使linker的衔接部分合理化的目的。
将OPC对接到PTS
为了进一步评估OPC在PTS内的合理程度,将OPC对接到 PTS口袋中,获得docking score。分子对接过程通常需要经过: 初始构象采样获得多个起始点,并从采样的起始点进行能量最小化。对于大体积的PROTAC分子来说,构象搜索空间将会变得很大,导致计算时间过长,而且无法保证warhead和E3-ligand在对接过程中保持与参考配体的构象一致。为了解决这个问题,作者首次采用AutoDock Vina的“local-only”模式进行对接,在这种模式下,会跳过初始构象采样阶段,而将输入构象作为构象优化的唯一起点。这种模式既最大程度的保持warhead和E3-ligand的构象不变,又能实现能量优化的目的,同时保持了对接的高效性。
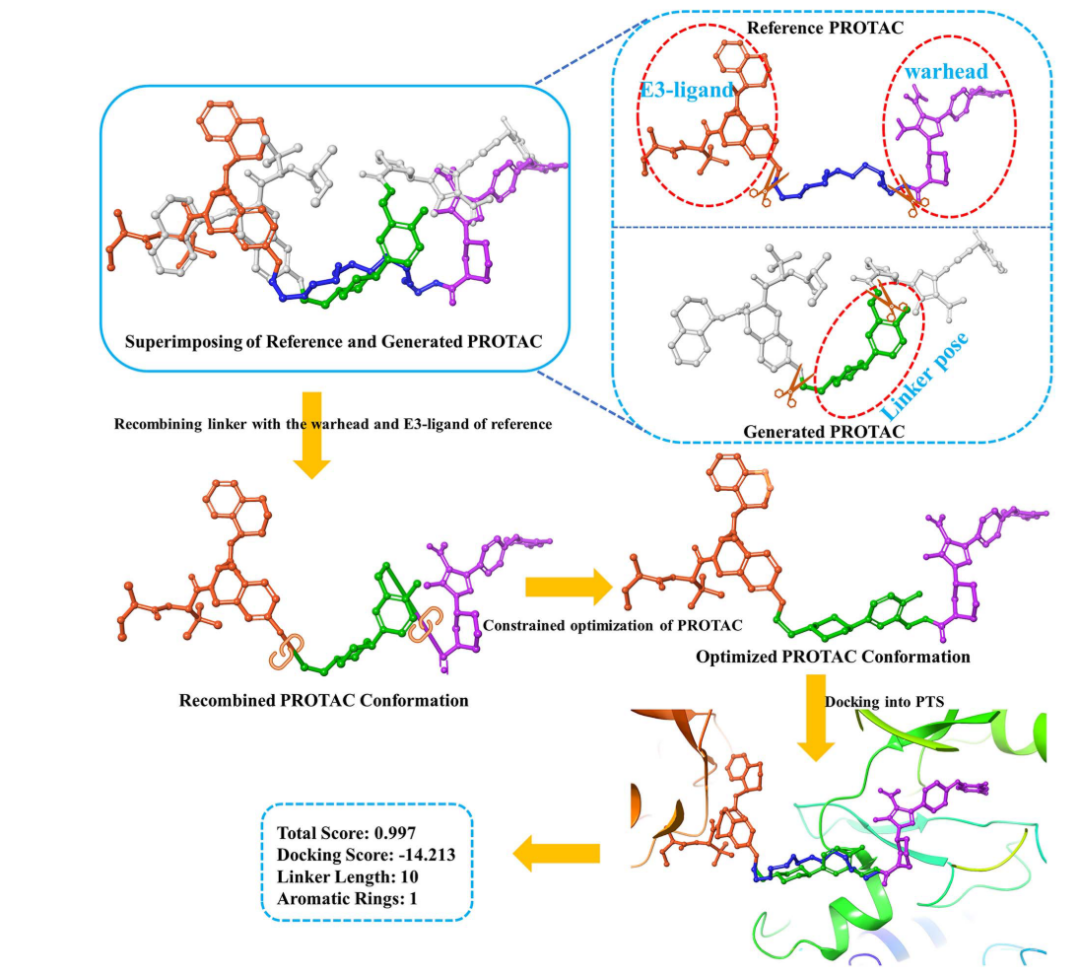
图3. PROTAC的3D构象生成过程示意图
PROTAC-INVENT的打分组件
该工作共考虑了Linker的长度、芳香环的数量、子结构的类型、对接打分以及对接后与参照分子两端结构的偏移度Ps等打分组件。这些组件通过加权和(公式1)和加权积(公式2)的形式整合在一起去评估生成的Linker的性质。
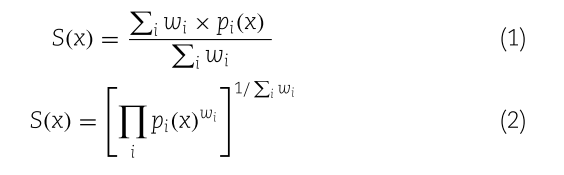
对接后与参照分子两端结构的偏移度Ps
DPC在PTS结合位点采用“local-only”模式进行对接后,PROTACs的两端结构有时会与参考PROTAC的两端发生较大的位置偏移。为了衡量这一偏移程度,作者设计了计算两个PROTAC的两端结构的Shape相似度的打分函数Ps。如图4所示的两个PROTAC分子a和b。分子a和分子b的Ps分数分别为0.903和0.687,而它们的对接分数大致相等(- 11.37 vs – 11.29)。显然,在分子对接后,分子a与参考配体的偏差比分子b小,说明a的linker更加适合该体系。
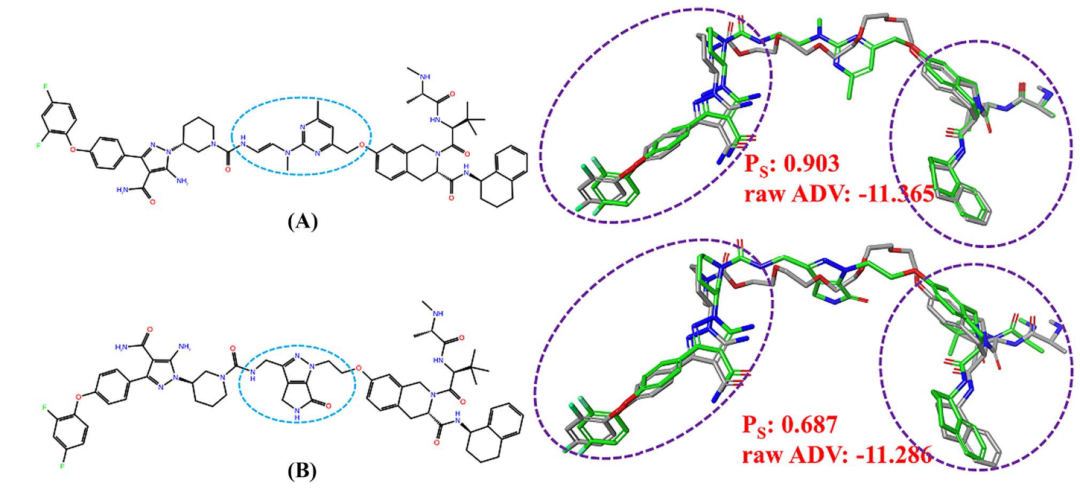
图4. PROTAC分子的Ps指标
案例分析
为了验证PROTAC-INVENT的实用性,作者选择了BTK体系(PDB code:6W8I),采用该算法去生成新颖的linker。图5展示了PROTAC-INVENT模型生成的PROTAC分子以及在PTS内构象,及其各种打分值。
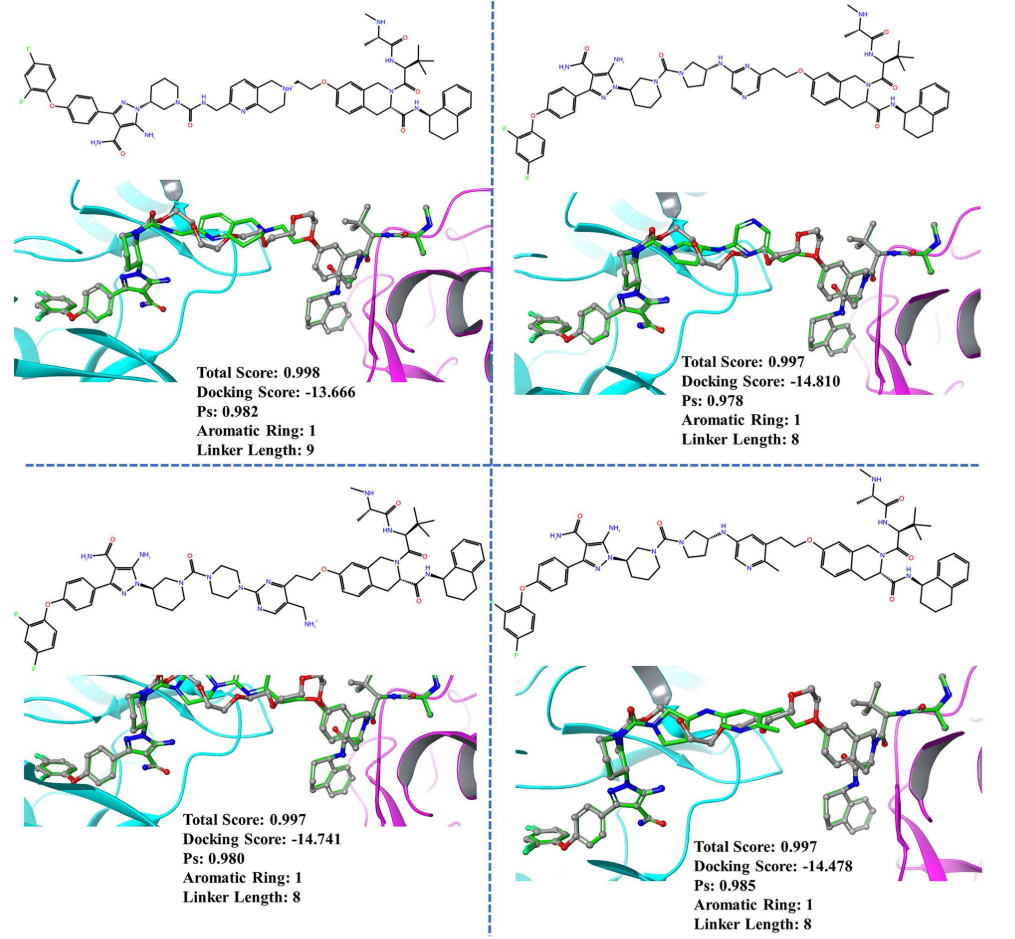
图5. PROTAC-INVENT针对BTK体系所生成的PROTAC分子
附加功能-PROTAC对接协议
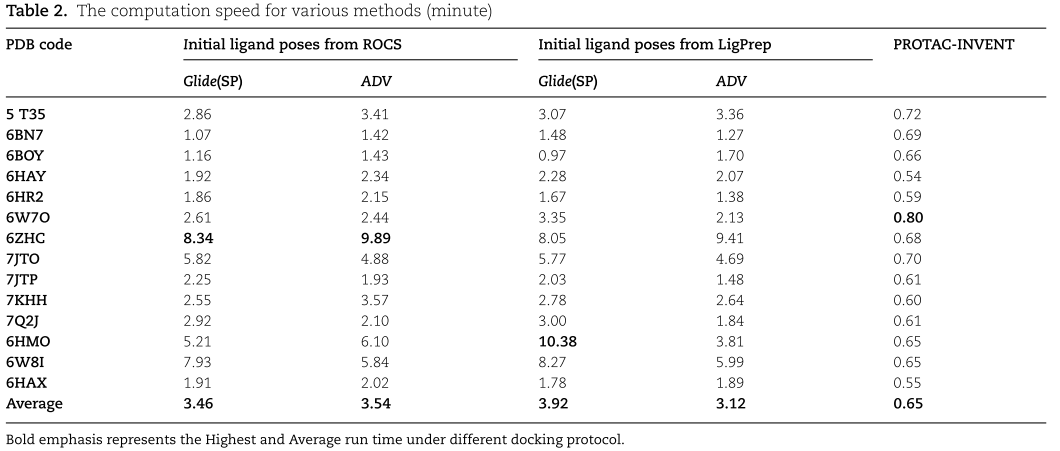
除了生成PROTAC的linker结构外,该套算法的工作流也可作为一种实用的专门针对PROTAC分子的对接算法。作者对比了PROTAC-INVENT和AutoDock Vina以及Glide算法下,对14种PTS体系下的PROTAC分子进行Redocking实验。结果发现,无论采用哪一种PROTAC的初始构象(LigPrep或Rocs),PROTAC-INVENT算法对接得到的结果都是速度最快的(如Table 2),且对接构象更加接近晶体结构构象。
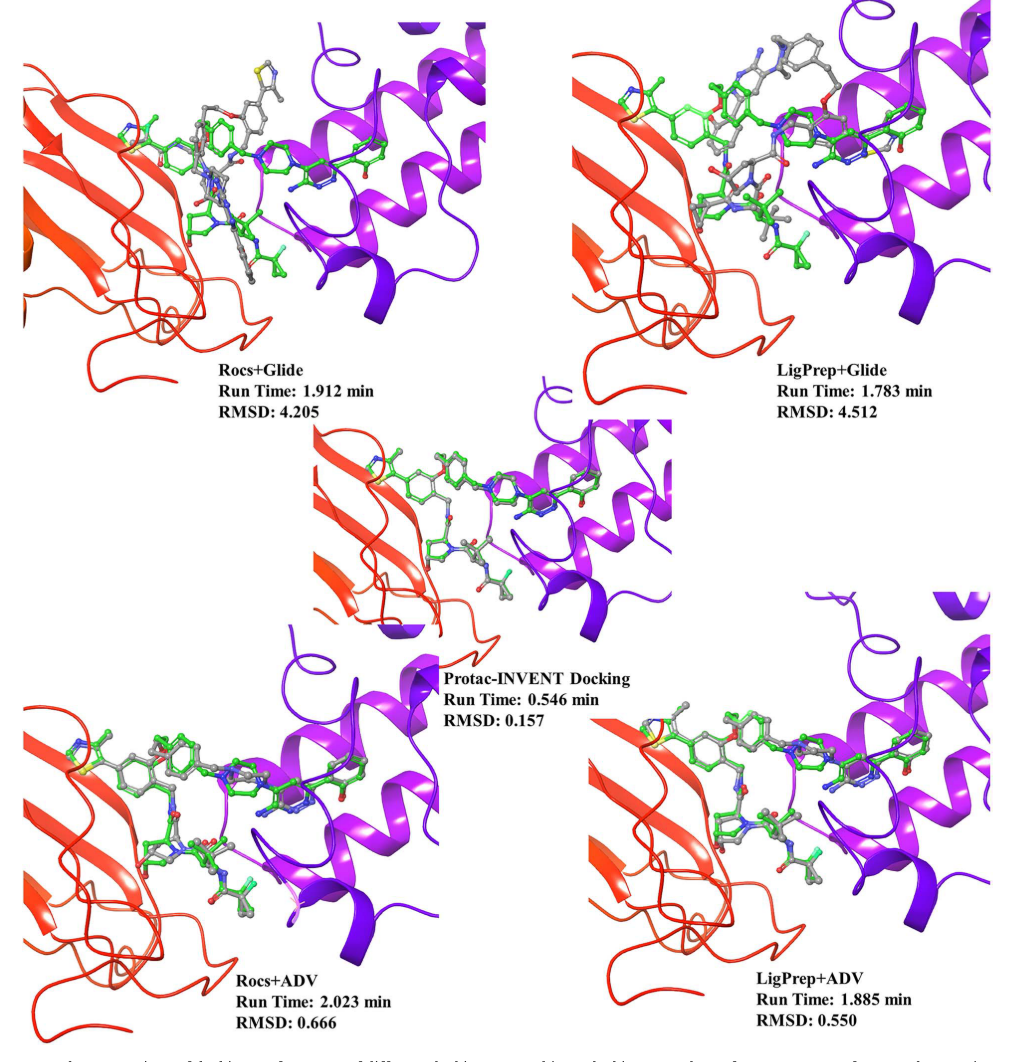
图6 BAF体系下,不同的对接方法对PROTAC的对接效果
结论
为了合理设计PROTAC分子,作者提出了一种新颖的3D的linker生成模型PROTAC-INVENT。该算法首次将linker在PTS内的影响考虑进来,实现了在保持原有warhead和E3-ligand构象不变的前体下,原位长出更加合理的linker,并形成了PROTAC的三维结合构象。同时将该算法应用到已知PROTAC晶体结构上进行Redocking验证,与Glide和Vina进行了性能比较,结果表明该模型在这些方法中RMSD和计算速度都达到了最优。
参考资料
-
Li B, Ran T, Chen H. 3D based generative PROTAC linker design with reinforcement learning. Briefings in Bioinformatics 2023; 24:1–13
-
Hawkins PCD, Skillman AG, Warren GL, et al. Conformer generation with OMEGA: Algorithm and validation using high quality structures from the protein databank and cambridge structural database. Journal of Chemical Information and Modeling 2010; 50:572–584
-
Hawkins PCD, Skillman AG, Nicholls A. Comparison of Shape-Matching and Docking as Virtual Screening Tools. Journal of Medicinal Chemistry 2007; 50:74–82



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


