——简介——
天然界存在很多具有良好机械牵拉应力的蛋白质,如弹性蛋白、丝蛋白、角蛋白和胶原蛋白等。为了适应下游的工程需要,需要对这些天然蛋白质进行人工设计,而此前的蛋白设计工作尚未涉及这一目标。2023年10月,来自MIT LAMM研究所Markus J. Buehler课题组的Bo Ni等人开发了在蛋白质序列预训练模型特征空间上进行扩散的蛋白质序列生成模型。该工作目前在预印本平台arXiv上发布[2]。作者使用较高通量的全原子拉伸动力学模拟数据作为训练集,发现生成的蛋白序列能很好地满足需要的拉伸应力特征,并和天然序列差别较大。
——方法——
作者先在PDB中选取了7026个单链结构,这些结构都不存在内部的破损,同时长度都小于128个残基。作者使用NAMD平台对每个结构进行拉伸动力学模拟,采用CGenFF力场对蛋白进行参数化,并搭配隐式水模型以加速模拟。作者直接固定N端,并沿着N端-C端向量方向施加弹性拉伸力,弹性常数为k=0.5 kcal / (mol Å2),拉伸速度为v = 0.1 Å / ps。拉伸一直持续到蛋白长度Lac达到Lcon。其中,Lcon = N × 3.6 Å,N为蛋白残基数。基于模拟所得的轨迹,可以得到随蛋白长度变化的末端应力Fp(Lac),通过对长度进行积分,可以得到蛋白的解折叠能量T,该能量某种程度上反映了蛋白的硬度,如下所示:
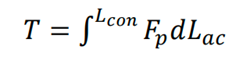
同时可以得到最大末端应力,如下所示:
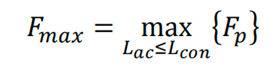
之后,对得到的末端应力Fp(Lac)分布进行采样,每个残基单位距离采样一次,这样得到一个维度最大为128的向量,此向量如下所示:
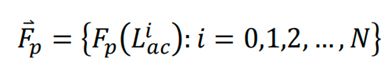
该向量记录了该蛋白的机械拉伸应力特征,并方便输入到深度神经网络中进行表示。 整体的数据集构建如下图1所示:
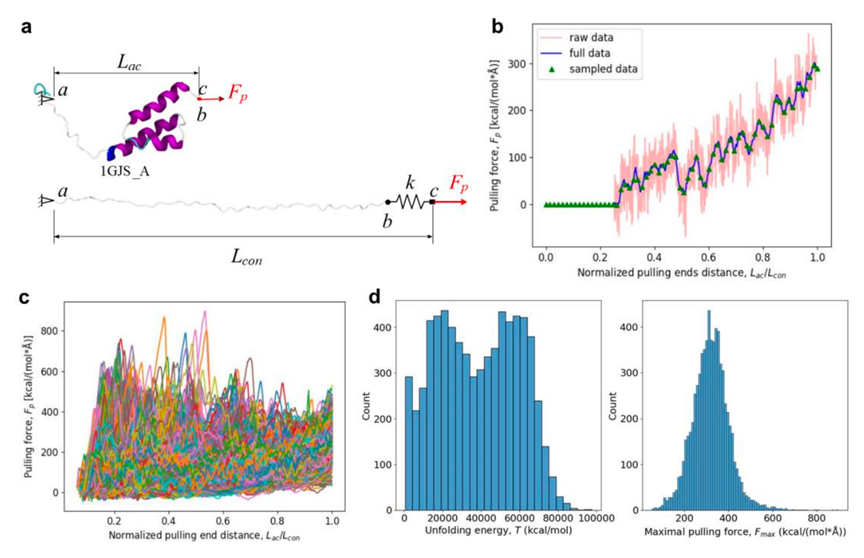
图1. 基于高通量分子动力学模拟的训练数据集
其中1.a表示蛋白在动力学模拟中被拉伸的过程。1.b表示某一蛋白模拟所得的应力-长度曲线,绿色三角代表后续输入到网络中的特征条件向量。1.c代表所有蛋白的应力分布,可见不同蛋白的应力特征多样性很高。1.d发现所有蛋白的解折叠能量呈现二元分布,而所有蛋白的最大应力则呈现正态分布。图2表示了具体的网络结构:
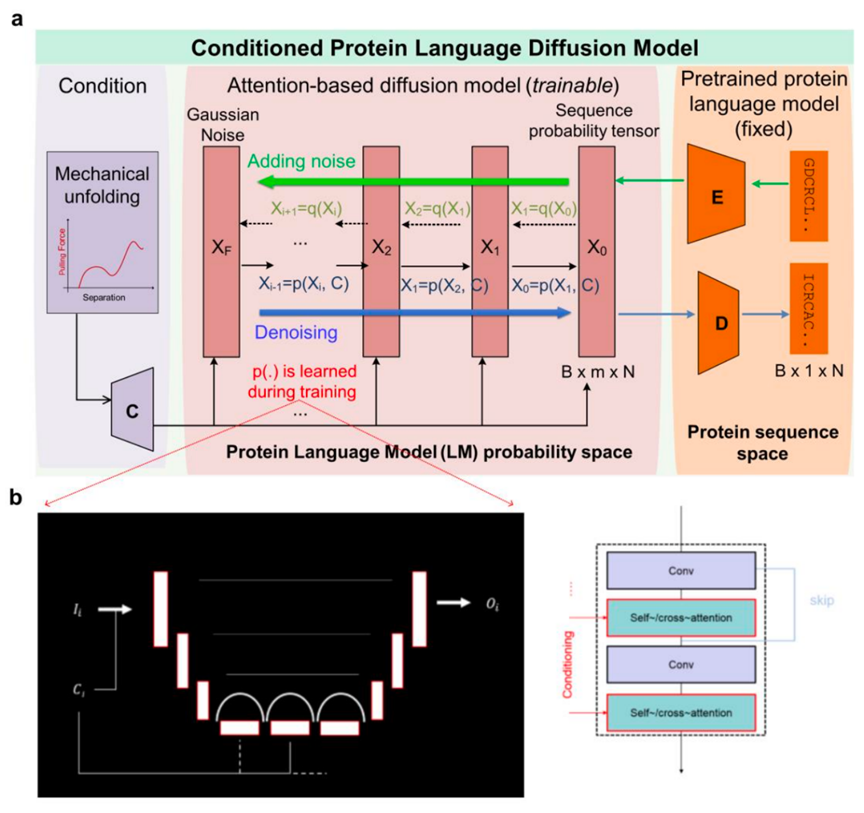
图2. 生成模型的网络架构
其中,2.a表示扩散模型的整体架构:从分子动力学中得到的机械应力向量作为条件输入,整个扩散和去噪发生在蛋白预训练的序列特征上。作者在这里实际用的是ESM-2预训练模型的150M参数量版本。而预测梯度的网络本身是1D U-Net结构,但是在每一层引入了注意力机制(图2.b)。每次扩散采样之后,将得到的序列特征再回归成具体的蛋白序列即可完成生成。作者的训练集-测试集划分为8.5:1.5。整体的生成-验证流程如图3:
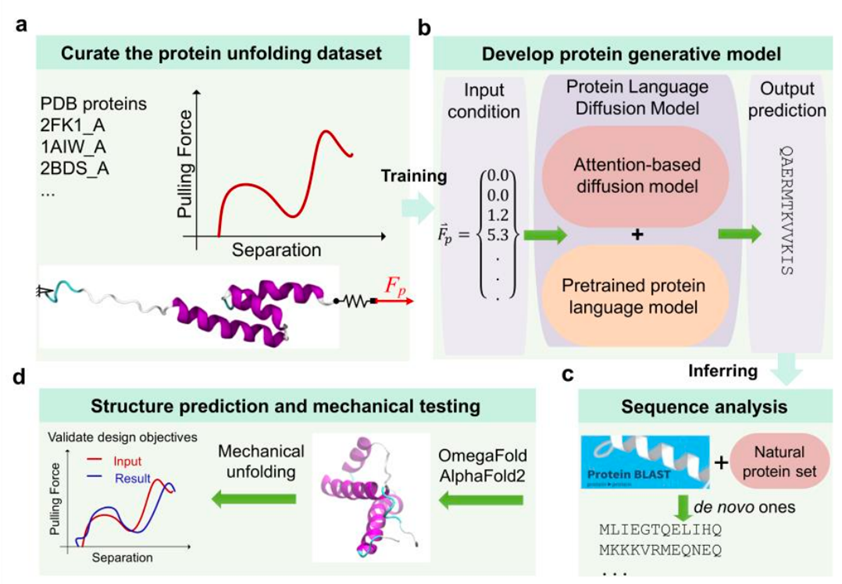
图3. ForceGen的整体流程
在完成序列生成后,作者使用OmegaFold对蛋白质进行折叠,之后进行和训练数据集同样条件的拉伸动力学模拟,之后将输入的应力分布和模拟所得的应力分布进行比较,用以评价。
——结果——
图4展示了一些测试集蛋白的生成结果:
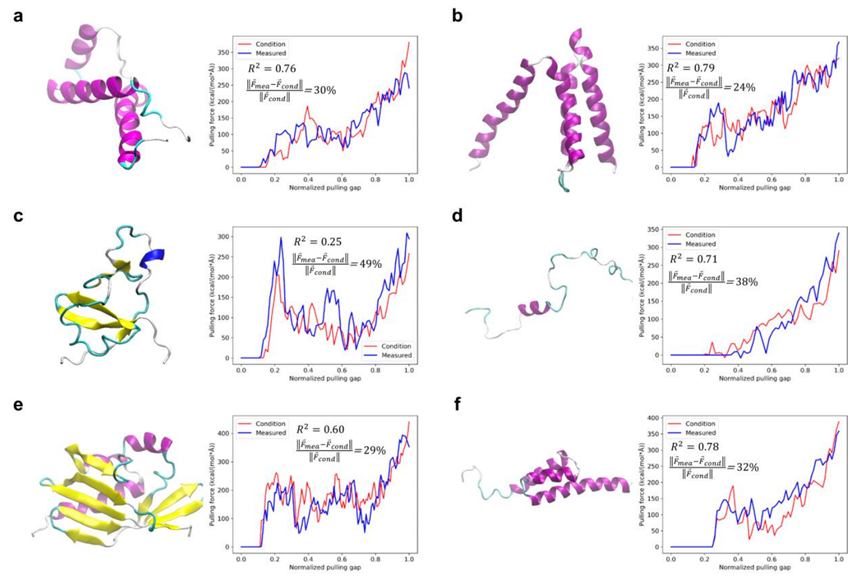
图4. 6种测试集蛋白的输入应力特征(红色)和模拟应力特征(蓝色)
作者在此主要使用R2和L2两种评价标准,其中R2越接近1越好,L2越接近0越好。从图4可见,作者展示的蛋白设计的序列都具有较好的动力学特征复原效果。 此外,作者在所有测试集上进行了总体的评价,如图5所示:
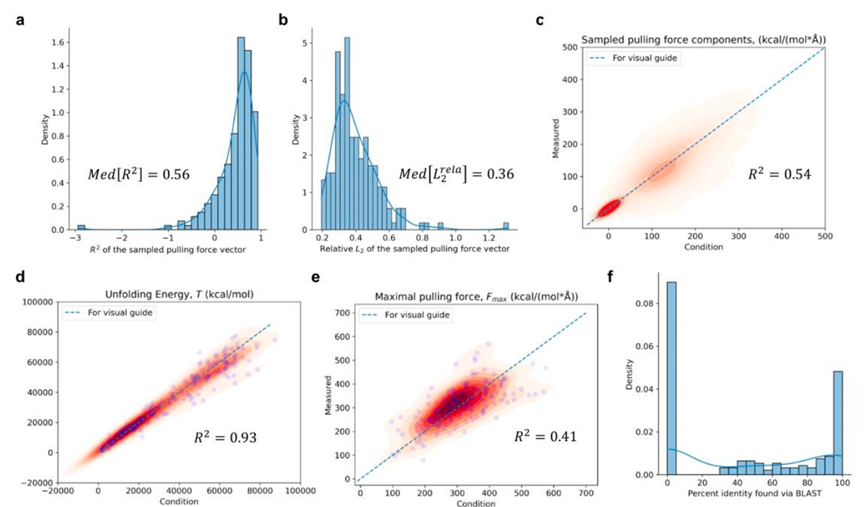
图5. 所有测试集的生成结果评价
其中,5.a和5.b分别展示了所有测试集蛋白的输入应力向量与生成序列模拟所得的应力向量的R2值与L2值。5.c、5.d以及5.e分别表示输入的应力向量、解折叠能量,以及最大应力的相关关系。5.f表示设计的序列和原始序列之间的identity。这些结果表明,在网络在一定程度上满足输入应力特征的要求时,还和原始序列有足够大的差别。随后,作者尝试对一些人造的应力特征进行了序列设计,如图6所示:
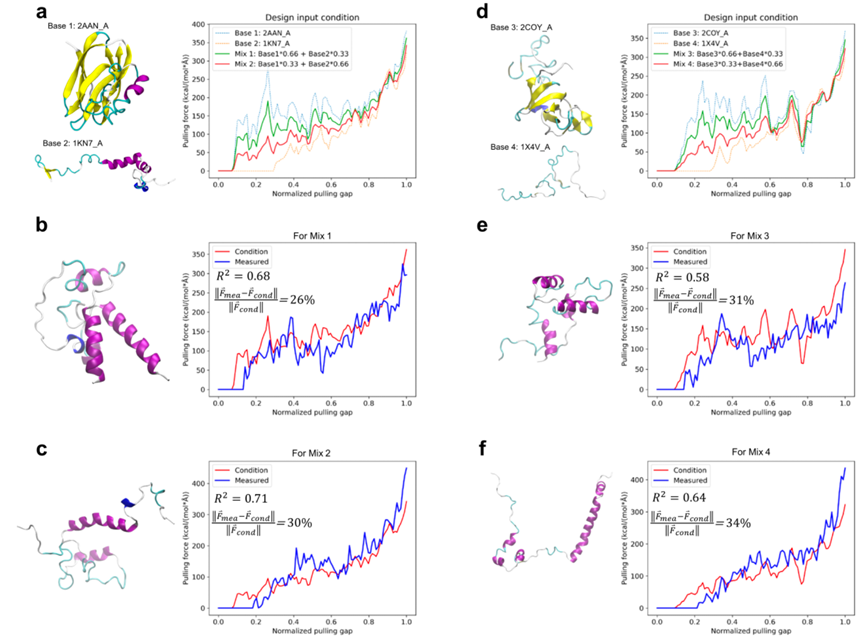
图6. 对两种天然蛋白的应力特征进行融合
其中,6.a和6.d分别是两组不同的天然蛋白,以及它们按不同比例融合出的中间态应力分布曲线。6.b和6.c是针对6.a中条件的设计结果;6.e和6.f是针对6.d中条件的设计结果。作者认为这一任务的设计同样较为成功,说明模型有设计全新应力特征蛋白的能力。
——小结——
总的来说,ForceGen针对一种较为冷门的蛋白设计任务提供了一个良好的深度生成模型。同时,应用MD模拟的方法对结果进行验证,初步证明方法的可行性。
此文的借鉴意义主要如下:1. 本文使用高通量的分子动力学模拟构建数据集,这在AI4S领域已经不是第一次出现,但在AI辅助蛋白设计这一子领域应用还不多,并且都还不太成熟,而本文提供了一种可行的应用思路。2. 网络架构部分将当前蛋白质深度学习领域两种最强的方法(即蛋白质大语言模型和扩散模型)进行了结合。3. 针对蛋白应力的从头设计可能会对生物高分子材料的工程制造提供新的活力。笔者认为,本文可能的问题有:1. 目前所有的验证都是基于MD模拟,没有更强的证据(比如实验)对设计的序列进行验证。2. 在结构预测部分使用了OmegaFold,虽然作者在绘图时绘制了AlphaFold的示意图,但实际上没有使用。OmegaFold的精度是否足够也需要考虑。3. 本文预测的很多蛋白可见有较长的IDR,这部分的动力学特征或许会较为复杂,单次采样的随机性需要被讨论。4. 本文对MD轨迹的利用率实际上还有所不足,既然已经做了AA-level的模拟,其实其中随轨迹变化的残基互作特征(如H-bond和DCCM等)或许都是能够被嵌入的特征。
参考文献:
[1] Wu, Kevin E. et al. “Protein structure generation via folding diffusion.” ArXiv abs/2209.15611 (2022): n. pag.[2] Jing, Bowen et al. “EigenFold: Generative Protein Structure Prediction with Diffusion Models.” ArXiv (2023): n. pag.[3] https://www.microsoft.com/en-us/research/blog/distributional-graphormer-toward-equilibrium-distribution-prediction-for-molecular-systems/
点击左下角的”阅读原文“即可查看原文章。作者:穆俊羲审稿:王宇哲编辑:王宇哲
GoDesignID:Molecular_Design_Lab( 扫描下方二维码可以订阅哦!)



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


