编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自 Hao Liu和 Liang Hong团队的一篇论文。基于深度学习的分子生成模型在新药设计领域引起了广泛关注。然而,大多数现有模型专注于基于配体或基于结构的策略之一,因此未能有效利用从配体和结合靶点的结构两方面获得的综合知识。在文章中,作者介绍了LS-MolGen,一种新颖的配体和结构整合型分子生成模型。该模型协同地结合了表示学习、迁移学习和强化学习。来自迁移学习的有针对性知识吸收,再加上强化学习中的高级探索策略,使LS-MolGen能够高效生成新颖且高亲和力的分子。

对于特定靶标的“de novo”(即新的、从无到有的)药物设计的主要目标是识别全新的活性分子,这些分子能够同时满足某些理想性质。近年来,各种基于人工智能(AI)的分子生成模型已经提出,以满足化学空间探索的潜力需求。这些生成方法主要分为两类:基于配体和基于结构。基于配体的方法主要关注已知配体及其性质,以生成新的分子。例如,VAE(变分自编码器)利用变分自编码器将分子的离散表示转换为多维连续表示,从而使在已知配体的化学空间内生成新的分子成为可能。相反,基于结构的方法考虑生物靶标的三维结构,例如蛋白质,并旨在生成能够与该靶标相互作用的分子。某些方法旨在弥合基于配体和基于结构方法之间的差距,整合了两者的知识。这些多样化的方法利用人工智能和机器学习来增强“de novo”药物设计,最终目标是发现新的分子,这些分子具有用于药物开发的所需性质。
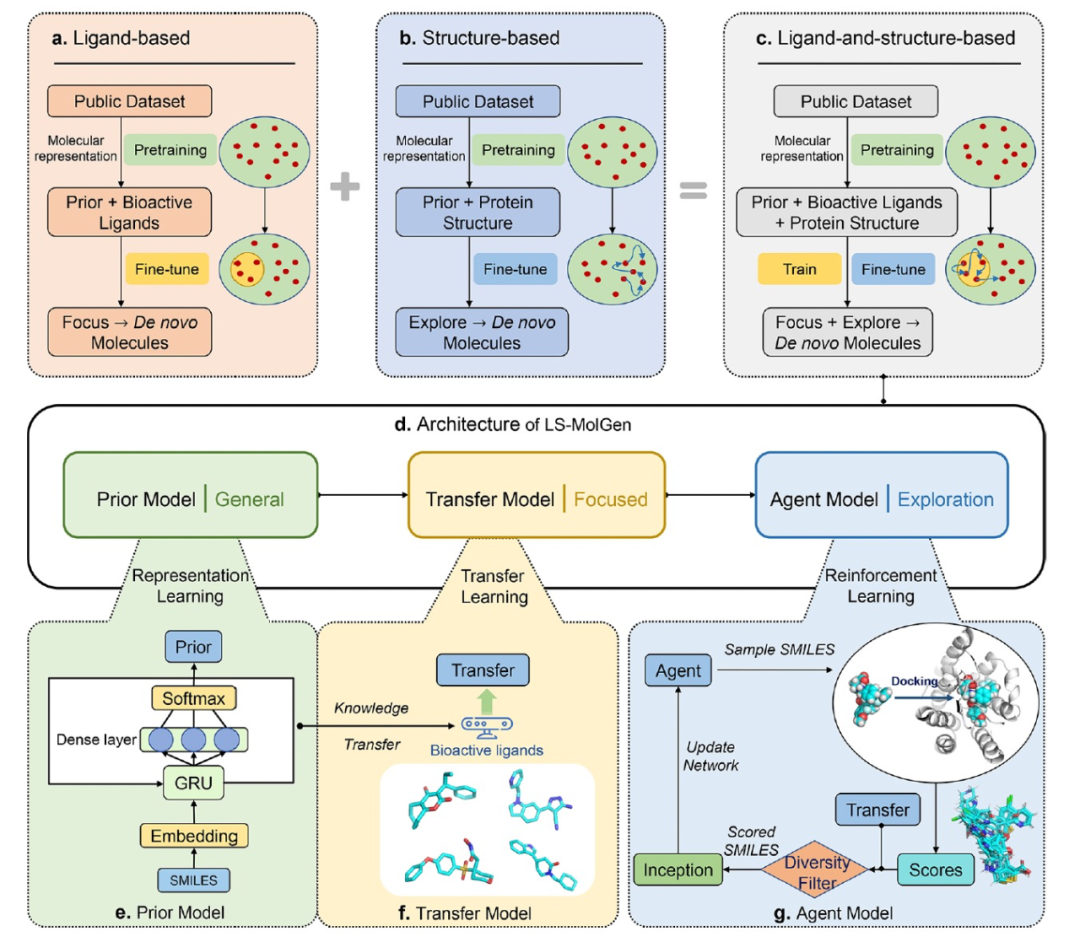
图 1
通常情况下,如图1a所示的基于配体的方法通常利用已知的生物活性化合物作为参考点,这些化合物已在细胞、动物甚至临床环境中证明了它们的有效性。如图1b所示的基于结构的方法侧重于生成与靶标蛋白质的结构的物理相互作用的分子。图1c所示的基于配体和结构的混合方法融合了先前两种方法的优点。通过利用已知化合物和靶标蛋白质结构的互补信息,这种方法促进了新化合物的生成,这些化合物不仅在化学上覆盖了已知的生物活性化合物,还有可能具有更高的结合亲和力。作者提出了一种基于配体和结构的双重驱动深度强化学习方法(LS-MolGen)用于特定靶标的分子生成。通过利用已知生物活性化合物和特定靶标的结构的互补知识,并在强化学习的起始阶段集成对接得分和多样性过滤器。测试结果显示,生成的分子具有更好的新颖性和亲和力。
数据来源
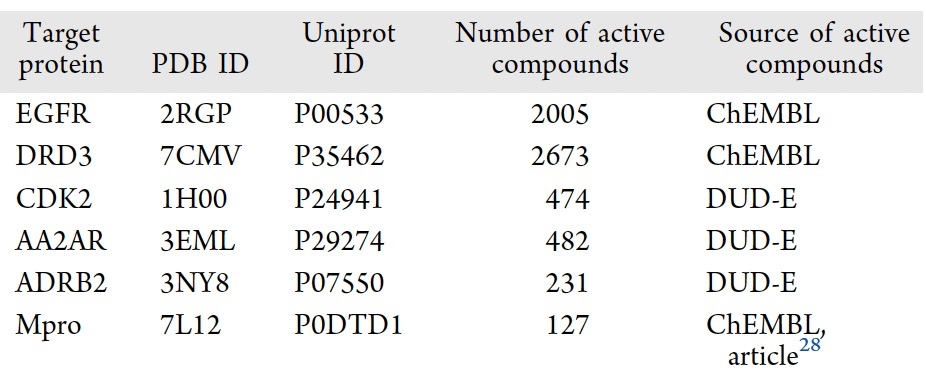
表 1
作者从ChEMBL数据库下载了以规范的SMILES格式表示的超过1.9百万个小分子。对于所有的SMILES字符串,需要进行数据预处理,去除重复和无效的SMILES,构建了一个包含150万个分子结构的基准数据集。在这项工作中,作者准备了六个不同的靶标蛋白,包括两种激酶(人表皮生长因子受体(EGFR)和细胞周期依赖性激酶2(CDK2)),三种G蛋白偶联受体(多巴胺受体D3(DRD3),腺苷A2a受体(AA2AR)和β-2肾上腺素受体(ADRB2)),以及SARS-CoV-2主蛋白酶(Mpro)(表1)。这些已知的活性化合物将用于在迁移学习过程中对生成模型进行微调。
模型设计
LS-MolGen模型的整体架构概述如图1d所示,包括三个关键子模型。第一个模型是用于通用知识的预训练的初始阶段模型,称为“先验模型”(图1e),基于递归神经网络(RNN)。第二个模型是一个迁移模型(图1f),基于与先验模型相同的架构,用于通过共享先验网络和层的重新加权,将通用知识进行迁移学习到关注的知识领域。最后一个是代理模型(图1g),通过分子对接引导的强化学习进行微调,用于基于结构的有理化学空间探索。在这里,用于估计靶标和配体之间相互作用强度的对接分数是从LeDock获得的。因此,LS-MolGen是一种双驱动模型,同时充分利用已知活性配体和蛋白质结构的知识,以生成具有高结合亲和力和新颖性的分子。
基准数据集中,以SMILES格式表示的每个分子都被分割成一系列token,然后收集所有token以构建SMILES词汇表,最终的词汇表包含101个token。通过从基准数据集中学到的属性语法,可以生成新的有效的SMILES序列,这些序列是从数值化的基准数据集中学到的。先验模型由标准的RNN模型组成,其架构如图1e所示。它包含六层:一个输入层,一个嵌入层,三个递归层和一个输出层。模型采用了最大似然估计作为损失函数来训练RNN。
为了引导生成模型朝着与化学空间中已知配体相似或具有高结合亲和力的区域发展,作者将其置于一个迁移学习场景中,旨在学习生物活性配体的分布。这个模型需要一个预先训练好的先前模型,具有生成能力,并有可能从相当广泛的化学空间中抽样化合物。然后,该先前模型经过迁移学习,使用较小的已知生物活性配体集合。迁移学习期间的损失函数与预训练期间相同。
作者将分子对接得分引入到强化学习的评分函数中,以引导代理模型在化学空间中探索新颖的化合物。强化学习中的代理模型是一个探索模型,其结构和词汇与先前模型相同。基本上,在强化学习的开始阶段,代理是由预训练的先前网络初始化的。作者采用了REINVENT2.0中的强化学习算法,为分子优化构建了一个定制的评分函数。在代理模型采样一批SMILES后,作者提出的模型奖励来自多个组成部分:对接得分、骨架多样性过滤器以及来自原迁移模型的概率。
实验部分
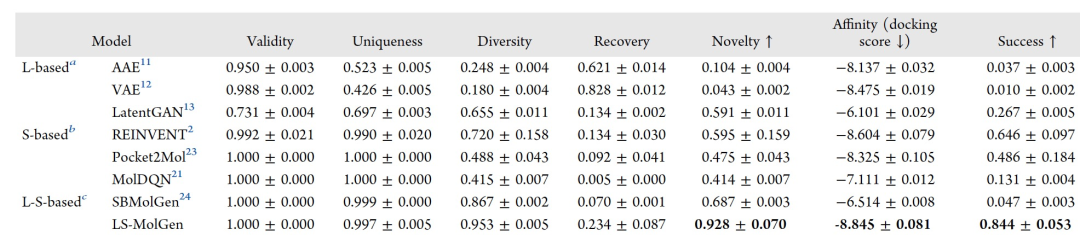
表 2
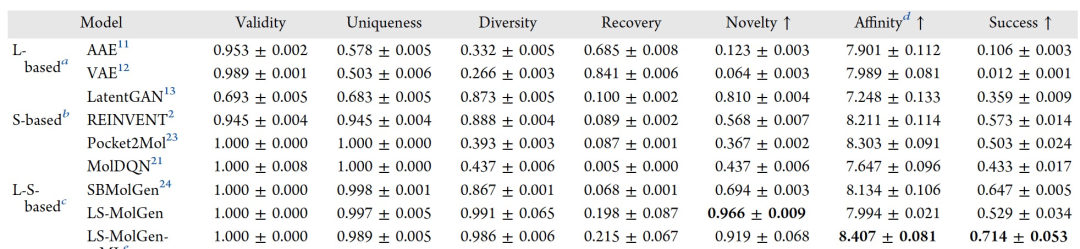
表 3
作者首先在EGFR和DRD3数据集上上分析模型,深入研究了LS-MolGen模型的综合评估结果。该模型是使用人表皮生长因子受体(EGFR)和多巴胺受体D3(DRD3)作为靶蛋白进行训练的。作者利用LS-MolGen来设计EGFR抑制剂和DRD3拮抗剂。用于基于配体的生成模型的模型包括AAE、VAE和LatentGAN。表2和表3分别展示了LS-MolGen和各种基线模型在生成EGFR和DRD3靶向分子方面的比较性能。在生成EGFR靶向分子时(表2),LS-MolGen在新颖性、亲和力和成功性方面优于所有基线模型。此外,LS-MolGen在有效性、独特性和多样性方面表现出色。尽管与基于配体的方法相比,恢复分数较低,但高新颖性分数表明LS-MolGen侧重于创建新颖的分子,而不是复制训练集中的分子。
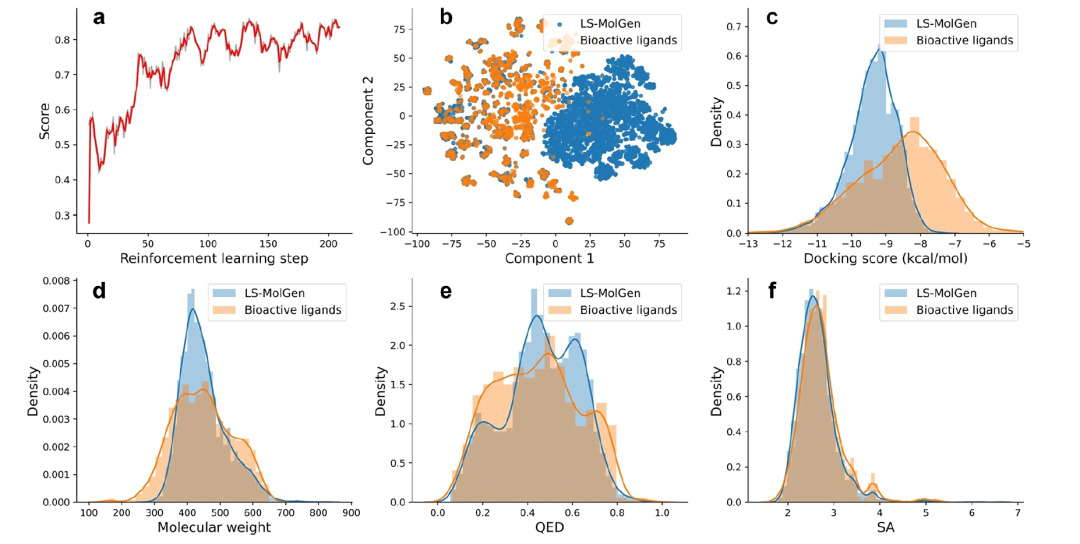
图 2
然而,对于亲和力,分子对接分数并不总是可靠的指标。对于DRD3拮抗剂的实验,已知活性化合物的分子对接分数与其实验测定的活性不一致。为了克服这一局限性,作者在强化学习过程中引入了一个基于机器学习的亲和力预测器,与LeDock结合使用。该预测器是一个回归模型,训练数据自ChEMBL的已知DRD3化合物为训练集,标签为pIC50(-log(IC50))。这个修改后的模型被称为LS-MolGen-ML,表现出更好的性能。与仅使用分子对接分数的LS-MolGen相比,LS-MolGen-ML,在其中结合了预测的pIC50,在生成具有高亲和力和成功率的分子方面胜过了其他方法(表3)。
图2a表明了强化学习过程中代理模型的探索工作。代理模型的分数来自分子对接分数、骨架多样性过滤器和基于原迁移模型的分子存在可能性,不断上升的分数表明了该模型有效地探索了新颖和活跃分子的化学空间的能力。为了更好地了解生成分子的化学空间,作者通过计算Morgan指纹,将其用作t-分布随机邻居嵌入(t-SNE)可视化,评估了生成分子的化学空间覆盖度。正如图2b所示的t-SNE图中,生成的分子不仅覆盖了已知EGFR抑制剂的化学空间,还将其扩展到了新的化学空间。图2c显示,生成的分子与已知的生物活性配体相比具有更高的亲和力。此外,还比较了分子性质的分布(分子量、QED和SA得分)。如图2d-f所示,生成分子的性质分布接近于已知配体的性质分布。考虑到上述定量评估,作者开发的模型LS-MolGen通过生成新颖且活跃的分子,在药物设计潜力方面表现出令人满意的结果,胜过其他方法。
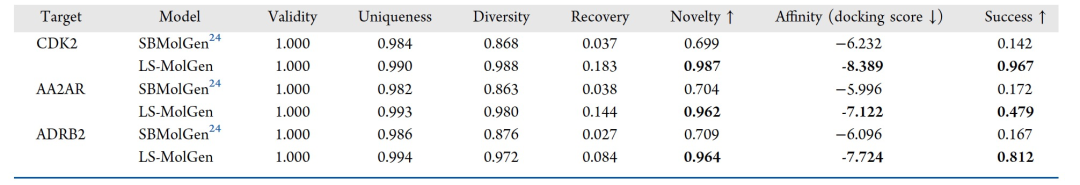
表 4
作者在表4对LS-MolGen模型与SBMolGen模型进行了全面评估,比较涉及了三个蛋白靶点 – CDK2、AA2AR和ADRB2。在生成有效和独特分子方面,两个模型都表现出令人印象深刻的性能。然而,在考察多样性、恢复性、新颖性、亲和力和成功率等指标时,LS-MolGen始终超越SBMolGen模型。对于CDK2蛋白靶点,LS-MolGen生成具有明显更高新颖性和亲和力的分子。对于AA2AR和ADRB2靶点,这一趋势也是一致的,表明LS-MolGen更有能力生成具有高结合亲和力的新分子。
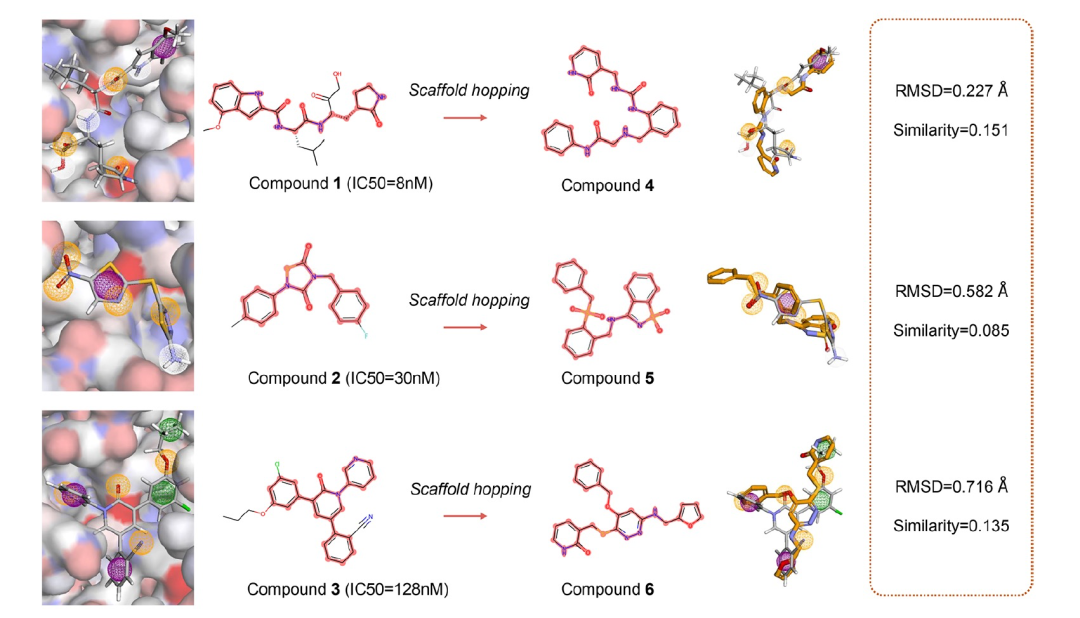
图 3
随后作者使用LS-MolGen,生成潜在的SARS-CoV-2主蛋白酶(Mpro)抑制剂。Mpro是COVID-19治疗发现的关键靶点。该过程涉及利用来自ChEMBL和最近发表的文献的已知Mpro抑制剂的集合。从RCSB PDB(ID:7L12)获得的与抑制剂复合物的Mpro共晶结构被用作结构参考。LS-MolGen生成了5,000个Mpro的潜在抑制剂。作者选择了三种SARS-CoV-2 Mpro的强效抑制剂进行比较,化合物1(PF-0835231)(IC50 = 8 nM),化合物2(IC50 = 30 nM),和化合物3(IC50 = 128 nM)。化合物4-6是从生成的分子中选择的,因为它们与这些抑制剂具有最匹配的药效特征(图3)。尽管抑制剂与相应生成的化合物之间的结构相似性较低,但分子骨架是全新的,表明模型成功进行了骨架跳跃生成。这一结果表明,LS-MolGen可生成与已知活性抑制剂在结构上完全不同的分子。
结论
这项研究中,作者提出了LS-MolGen,这是一种先进的分子生成模型,用于全新的药物设计,它整合了关于活性化合物和靶标蛋白结构的知识。该模型由表示学习、迁移学习和强化学习子模块组成,提供了一种多功能方法来理解SMILES字符串规则,专注于活性化合物的化学空间,并探索潜在的化学空间。
参考资料
Li, S., Hu, C., Ke, S., Yang, C., Chen, J., Xiong, Y., … & Hong, L. (2023). LS-MolGen: Ligand-and-Structure Dual-Driven Deep Reinforcement Learning for Target-Specific Molecular Generation Improves Binding Affinity and Novelty. Journal of Chemical Information and Modeling.




 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


