编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Alexander Gusev团队的一篇论文。原发部位未知癌症(Cancer of unknown primary,CUP)是一种无法追溯到其原发部位的癌症,占所有癌症的3-5%。CUP缺乏已建立的靶向治疗方法,导致普遍预后不佳。作者开发了OncoNPC,这是一个基于机器学习的分类器,使用来自三个机构的22种癌症类型中的36,445个肿瘤的定向次世代测序(NGS)数据进行训练。肿瘤NGS基础的原发癌类型分类器(OncoNPC)在保留的肿瘤样本上取得了加权F1分数为0.942的高置信度预测(≥ 0.9)。

当标准化的诊断流程,包括影像学和病理学评估,未能确定转移性癌症的原发部位时,将其诊断为原发部位未知的癌症(Cancer of Unknown Primary,CUP)。CUP在全球范围内占所有癌症的约3-5%,以其侵袭性进展和预后不良(生存期为6-16个月)为特点。原发部位的隐藏性质限制了治疗选择,因为临床对某些治疗的反应因患者的肿瘤类型而异。新兴的癌症治疗针对可操作的分子改变通常是针对特定癌症类型开发的,因此对于CUP患者意义不怎么大。因此,准确地确定CUP肿瘤的潜在原发部位,并证明针对特定部位的治疗在临床上的益处,可能会为CUP患者开启许多现有的治疗选择。
病理学评估在根据免疫组织化学(IHC)结果以及肿瘤形态学和临床发现确定恶性肿瘤的原发癌症类型方面发挥着关键作用。然而,高度转移性或分化差的肿瘤的病理诊断可能具有挑战性。对于已知的癌症类型,先前的研究显示基于IHC的诊断流程可以正确识别77-86%的原发肿瘤,而对于转移性肿瘤则降低到60-71%。对于CUP患者,表明单一原发诊断的IHC结果仅占肿瘤的25%。分子肿瘤分析已被提出作为原发部位分类的一种替代方法,可能适用于CUP肿瘤,因为其具有定量性质,并且在已知癌症类型的肿瘤中具有高准确性。这些工具依赖于微阵列DNA甲基化、全基因组测序、RNA测序数据或基因表达谱。然而,尽管它们是有效的,但这些测序技术尚未整合到标准护理中,而且往往成本较高。在最近的研究中,已经证明通过针对目标区域的下一代测序(NGS)可以进行准确的原发癌症类型分类,这些测序数据现在在许多癌症中心收集。然而,它在诊断和辅助CUP患者治疗方面的临床效用尚未得到系统地调查。在这里,作者使用了来自三个机构的36,445个肿瘤样本的NGS目标面板测序数据,以训练和评估一个机器学习分类器,用于预测给定肿瘤样本的原发癌症类型。
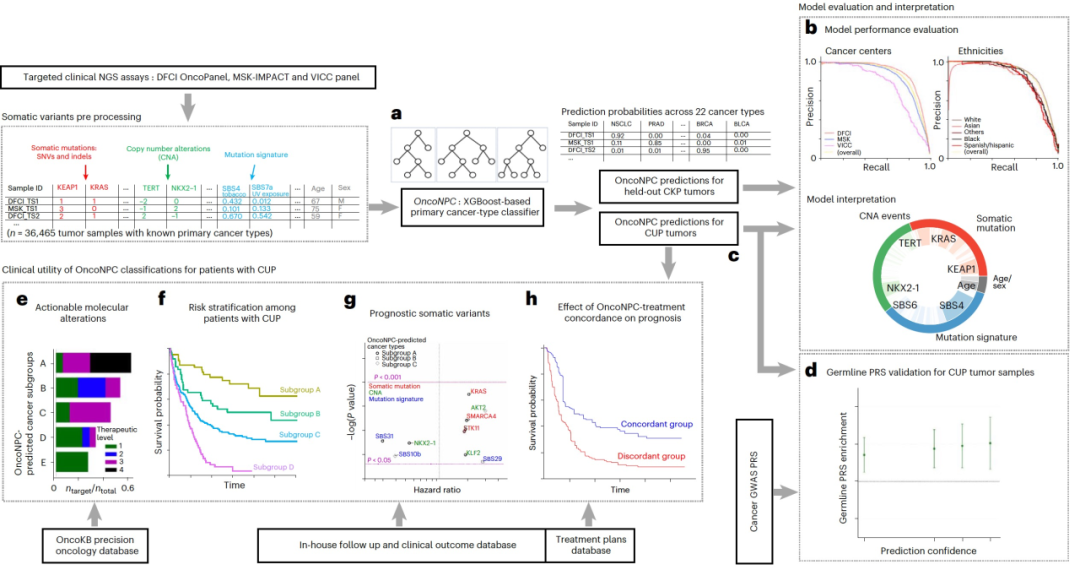
图 1
OncoNPC准确地分类了22种已知的癌症类型
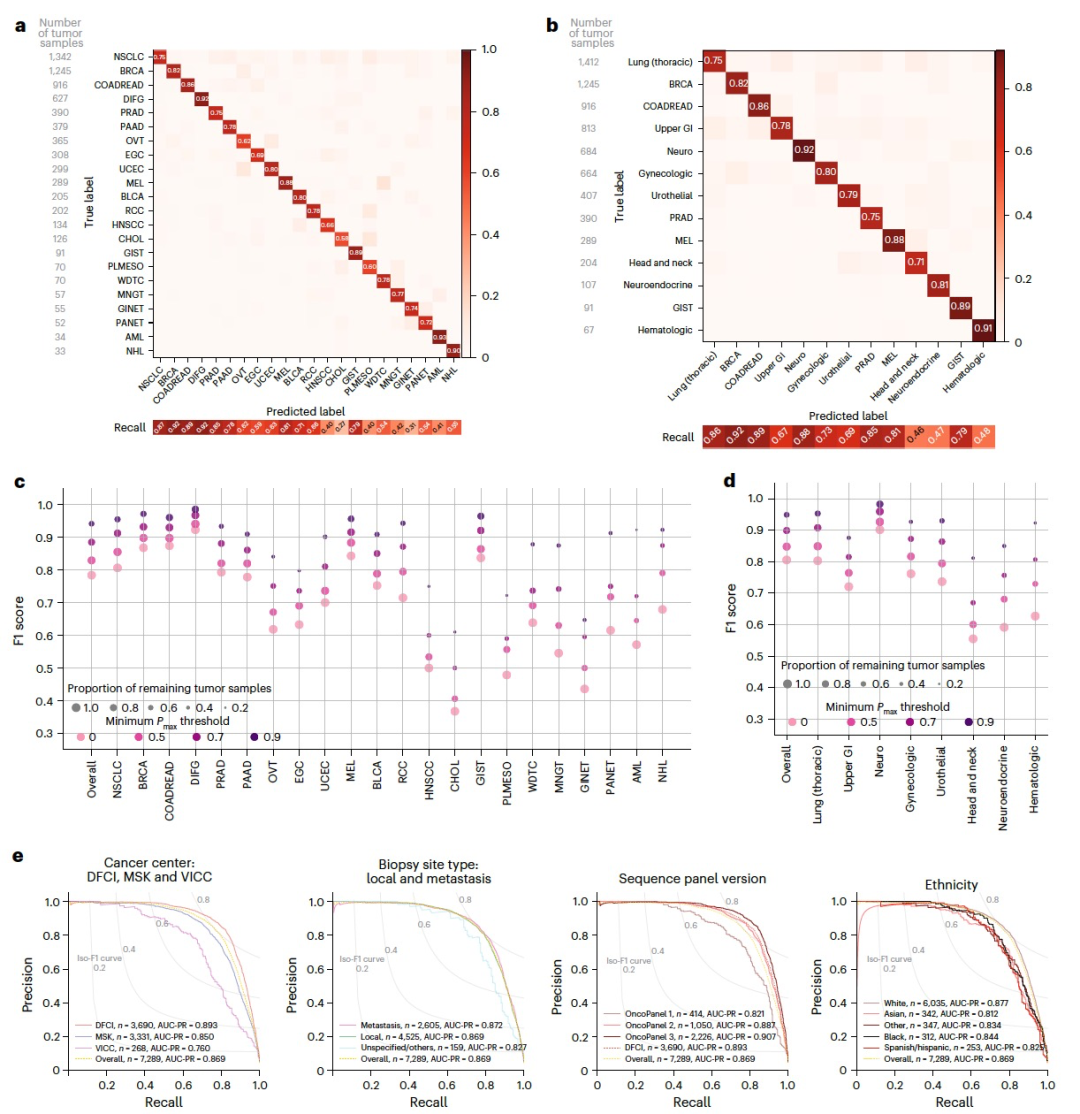
图 2
作者开发了OncoNPC,这是一个基于多中心目标面板测序数据训练的分子癌症类型分类器(图1)。OncoNPC使用体细胞变异,包括突变(单核苷酸变异(SNVs)和插入/缺失突变),突变标记、拷贝数变异(CNAs)以及测序时的患者年龄和性别等特征,使用XGBoost算法进行癌症类型预测。OncoNPC在包括来自Dana-Farber癌症研究所(DFCI)、Memorial Sloan Kettering(MSK)癌症中心和Vanderbilt-Ingram癌症中心(VICC)的22种已知癌症类型的29,176个原发肿瘤和转移肿瘤样本的处理数据上进行了训练和验证。在所有22种癌症类型中,OncoNPC在测试样本上实现了0.784的加权F1分数,其中包括7,289个肿瘤样本(加权精度和召回率分别为0.789和0.791)。在13个癌症组中(根据部位和治疗选择分组;表1),OncoNPC实现了0.806的整体加权F1分数(加权精度和召回率分别为0.810和0.809)。尽管癌症类型之间存在明显的类别不平衡,OncoNPC在癌症类型(图2a)和癌症组(图2b)之间展现出良好的平衡精度。作者在以下四个不同的预测置信水平下评估了OncoNPC的性能,这些水平基于pmax(即在22种癌症类型中的最大预测概率):0.0(涵盖所有样本),0.5,0.7和0.9。基于pmax的阈值应用结果进一步改善了性能,当pmax≥0.5时,加权F1得分为0.830,当pmax≥0.9时,加权F1得分为0.942(图2c、2d)。虽然罕见的癌症类型通常具有较低的整体性能,但增加pmax阈值可以减小常见/罕见癌症类型之间的差异。特征去除研究证明,在仅保留基因组特征的前50%的情况下,OncoNPC仍然可以获得高性能(整体加权F1得分为0.757,而在pmax阈值为0和0.9时分别为0.777和0.950)。
应用OncoNPC于CUP肿瘤样本
作者将OncoNPC应用于对971例CUP肿瘤的分类,这些肿瘤样本来自于被送往DFCI(Dana-Farber癌症研究所)并作为常规临床护理的一部分进行了测序。与DFCI的3690个已知原发癌肿瘤(CKP)的OncoNPC分类相比,CUP肿瘤的预测概率平均较低(0.764与0.881相比),但与DFCI的8025个CKP,包括未在OncoNPC中建模的肿瘤类型,的预测概率相当(0.769)。这表明CUP肿瘤可能包含其他罕见的肿瘤类型。尽管如此,41.2%的CUP肿瘤(971个中的400个)仍然可以高度自信地分类(即,pmax ≥ 0.9),并且多个分类的肿瘤类型,包括NSCLC、BRCA、PAAD和PRAD,其预测概率分布与其对应的CKP相当(图3a)。有趣的是,尽管在训练集中这些类型的肿瘤样本数量很少(n = 359,占训练集的0.99%),但被预测为GINET的CUP仍然高度自信,这表明一些罕见的肿瘤类型可能仍然可以自信地识别出来。如图3b所示,最常见的CUP癌症类型是NSCLC、PAAD、BRCA、EGC和COADREAD。NSCLC、BRCA和COADREAD也是最常见的CKP类型。这与以前的研究结果大致一致,即通过尸检揭示的CUP的潜在原发癌中,最常见的包括肺癌、大肠癌和胰腺癌。最后,将OncoNPC应用于MSK癌症中心的581个CUP肿瘤时也观察到了类似的比率。
解释OncoNPC癌症类型预测
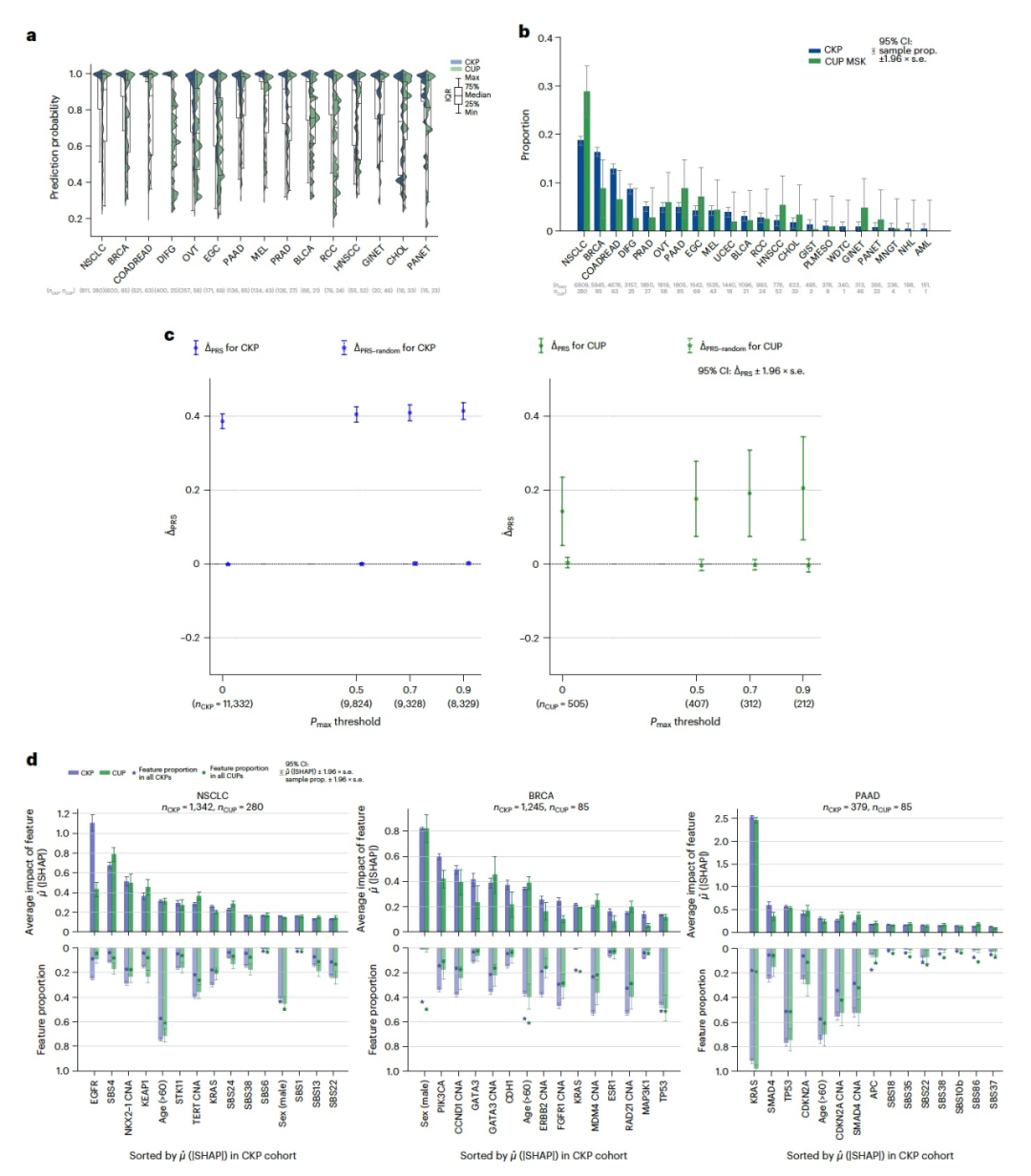
图 3
OncoNPC学习了输入体细胞变异和临床特征之间的复杂非线性关系,并提供可解释的原发癌症类型预测,其中每个输入特征对预测的影响通过SHAP值进行量化。作者调查了在CKP和CUP队列中预测每种癌症类型最有影响力的特征,以评估OncoNPC的有效性(有关CUP队列中前三个最常预测的癌症类型的信息,请参见图3d:NSCLC、BRCA和PAAD)。对于NSCLC,最重要的特征是EGFR突变和SBS4,这是与烟草吸烟相关的突变特征,分别用于预测CKP肿瘤样本和预测为NSCLC的CUP肿瘤样本,与肺癌的已知病因一致。EGFR基因的体细胞突变在NSCLC肿瘤中经常观察到,并且该基因本身是NSCLC患者的已知治疗靶点。烟草烟雾中的致癌物质已被认为是导致肺癌的原因。对于BRCA,对于CKP和CUP肿瘤样本,最重要的特征是性别,如预期的那样,其次是PIK3CA的体细胞突变和CCND1基因的变异,这些在乳腺癌中是已知的驱动基因和预后指标。对于PAAD,KRAS突变明显高于人群平均水平,并且是最重要的体细胞特征。KRAS基因的突变在胰腺癌患者中经常发生,并且已知具有预后意义。OncoNPC提供了直观的可视化,以解释个体级别的预测。
参考资料
Moon, I., LoPiccolo, J., Baca, S.C. et al. Machine learning for genetics-based classification and treatment response prediction in cancer of unknown primary. Nat Med 29, 2057–2067 (2023).
https://doi.org/10.1038/s41591-023-02482-6



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


