——简介——
AF2可以在有MSA足够多的情况下,即使没有模板也可以以非常高的置信度预测单体蛋白的结构;而对于瞬时作用的蛋白质复合物而言,这种信号却不是那么容易捕获。容易想到的就是增强采样结合打分函数来筛选出高置信的结构。本文主要介绍目前常见并可泛化的几类方法,并用例子加以说明。
——方法一:更改MSA输入——KaiB是在蓝藻细菌中发现的一种昼夜节律蛋白,它采用两种具有不同二级结构的构象作为其功能的一部分:在白天,它主要采用“基态”(ground state)构象,其二级结构为βαββααβ(图1A);而到了晚上,他以fold-swith(FS)构象结合KaiC,此时二级结构类似硫氧还原折叠βαβαββα(图1B)。直接对其进行结构预测会发现AF2更偏向于动力学上折叠较困难的FS态(图1C)。通过更改MSA的输入[1],使用相似度最高的50条序列,结果预测得到了白天的“基态”结构,而用相似度最高的100条序列,却得到了FS构象。为了进一步解释,作者对MSA序列信息进行聚类,随后对每一类进行结构预测,结果发现每一类都对应一个结构分布,而这些打分较高的区域就对应到了基态构象和FS构象。随后作者还做了进一步的进化分析,发现当突变两个残基V68E和I83K时,就足以使得KaiB构象从基态转变至FS态(图2AB),而从结构上分析可以发现,这两个疏水残基的变化使得原本埋藏位置变得暴露,进一步增加了构象转变的可能(图2E)。此外,也有类似的方法用于研究GPCR构象转变采样[2]。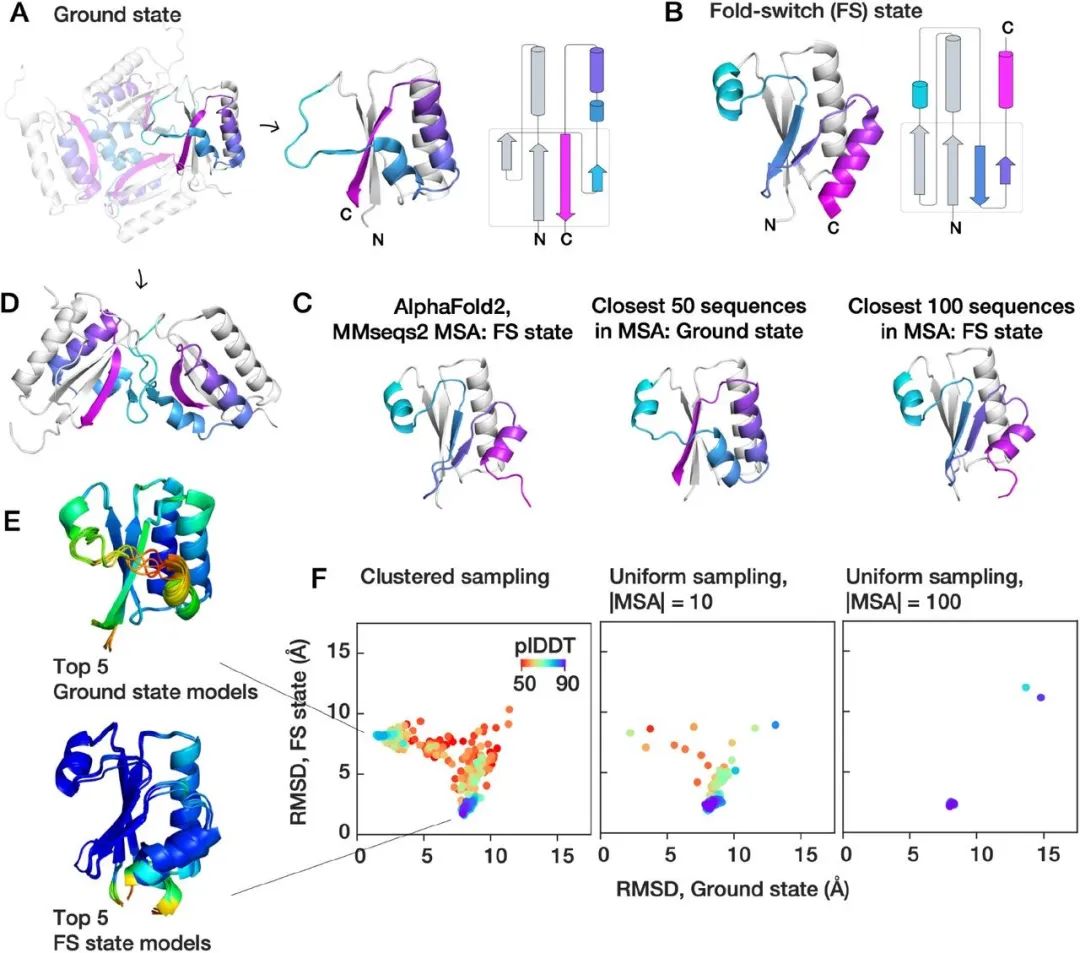 图1. 基于不同MSA输入的预测结构
图1. 基于不同MSA输入的预测结构
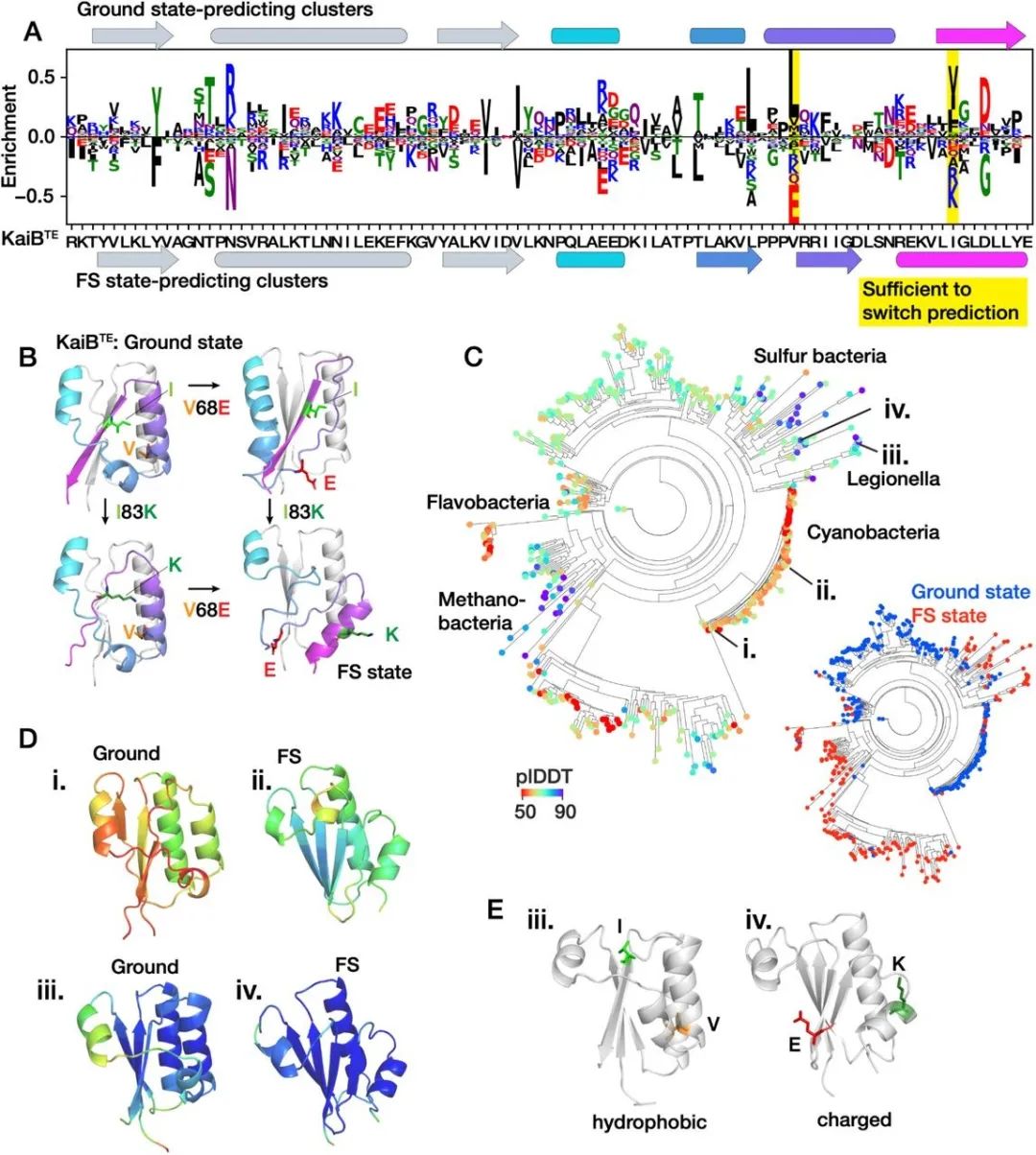
图2. 突变体的构象预测
——方法二:更改模型的dropout参数——
从之前的AF2 monomer版本和MultimerV2可以看到,模型采样数从5变到了25,这进一步说明采样的重要性。而dropout作为一种常见的过拟合正则化手段广泛用于深度学习训练中,但是却很少用于模型的预测中,通过将这一选项打开,例如在colabFold中,可以进一步增加模型输出的多样性。这里以AFsampler[3-4]为例,来说明这种方法的有效性。AFsampler通过将Evoformer部分的dropout激活,结构模块保持不变,最终产生6000个结构。通过在CASP15上的发现,与AF2multimer比起来,AFsampler在8个靶标上有了明显提升(图3ab)。
这里以H1144和T187o为例说明,H1144是nanobody相互作用,在这6000个模型中,只有3个模型给出了高质量的置信分数,即DockQ>0.8,而事实上,这堆生成的模型里边也只有5个模型是高质量的(图3cd)。如果没有这些采样,这些结构可能是采不到的。另一个例子是T1187o,这个例子是一个二聚体,AF2对于单体预测而言,置信度非常好,而对于dimer,DockQ值却接近0,通过提高采样数量,可以挑选出38个高质量的模型(图3ef);但是这些置信打分高的却未必是真实的模型,因此发展更准确的模型评估打分函数是非常有意义的,例如张贵军老师课题组开发的DeepUMQA3[5]和Cao Renzhi[6]老师课题组开发的ComplexQA。
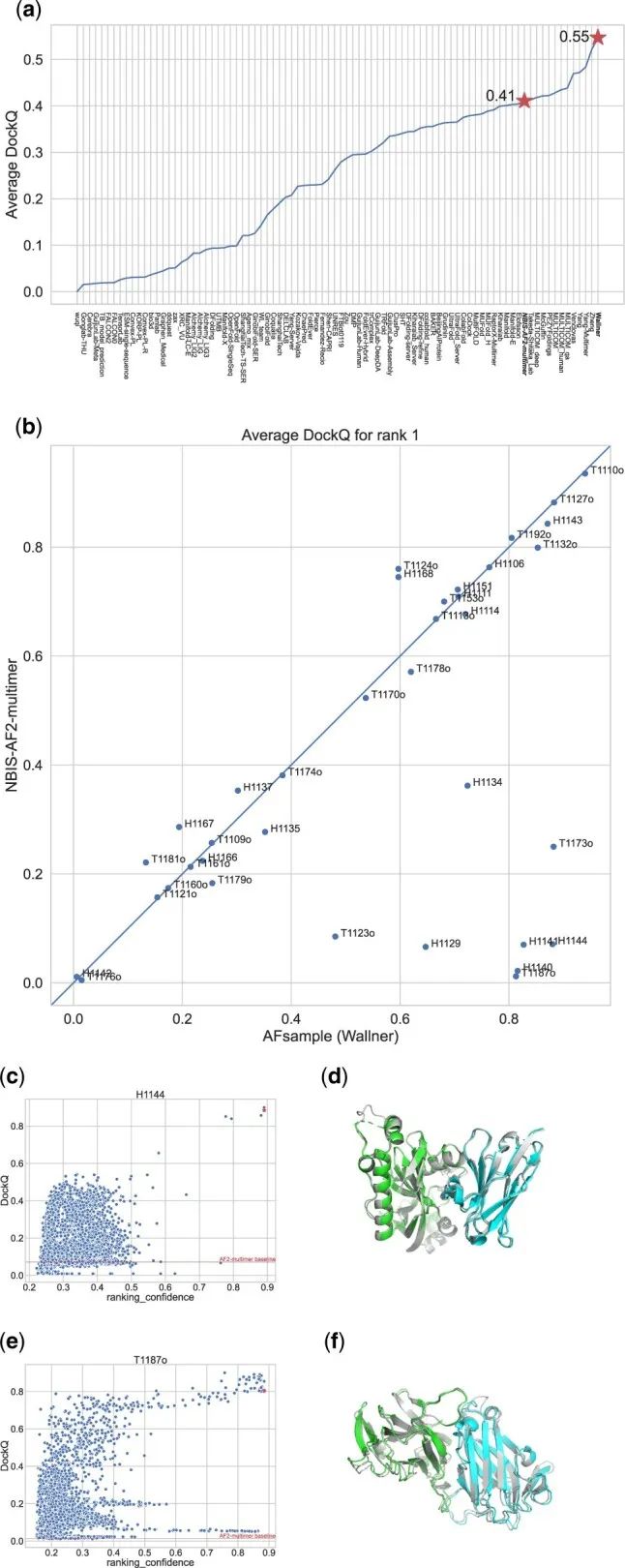
图3. AFsample性能比较和实例研究
——总结——
最近也有一些基于diffusion model做构象生成器的方法,相比于传统MD方法,这些方法效率会高很多.通过采取不同的构象,可以很好地为进一步研究蛋白质功能提供帮助。
参考文献:[1] Wayment-Steele, Hannah K., et al.
“Prediction of multiple conformational states by combining sequence
clustering with AlphaFold2.” BioRxiv (2022): 2022-10.
[2] Del Alamo, Diego, et al. “Sampling
alternative conformational states of transporters and receptors with
AlphaFold2.” Elife 11 (2022): e75751.
[3] Wallner, Björn. “Improved Multimer
Prediction using Massive Sampling with AlphaFold in CASP15.” (2023).
[4] Wallner, Björn. “AFsample: improving
multimer prediction with AlphaFold using massive sampling.” Bioinformatics 39.9
(2023): btad573.
[5] Liu, Jun, Dong Liu, and Gui-Jun Zhang.
“DeepUMQA3: a web server for accurate assessment of interface residue
accuracy in protein complexes.” Bioinformatics 39.10
(2023): btad591.
[6] Zhang, Lei, et al. “ComplexQA: a deep
graph learning approach for protein complex structure assessment.” Briefings
in Bioinformatics 24.6 (2023): bbad287.
作者:刘佳乐审稿:王丽莹编辑:王丽莹
GoDesignID:Molecular_Design_Lab( 扫描下方二维码可以订阅哦!)





