今天为大家介绍的是于2023年发表在Frontiers in Bioengineering
and Biotechnology的一篇研究蛋白质二级结构预测(Protein secondary structure prediction,PSSP)模型的论文。本文提出了一种将BTCN、BLSTM、MSBTCN相结合的模型,在使用较小模型体积的基础上,平衡序列的局部和全局特征,提高了PSSP的精度。

1. 介绍
蛋白质二级结构预测(Protein secondary structure prediction,PSSP)作为生物信息学的一大研究热点,是一项重要的任务。蛋白质一级结构由氨基酸残基的线性排列组成,二级结构是肽链按一定规律卷曲或折叠形成的特定空间结构,基于二级结构进一步折叠可以形成三级结构。蛋白质二级结构作为连接一级结构和三级结构的桥梁,PSSP技术的改进不仅有助于理解蛋白质的结构和功能,而且可以更好地预测三级结构,此外,PSSP还可以促进药物设计。然而,用于PSSP的生物技术耗时且昂贵,因此本文使用计算机和深度学习改进二级结构预测的方法。
传统的SVM等机器学习方法不能很好地提取序列中的全局信息,用于PSSP领域的深度学习方法通常具有复杂的网络结构和较高的计算成本,且对于高维长蛋白质序列,现有方法缺乏长距离特征提取能力,忽略长程依赖关系,现有的单一模型无法提取出复杂序列中的关键信息。对于目前PSSP领域的上述问题,做出了六项贡献:①基于TCN模型进行改进,提出BTCN模型,能够提取序列中的双向依赖关系;②提出MSBTCN模型,不仅能够提取残基的双向依赖特征,并且能够保留隐藏层的特征信息;③提出将BTCN、BLSTM、MSBTCN相结合的模型,提高了PSSP的精度;④分别将BLSTM与五种模型相结合,可以有效解决模型对长序列中长距离依赖特征提取能力低的缺点;⑤通过实验表明二级结构反向预测优于正向预测;⑥通过实验证明,三态和八态PSSP特征融合可以进一步提高二级结构的预测性能,为PSSP提供了一种新的思路。
2. 方法
BLSTM模型:
LSTM由前向LSTM和后向LSTM组成。LSTM可以自动决定丢弃不重要的信息并保留有用的信息。对于时间t的标准LSTM单元,输入特征表示为xt,输出表示为ht,单元状态表示为ct。LSTM单元中的遗忘门f、输入门i和输出门o计算如下:
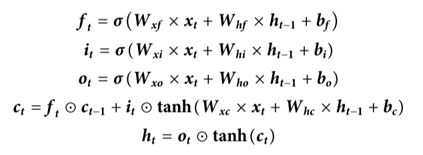
其中σ是sigmoid函数,W是权重矩阵,b是偏置项,☉是逐元素乘法,tanh是双曲正切函数。
TCN模型:
TCN在序列处理方面具有优秀的性能,能够避免训练过程中的梯度问题,此外,TCN还具有计算速度快、内存少、并行操作、感受野灵活等特点。
TCN使用一维全卷积网络架构,输入层的长度与每个隐藏层的长度相同,并添加零填充以保持前后层的长度相同。因此,TCN可以将任何长度的序列映射到相同长度的输出序列。此外,TCN使用因果卷积,其中当前时间的输出仅由当前时间和过去时间的特征输入确定,因此,TCN中的信息不会从未来泄漏到过去。
然而,因果卷积在处理需要长期历史信息的序列时具有不可避免的局限性。因此,网络使用扩张卷积来增加感受野,获得非常长的有效历史信息,定义为:
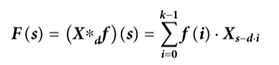
其中F(s)是扩张卷积运算,X是输入特征,d是扩张因子,f是滤波器,s是序列的元素,k是滤波器大小,s-d· i表示过去的方向。随着层数和膨胀因子继续增加,顶层的输出将包含更宽范围的输入信息。
如图1所示,网络引入残差连接以保证高维输入的训练稳定性,定义为:
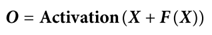
其中X表示块的输入,F(X)表示一系列运算后块的输出。
TCN架构由多个残差块组成。如图1所示,残差块包含膨胀的因果卷积层、权重归一化层、ReLU层和dropout层。TCN将每个块的输入与块的输出相加(当输入和输出之间的通道数不匹配时,对输入特征进行1 × 1卷积)。
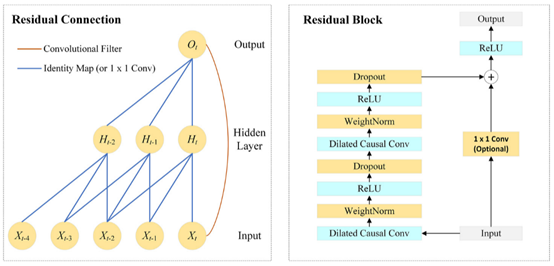
图1 TCN模型架构
BTCN模型:由于TCN使用膨胀因果卷积,它只能将信息从过去传输到未来。然而,二级结构的识别不仅由先前位置的氨基酸决定,而且还受后续位置的氨基酸的影响。单向传输的TCN显然不能满足氨基酸特征的全面提取,因此本文提出BTCN模型,以充分捕捉残基之间的双向深度依赖关系。
如图2所示,BTCN的架构由前向TCN和后向TCN组成。由于扩张因果卷积对序列进行单向运算,BTCN将反向序列输入到后向TCN中进行网络的反向特征提取。
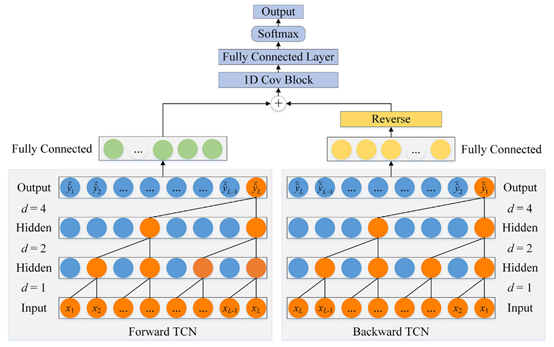
图2 BTCN模型架构
令X表示蛋白质序列,L是X的长度,BTCN可以表示如下:
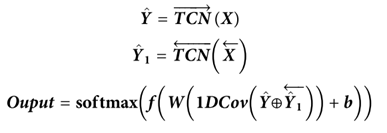
网络在当前时刻t的输出由整个序列的输入确定,其计算为:
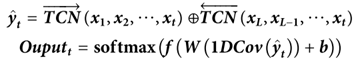
BTCN可以利用双向深度相互作用,通过前向和后向提取残基特征来促进二级结构识别。此外,BTCN不限于PSSP,它适用于所有需要全局语义的序列。
MBTCN模型:
如图3所示,MSBTCN可以利用所有层的完整信息进行预测,有效防止了隐藏层权重信息的浪费,具有n个残差块的单向MSTCN的输出为:
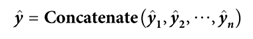
MSBTCN不仅可以捕获双向深度特征,而且可以利用中间残差块的关键信息进行预测。
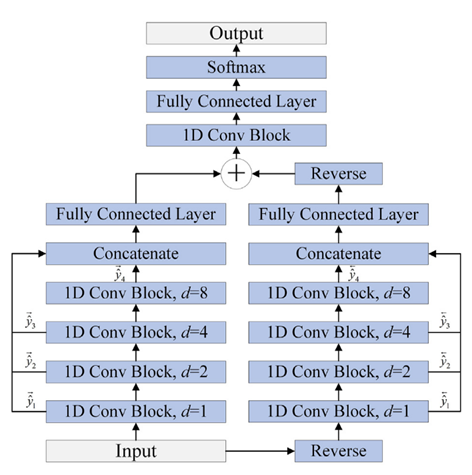
图3 MSBTCN模型架构
本文提出的模型:
为了更好地改进二级结构的预测,本文提出的模型使用BTCN、BLSTM和MSBTCN来提取残基序列的深度相互作用,模型架构如图4所示。该模型可分为五个部分:输入、BTCN模块、BLSTM模块、MSBTCN模块和输出。
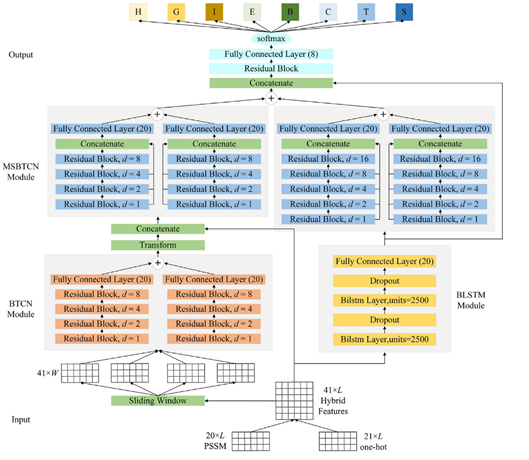
图4 本文提出模型的架构
在输入部分,模型首先将蛋白质序列转化为20维PSSM特征和21维one-hot特征,然后,本文使用混合特征PSSM+one-hot作为模型的输入。
在BTCN模块中,为了使网络能够提取局部特征,本文使用滑动窗口技术将输入特征分割成41 × W的短序列,其中W是窗口大小。BTCN用于序列到序列预测的输入和输出必须是相同的长度,因此本文将与分段氨基酸特征对应的二级结构标签放在W位置,并将剩余位置填充为0。然后,本文使用修改后的BTCN提取氨基酸序列中的双向深度局部依赖。由于W通常小于20,因此本文使用四个残差块足以捕获序列中的双向氨基酸信息。本文在残差块中使用三个具有相同膨胀因子的膨胀的因果卷积层。在膨胀的因果卷积层后添加实例规范化层用来加速模型收敛,添加ReLU激活层用来防止梯度消失,添加dropout层用来避免模型过拟合。本文使用Transform层将提取的局部特征处理成20 × L序列,然后使用Concatenate层将局部特征与输入特征合并为61 × L的序列特征矩阵。
在BLSTM模块中,本文使用两个具有强大分析能力的双向LSTM层提取蛋白质序列中的关键全局相互作用,此外,本文添加两个dropout层,以确保训练期间的梯度稳定性。
在MSBTCN模块中,本文分别使用四个和五个残差块优化提取的局部和全局特征,同时进一步捕捉氨基酸残基之间更深层次的双向长程依赖关系,可以更全面地保留隐藏层的重要信息。由于MSBTCN具有灵活的感受野和稳定的计算能力,可以更准确地交互和控制序列信息,同时快速优化和融合提取的特征。在输出部分,本文使用一个残差块来处理和优化MSBTCN和BLSTM模块提取的特征,最后使用一个全连接层和softmax函数来完成分类。
此外,本文还分别提取了三态和八态PSSP中模型输出部分的Concatenate层特征,并将其融合为80维特征用于二级结构预测,其中融合特征表示为FF 3 – 8,可以更好地模拟输入特征和二级结构之间的序列–结构映射关系。该方法可以有效地利用更复杂和更长期的全局依赖关系,通过对蛋白质序列的综合处理来提高PSSP的准确性。
3. 结果
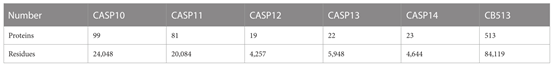
本文使用PISCES服务器从蛋白质数据库PDB中生成序列列表,共选择14991条序列组成CullPDB数据集,这些序列基于25%的同一性截止值、0.25的R因子截止值和3μm的分辨率截止值进行筛选。本文去除长度小于40或大于800的蛋白质,最终的CullPDB数据集包含14562条蛋白质序列。为了更好地评估模型性能,本文进一步将数据集随机分为三个部分:训练集(11650条序列),验证集(1456条序列)和测试集(1456条序列),所有实验的结果均由三次独立实验的平均值获得。为了评估所提出的模型的性能,本文还使用CASP10、CASP11、CASP12、CASP13、CASP14和CB513数据集作为测试集,这六个独立测试集的蛋白质序列和残基的数量如表1所示。
表1 六个独立测试集所含蛋白质序列和残基的数量
本文对模型的性能进行分析,所使用的数据集是CullPDB的验证集和测试集,模型中没有使用FF3-8特征。从图5(A)和图5(B)中可以看出当滑动窗口设置为19时,模型在三态和八态蛋白质二级结构预测上的性能都为最优。从图5(C)和图5(D)中可以看出,当设置滤波器个数为512个,卷积核大小为5时,模型的性能达到最优。造成这种实验结果的主要原因是卷积核的大小决定了局部捕获的程度,从而影响残差之间关键特征的提取。此外,卷积中的通道数量不仅影响预测性能,而且还决定了模型的大小和训练时间。
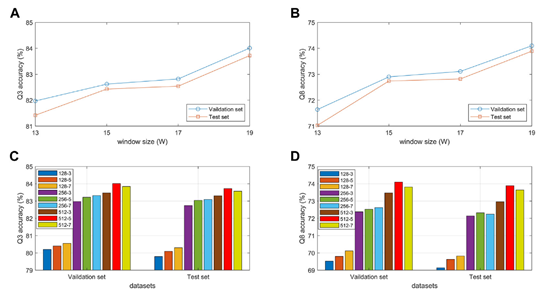
图5 模型性能分析实验(BTCN)
为了验证BLSTM模块中隐藏层单元个数对所提出的模型性能的影响,本文对具有不同隐藏层单元数1000,1200,1500,1800,2000,2200和2500的模型在验证集和测试集上进行了实验分析。图6(A)和图6(B)展示了模型在两个数据集上的分类准确度随着隐藏层单元的数量增加而增加。当隐藏层单元数为2500时,模型在的预测性能最好。主要原因是隐藏层单元的数量决定了高维蛋白质序列的表达能力。但需要注意的是,如果隐藏单元的数量太大,模型不仅会减慢训练速度,还可能出现过拟合问题。
由残差块组成的MSBTCN模块的性能与残差块的数量密切相关,因此本文分别用不同的残差块数量来优化提取的局部特征。图6(C)和图6(D)展示了所提出的模型在验证集和测试集上的预测精度。从图6中可以看出,当模型使用4个残差块时,两个数据集上的Q3精度达到最大值,而当分别使用4个和5个块时,两个数据集上的Q8精度达到最大值。原因是当模型的深度不够时,模型无法捕获更深的依赖关系,但随着深度的增加,模型会增加复杂性和过拟合的风险。
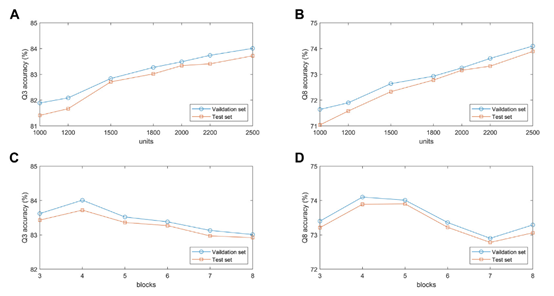
图6 模型性能分析实验(BLSTM/MSBTCN)
为了证明BTCN模型对PSSP的有效性,本文在七个数据集上比较了TCN,RTCN和BTCN的性能。在实验中,本文使用四个残差块来提取特征,其中所有模型具有相同的参数。模型输入是大小为41 × L的混合特征,其中L是蛋白质序列的长度。如表2所示,BTCN的预测性能在所有数据集上都有很大的提高。与TCN相比,BTCN的Q3精度和Q8精度在7个数据集上分别平均提高了4.74%和5.43%。实验结果充分证明了BTCN模型的优秀性能,能够有效捕捉残基间的双向深度相互作用,提高预测精度。
表2 单向和双向PSSP实验(总体性能)
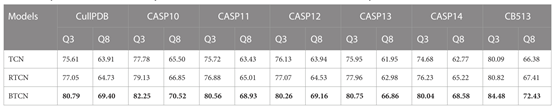
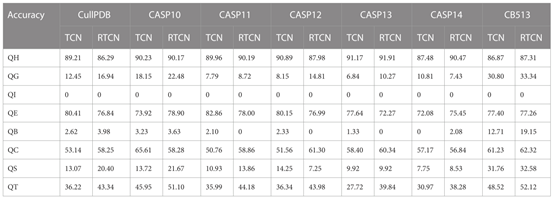
此外,表2还显示,在7个数据集上,RTCN的预测精度始终优于TCN,Q8精度平均提高了1.18%。结果表明,二级结构的反向预测上级优于正向预测,这也表明单向提取特征时,位于较后位置的氨基酸对二级结构的整体识别影响更大。TCN预测准确率低的主要原因是其较宽的感受野在不使用滑动窗口技术时忽略了相邻氨基酸的重要信息,但对整个序列的预测能更好地反映前后位置氨基酸对PSSP的影响。TCN和RTCN的单种预测精度见表3。该表显示,在大多数数据集上,H、G、B、C、S和T类型的准确性有所提高,而E类型的准确性有所下降。这也证明了大多数二级结构类型的识别与来自后面位置的氨基酸信息更相关。
表3 单向和双向PSSP实验(每种类型的性能)
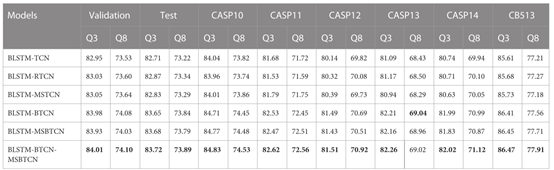
为了验证PSSP中不同特征提取模块的性能,本文分别将BLSTM与TCN,RTCN,MSTCN,BTCN和MSBTCN相结合,提出了六种新的深度学习模型,本文在所有模型中使用相同的特征提取过程和参数。如表4所示,BLSTM-BTCN-MSBTCN模型在除CASP13外的8个数据集上实现了最高的Q3和Q8精度。此外,可以观察到BLSTM-RTCN对大多数序列的预测性能优于BLSTM-TCN,这表明二级结构的反向预测可以达到更高的准确性。序列经过滑动窗口法处理后,前后位置的氨基酸对预测性能的影响差别不大,因此RTCN的优势并不明显。表4显示,BLSTM-BTCN和BLSTM-MSBTCN模型的预测性能明显优于所有数据集上单向特征提取的模型,这证明本文提出的BTCN和MSBTCN可以充分利用双向长距离相互作用来提高预测精度。虽然BLSTM-MSBTCN可以捕获多尺度特征信息,但在大多数数据集上,其预测精度不如BLSTM-BTCN。其主要原因是BTCN在提取滑动窗口分割的短序列局部特征时不会过多地浪费中间层信息,而MSBTCN则可能引入不必要的信息。为此,本文在BLSTM-BTCN-MSBTCN模型中分别使用BTCN和MSBTCN提取双向局部和长程特征,不仅能更好地提取短序列中的特征,而且能更好地保留长序列中的有用信息。BLSTM-BTCN-MSBTCN模型可以联合收割机各模块的优点,提取不同的特征,提高预测精度。
表4 六种组合模型实验
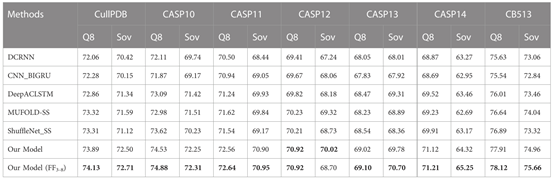
本文在七个数据集CullPDB,CASP10,CASP11,CASP12,CASP13,CASP14和CB513上将所提出的模型与五个最先进的模型进行比较。在比较的模型中,DCRNN是一种端到端的深度网络,它使用具有不同内核大小的卷积神经网络和具有门控单元的递归神经网络来提取蛋白质序列中的多尺度局部特征和长程依赖关系。CNN_BIGRU结合卷积网络和双向GRU来预测二级结构。DeepACLSTM结合了非对称卷积网络和双向长短期记忆网络,以提高二级结构预测精度。这三种模型都是卷积神经网络和递归神经网络的组合,但它们的结构不同。MUFOLD-SS使用深度inception-inside-inception网络来处理序列中的局部和全局依赖关系。ShuffleNet_SS使用轻量级卷积网络和标签分布感知的保证金损失来提高网络对稀有类的学习能力。为了进行公平的比较,本文使用相同的数据集进行模型训练,输入是混合特征PSSM+one-hot。
所提出的方法和五种先进模型在基准数据集上的预测结果如表5和表6所示。表中显示,本文的模型在7个数据集上的预测性能始终优于五种先进模型。这主要是因为本文提出的模型具有强大而全面的特征提取能力,使得残差序列中的双向深度局部和远程相互作用能够被充分提取并用于预测。与本文没有使用FF3-8特征的模型相比,使用FF3-8特征的模型在所有数据集上都实现了最佳的三态PSSP性能,在大多数情况下也实现了最高的八态PSSP精度。实验结果表明,三态和八态PSSP之间的重要相关性可以相互促进二级结构的识别,在添加八态PSSP特征之后,三态PSSP的精度显著提高。
此外,本文模型大小为13.8MB,使用F3-8特征的模型大小为14.3 MB。DCRNN、CNN_BIGRU、DeepACLSTM、MUFOLD-SS和ShuffleNet_SS的模型大小分别为18.1MB、15.8MB、20.6MB、17.6 MB和3.9MB。虽然本文的模型参数大小仅优于四种先进模型,但它在PSSP任务中达到了最先进的性能。对于高维长序列,本文模型还可以有效利用广泛而灵活的感受野来捕获残基之间的长期关键依赖关系,因此可以更好地模拟序列和结构之间的复杂关系。
表5 与先进模型的对比实验(三分类)
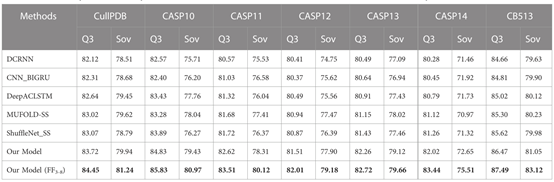
表6 与先进模型的对比实验(八分类)
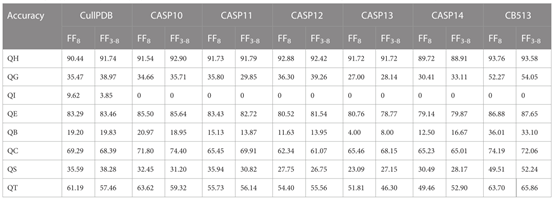
在八态PSSP中,所提出的模型在7个数据集上不使用F3-8特征的模型和使用F3-8特征的模型的单类型准确度如表7所示。从表7中可以看出,八种结构类型的预测精度存在明显差异。主要原因是各类型出现的频率相差太大,结构类型I出现频率几乎为0。从表7中可以看出,添加了三态PSSP的特征之后,在大多数数据集上,结构类型G、E、B和T的准确性得到了提高。
表7 FF8与FF3-8特征对比实验(八分类)
4. 结论
本文提出了一种新的PSSP深度学习模型。该模型基于滑动窗口技术使用BTCN模块提取双向局部依赖特征;同时使用BLSTM模块提取序列的全局依赖特征;最后使用MSBTCN模块进一步捕获残基之间的双向关键长程依赖关系,同时更好地优化和融合提取的特征,从而防止隐藏层中的信息浪费。本文提出的模型具有较强的稳定性和特征提取能力,不仅能够有效解决序列中长程依赖特征提取不足的缺点,也克服了各模块的弱点。
由于三态二级结构预测和八态二级结构预测之间的密切相关性,本文使用融合特征FF3-8,以进一步提高PSSP的性能,这是PSSP领域创新性的想法。最后,本文通过完整且全面的实验分析,证实了所提出模型的先进性能。



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


