编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Fabian J. Theis & Nir Yosef团队的一篇论文。当前估算 RNA 流速的方法缺乏有效的策略来量化不确定性并确定其对感兴趣系统的整体适用性。在这里,作者介绍了 veloVI(velocity variational inference),这是一个用于估算 RNA 流速的深度生成模型框架。veloVI 学习了基因特异性的 RNA 代谢动态模型,并提供了转录组范围内的流速不确定性量化。

单细胞 RNA 测序(scRNA-seq)技术的进步有助于高分辨率解析细胞分化和其他时间过程的机制。尽管 scRNA-seq 是一种破坏性的检测方法,但在轨迹推断的任务中,科研人员开发了一系列计算方法,利用生物过程的动态和不同步性,对细胞按照所谓的“伪时间”进行排序。传统的轨迹推断方法通常要求知道底层生物过程的初始状态,并使用流形学习来确定一个度量空间,其中的距离能够捕捉分化状态的变化。近来,RNA 流速已经成为轨迹推断任务的一种自底向上的机械性方法。RNA 流速描述了剪接的信使 RNA(mRNA)随时间的变化,它利用标准的 scRNA-seq 协议同时检测未剪接和剪接的 RNA 转录本。在估算 RNA 流速后,通常有两种方式将其纳入分析:(1)推断细胞特异性的分化伪时间;(2)构建一个过渡矩阵来诱导数据上的马尔可夫链,从而确定细胞的初始、过渡和终止亚群。
目前有两种流行的方法用于估算 RNA 流速。第一种方法被称为稳态模型,它假设(1)RNA 的转录和降解率是恒定的;(2)存在单一的全局剪接率;(3)细胞在诱导阶段达到平衡状态,并不包括基础转录;(4)基因间的独立性。第二种方法被称为 EM 模型,它在 scVelo 包中得到了描述和实现。与稳态模型不同,EM 模型不假设系统达到了稳态,它推断一整套转录参数,并通过期望最大化(EM)框架为每个细胞和每个基因估算潜在时间。虽然这些方法已经成功地用于解释单细胞动态,但它们也受到模型假设和下游使用的限制。例如,这两种方法都缺乏全局的不确定性概念。因此,评估 RNA 流速估计的稳健性,或决定流速分析在多大程度上适用于给定的数据集可能是困难的。尽管 EM 模型可以用来对潜在的“驱动”基因进行排名,但基因的可能性与可视化和正确性之间没有直接的联系。
为了解决这些问题,作者提出了 veloVI(velocity variational inference),一种用于估算 RNA 流速的深度生成模型。VeloVI 通过一个模型重新表述了 RNA 流速的推断,该模型在所有细胞和基因之间共享信息,同时学习与 EM 模型中相同的量,即动力学参数和潜在时间。作为其输出,veloVI 返回 RNA 流速的经验后验分布(由细胞、基因和后验样本组成的矩阵),这可以被纳入对结果的下游分析中。在这里,作者展示 veloVI 在数据拟合方面相对于 EM 模型有显著的改进。此外,它还提供了一层缺失于先前方法的解释和模型评估,同时也极大地提高了模型扩展的灵活性。
用于估算 RNA 流速的变分推断模型
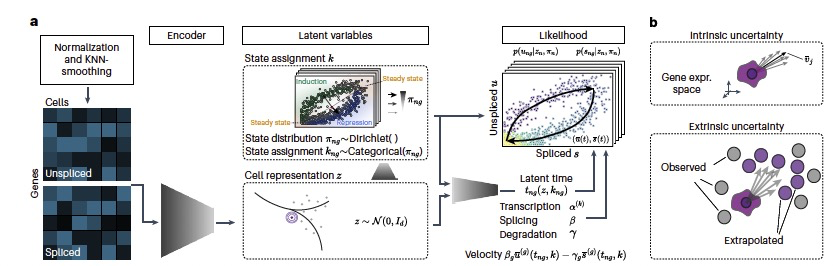
图 1
VeloVI 假设每个基因在细胞中的未剪接和剪接 RNA 的丰度是由动力学参数(转录、剪接和降解速率)、潜在时间和潜在转录状态(诱导状态、抑制状态及其各自的稳态)的函数生成的。此外,veloVI 假设每个基因的潜在时间(每个细胞)通过细胞表示的低维潜变量相连。这些表示捕捉到一个细胞的观察状态是多个同时进行的过程的组合,这些过程共同构成了表型流形。这种建模选择是由观察结果证明的,即在 EM 模型中,这是独立于基因拟合的,推断的潜在时间矩阵(细胞与基因的形状)具有低秩结构。
VeloVI 的完整架构表现为一个变分自编码器。编码器神经网络以细胞的未剪接和剪接丰度为输入,并输出细胞表示和潜在转录状态变量的后验参数。基因级别的、状态特定的、潜在时间由一个神经网络参数化,该神经网络以细胞表示的样本为输入。然后,细胞未剪接和剪接丰度的似然函数是潜在时间、动力学速率参数和状态分配概率的函数(图 1a)。模型的参数使用标准的基于梯度的程序同时优化。优化后,细胞基因特定的速度被计算为降解率、剪接率和拟合的未剪接和剪接丰度的函数,这些直接合并了时间和转录状态上的后验分布。作为一个贝叶斯深度生成模型,veloVI 可以输出一个在细胞基因级别上的速度的后验分布。这个分布可以用来量化细胞在基因空间中第一阶方向上的内在不确定性。在下游分析中,速度通常用于构建细胞-细胞转换矩阵,该矩阵根据细胞和其邻域在第一阶位移的相似性,对最近邻图的边缘进行重新加权。通过将后验速度样本传递到这个计算中,作者还量化了一个外在不确定性,反映了内在不确定性和细胞在基因空间中邻域的变异性(图 1b)。与此相反,EM 模型和稳态模型都不携带任何明确的不确定性概念。实际上,这两种先前的模型只允许在事后评估不确定性,基于量化细胞邻域上的速度变化。最后,对于一个细胞平均样本的速度的点估计,使得 veloVI 的输出可以直接用于 scVelo 的下游可视化和图构建。
veloVI改进了数据拟合效果
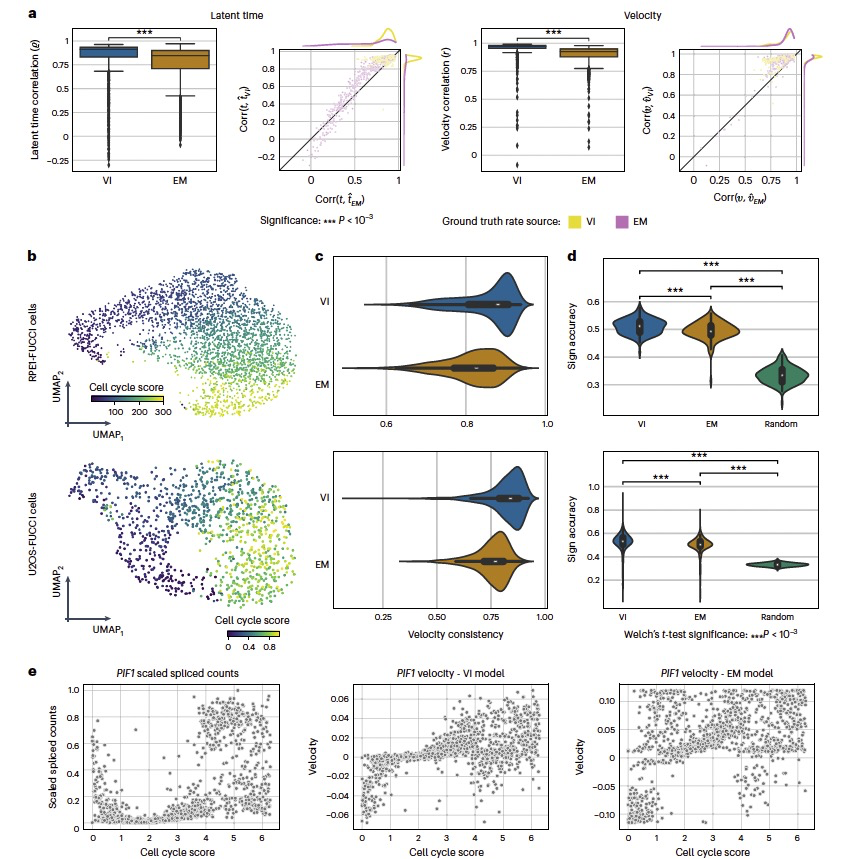
图 2
作者做了多方面的分析,以评估veloVI在一系列模拟和真实数据集中稳健拟合转录动态的能力,与EM模型和scVelo包中实现的稳态模型方法进行比较。作者首先评估了每个模型在模拟数据中恢复动力学参数的能力。veloVI的性能优于EM模型,并且在恢复每个基因的降解率和剪接率的模拟比率方面优于稳态模型。同样,与使用真实数据预先估计的参数模拟数据的EM估计相比,veloVI推断的潜在时间和速度与基本事实显著更好地相关。值得注意的是,这些模拟反映了一种理想化的情景,因为细胞是通过EM模型的生成过程模拟的。尽管如此,即使在这些有利于EM模型的条件下,veloVI的性能也优于EM模型。作者还测试了veloVI和EM模型的运行时间。对于这个比较,作者在包含大约114,000个细胞的小鼠视网膜数据集的子样本上运行了两个模型。在多个子样本中,使用veloVI的推断速度明显快于EM模型。为了进一步验证veloVI的准确性,作者在细胞周期数据集上比较了veloVI和EM模型。所用数据集是基于荧光泛素化细胞周期指示器(FUCCI)的RPE1和U2OS细胞的细胞周期数据集,它提供了通过蛋白质衍生的细胞周期得分的方向性/时间的正交验证(图2b)。为了评估模型性能,作者首先比较了每个模型生成的速度矢量场的局部一致性。这种一致性度量量化了具有相似转录组轮廓的细胞(最近的邻居)的速度在多大程度上一致,并依赖于速度在表型流形上平滑变化的假设。与EM模型相比,veloVI实现了更高的速度一致性(图2c)。作者还测试了基因水平上的速度方向是否与基于细胞周期的基本事实启发式方法一致。与以前一样,veloVI产生了一致的结果,并优于EM模型(图2d)。
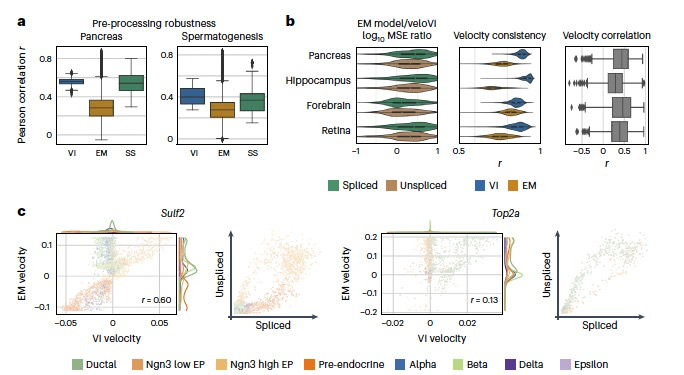
图 3
随后,作者评估了在用12种不同的RNA丰度量化算法处理的真实数据集上的速度估计的稳定性。为此,作者在五个基准示例上测量了每个细胞的速度之间的相关性,分别是胰腺内胚胎发生,以及精子发生,小鼠发育中的齿状回,小鼠前额叶皮层和21-22个月大的小鼠大脑的数据集。与EM模型相比,veloVI在相关性上更好并且有更低的方差。与更简单的稳态模型相比,veloVI具有相似的平均相关性,但方差更低(图3a)。为了评估推断的动态反映观察到的数据的程度,作者计算了未剪接和剪接丰度拟合的均方误差(MSE),并将MSE与EM模型在一组数据集上的MSE进行比较,veloVI在每个数据集的大多数基因中表现更好(图3b)。
veloVI 实现了可解释的速度分析
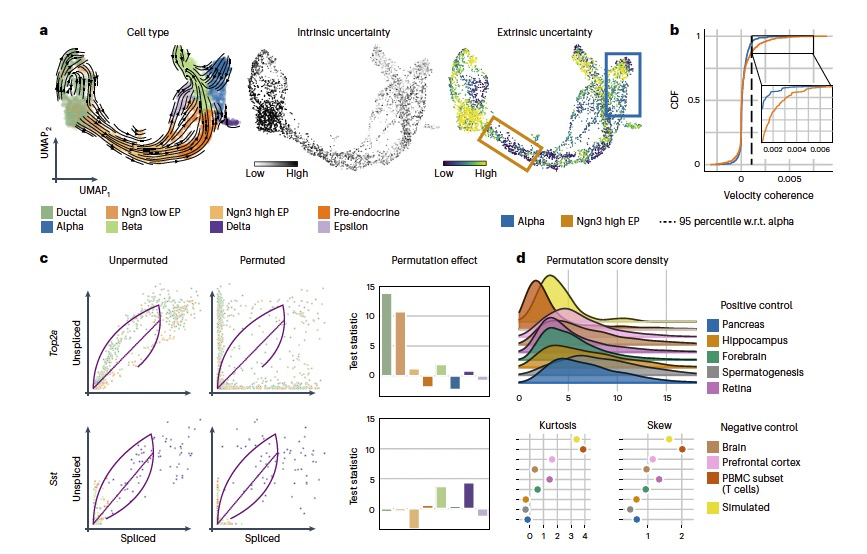
图 4
接下来,作者调查了veloVI的速度估计的不确定性如何用于检视其输出结果,包括细胞层面(可能被错误建模的)和单个基因层面(可能与汇总的细胞层面输出不一致的)。作者利用这种不确定性来(1)测量每个细胞中由速度向量建议的表型方向性的变异性(这里称为内在不确定性),以及(2)量化在速度诱导的细胞-细胞转换矩阵下预测的未来细胞状态的变异性(这里称为外在不确定性;图1b)。作者将这些不确定性度量应用于胰腺数据集(图4a)。可以观察到,在导管细胞和低表达Ngn3的内分泌前体细胞群体中,内在不确定性较高。同时,外在不确定性不仅在这些细胞群体中较高,还在终末的α和β细胞群体中较高。这些结果表明,较低的内在不确定性并不一定预示着较高的外在不确定性。前者依赖于估计速度向量(这是细胞内在的),而大多数速度线还会考虑数据集中的其他细胞,这些细胞可能代表细胞的潜在过去和未来状态,以确定细胞转换。为了进一步了解这些不确定性度量捕获的数据方面,作者通过多种扰动胰腺数据集的方式,发现对于每种扰动源,内在不确定性随着扰动强度的增加而增加,外在不确定性的反应类似。这些结果表明,不确定性度量可以捕获数据中的随机噪声,以及转录本的测量方式的偏差。
参考资料
Gayoso, A., Weiler, P., Lotfollahi, M. et al. Deep generative modeling of transcriptional dynamics for RNA velocity analysis in single cells. Nat Methods (2023).
https://doi.org/10.1038/s41592-023-01994-w



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


