点击蓝字 关注我们
AItellU
编者 | 刹那芳华
制版 | 戈戈

第一作者:Gi Bae Kim
通讯作者:Sang Yup Lee
DOI:10.1038/s41467-023-43216-z
1
全文一句话速览
本文开发深度学习模型DeepECtransformer,利用Transformer作为神经网络结构来预测未注释基因的酶学委员会(EC)编码,并通过实验验证了三种蛋白质(YgfF、YciO和YjdM)的酶活性预测。
2
研究背景
酶在生物体内催化各种反应中起着关键作用,理解酶的功能对于理解代谢过程十分重要。EC编码系统基于酶的功能进行分类,已经开发了深度学习模型来预测酶的EC编码。本文着重介绍了一个名为DeepECtransformer的深度学习模型的开发和评估,该模型利用Transformer层来有效预测各种酶的EC编码。DeepECtransformer模型在大型酶序列数据集上进行了训练,并表现出优于其他方法的性能。它还证明了纠正UniProt知识库中的错误注释的能力。
3
全文亮点
1. DeepECtransformer能够有效地预测各种酶的EC编号,并且在性能上优于其他方法
2. 能够纠正UniProt Knowledgebase中错误的EC编号,并学习识别酶序列中的重要区域
4
图文解析
DeepECtransformer的开发和评估
DeepECtransformer的网络架构,包括两个transformer编码器、两个卷积层和一个线性层(图1a)。通过对酶的氨基酸序列进行预测,该神经网络可以预测酶的EC编号。
在评估方面,作者使用了一个测试数据集,将DeepECtransformer与DeepEC和基于同源搜索的工具DIAMOND进行了比较。结果显示,DeepECtransformer在精确率、召回率和F1分数方面表现优秀,并且对于与训练数据集中的序列相似度较低的酶,其预测性能更具优势(图1b-d)。此外,DeepECtransformer还能够纠正UniProt Knowledgebase中误注的EC编号。
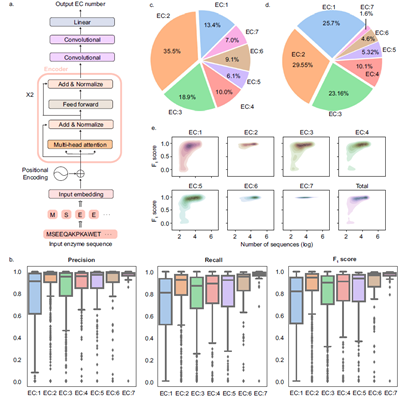
图1 DeepECtransformer的网络结构和神经网络的预测性能
人工智能学习酶的功能区域
作者使用了DeepECtransformer神经网络模型的自注意力层来识别酶的功能区域,并观察了模型在分类酶功能时所关注的区域。结果显示,AI能够有效地检测到一些功能区域,如活性位点和配体结合位点,以及一些已知的功能域,如Pfam域(图2)。这些发现有助于增进我们对酶功能的理解,并且可能有助于发现以前未知但关键的酶功能特征。
此外,作者还通过使用DeepECtransformer模型预测E. coli中未知蛋白质的功能,并通过体外酶活性实验证实了预测的酶功能。这表明DeepECtransformer不仅能够快速为越来越多的DNA序列注释酶功能,还能够发现以前未知的蛋白质的代谢功能。
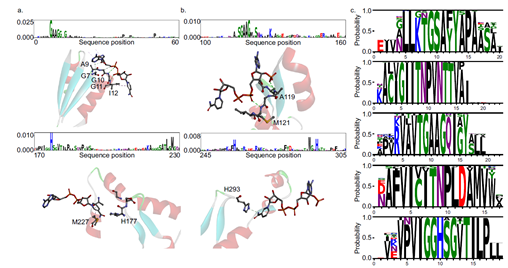
图2 通过DeepECtransformer神经网络突出显示氨基酸残基
大肠杆菌菌株等位基因代谢功能分析
使用DeepECtransformer模型和DIAMOND算法对1122个E. coli菌株的312,274个蛋白进行EC编号预测,以评估DeepECtransformer在预测不同菌株之间代谢功能变化的能力(图3a,b)。通过比较DeepECtransformer和DIAMOND的预测结果,发现在总体上有68.87%的基因的至少90%的等位基因在两种方法中具有相同的预测结果。
值得注意的是,通过分析预测结果不一致的基因,可以分析这些突变如何影响它们的代谢功能(图3c,d)。例如,本文报道了arol基因的若干等位基因发生了额外的代谢功能变化。这些观察结果从代谢角度为这些菌株的进化轨迹提供了宝贵线索。
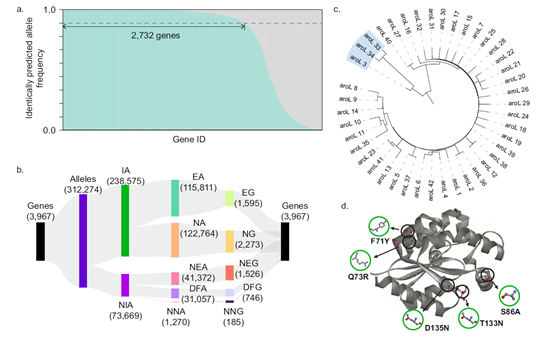
图3 1122株大肠杆菌312274个等位基因EC数预测结果
发现大肠杆菌k-12 MG1655中酶的未知功能
在 E. coli K-12 MG1655 的 y-ome 即功能未知区域中共有1600个基因。利用DeepECtransformer,从这些基因中成功预测了464个蛋白质的EC编号,并有390个蛋白质预测到了完整的四位数EC编号(图4a)。相比之下, DeepEC预测了82个蛋白质的EC编号,其中71个被预测为完整的四位数,而与其相关的UniProt数据库为这71个蛋白质提供了注释。这些结果表明DeepECtransformer能够准确预测酶功能,特别是对于功能尚未完全了解的基因。此外,通过分析模型预测中的重点区域,如活性位点或辅因子结合位点,揭示了DeepEC-transformer熟练识别酶序列中的重要区域的能力。最后,通过实验验证了模型预测的三个蛋白质(YgfF,YciO和YjdM)的酶活性(图4b),并证明了DeepECtransformer快速注释酶功能以及发现前所未知蛋白质代谢功能的能力。
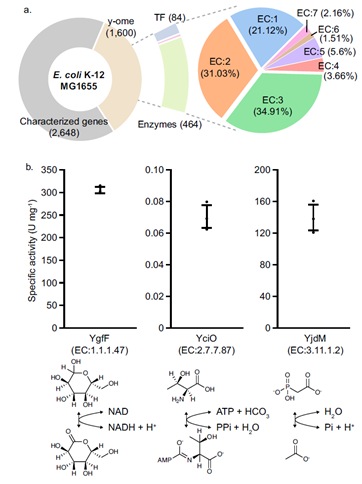
图4 大肠杆菌K−12 MG1655 y-ome蛋白的EC编号预测结
5
通讯作者介绍
Sang Yup Lee,韩国科学技术院化学与生物分子工程系教授。研究方向:1.代谢工程;2.蛋白质工程;3.生物高分子材料;4.基因组学;5.生物信息学。
6
编者有话说
DeepECtransformer算法通过结合深度学习和Transformer层来预测编码酶的功能注释。该算法使用神经网络和同源搜索两个预测引擎,其中神经网络利用Transformer架构从酶的氨基酸序列中提取潜在特征来预测EC编号。通过对酶的重要功能区域进行分析,DeepECtransformer还能够识别如活性位点和辅因子结合位点等。利用该算法,可以快速注释酶功能,发现以前未知的蛋白质代谢功能,并通过实验验证预测的酶功能。总的来说,DeepECtransformer算法在酶编码基因的功能注释中具有重要的作用。
转载须知
【原创文章】AItellU原创文章,欢迎个人转发分享,未经允许禁止转载,所刊登的所有作品的著作权均为AItellU所有。AItellU保留所有法定权利,违者必究。

投稿、转载及合作邮箱:
zjhuangjunjie@163.com
原文链接:
https://www.nature.com/articles/s41467-023-43216-z
点击下方蓝字阅读原文




 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง




