
导语
生命起源和人造生命是两个令人着迷且相互关联的领域,共同构成我们探索生命本质的历程。近日,来自北京师范大学、集智科学研究中心、上海交通大学的研究者合作在期刊 Quantitative Biology 上发表最新研究,回顾了人造生命领域常用的两种主要方法——自上而下的方法通过简化现有生物系统的复杂性,自下而上的方法通过整合定义明确的组件——并提出另一种“从生命起源开始的自下而上方法”。该方法仍然基于自下而上方法,同时汲取“生命起源”领域所收集的丰富结果,希望能够激发合成生物学家和实验者考虑更理论的角度,并促进生命起源研究者与人工合成生命研究者之间的交流。论文作者仇玮祎、刘宇、周理乾是集智俱乐部“自生成结构读书会”发起人,张泽成是集智科学研究中心生命起源知识图谱项目骨干。
关键词:人造生命,自催化集,梯径理论,生命起源,合成细胞
彭璐等 | 作者

论文题目:Theoretical perspective on synthetic man‐made life: Learning from the origin of life
作者:彭璐,张泽成,汪显意,仇玮祎,周理乾,肖辉,刘春秀子,唐绍华,秦志伟,蒋家坤,狄增如,刘宇
论文地址:
http://doi.org/10.1002/qub2.22
目录
摘要
1. 引言
2. 已有的努力:自上而下和自下而上的方法
3. 从生命起源的研究中学习:建立复杂性
4. 讨论和结论
摘要
在实验室中创造人造生命是生命科学中最引人入胜但也最具挑战性的问题之一。系统生物学与合成生物学的相关理论为人造生命领域的进一步探索和理解奠定了基础。但在这个领域中关于生命设计的指导理论尚有巨大空白,而与生命起源相关的理论将有可能为人造生命提供大量的定量数学模型和工具。本文回顾了人造生命领域常用的两种主要方法:自上而下的方法可以简化现有生物系统的复杂性,而自下而上的方法通过将定义明确的组件集成、组合、拼装来实现。本文将介绍它们的理论基础、最新进展以及局限性。之后,我们提出了这两种方法之外的另一种可能的方法,即“从生命起源开始的自下而上方法”:从建立自催化化学反应网络开始,利用物理边界作为初始区室,然后设计定向进化系统,期望独立区室最终出现,使得系统成为自由生命体。这种方法实际上类似于生命起源的过程。通过本文,我们旨在激发合成生物学家和实验科学家考虑更理论的角度,并促进生命起源研究者与人工合成生命研究者之间的交流。
我所不能创造的,我也无法理解。
——写在Richard Feynman的黑板上
1. 引言
在生命科学中,如何在实验室中创造生命是最引人入胜的问题之一。关于生命起源的现代研究始于20世纪40年代的Miller-Urey实验[1],该研究清楚地表明,无机物可以转化为有机物,生命与非生命之间没有根本的障碍。自那时以来,人们意识到创造一个简单的人造生命——即一个在地球上从未存在过的简单生命形式——至少是可能的。如果强调其“人造”属性,则这种生命可能被称为人造生命、合成生命或湿式人工生命 (术语“人工生命”通常特指关于生命基本过程的计算机研究);如果强调其“细胞”属性,则可能被称为人造细胞、合成细胞、最小细胞或原细胞。在本文中,我们仅使用宽泛的术语“人造生命”,不做特别区分。一方面,有些人试图根据地球上现有的生命形式创造人造生命,试图将生命系统的复杂性 (从现有的复杂生命中) 减少到最小水平,以便揭示生命的最基本和必要条件。因此,这通常被称为自上而下的方法。另一方面,也有人试图找到定义生命的条件,并创建满足条件的系统,即将整合定义明确的组件集成、组合、拼装来实现。由于这种方法试图找到生命的最基本形式,因此通常被称为自下而上的方法。
由于合成生物学的迅速发展,自上而下的方法对人造生命做出了实质性贡献。事实上,人们已经成功地在实验室中“创造”了生命,例如Venter实验室及徐灿等的工作[2, 3]。尽管如此,这种方法所取得的成就并不是从非生命中创造生命,而是从生命中创造生命。它使用现有生命可以产生的物质来组装一个活细胞。尽管这些研究都可被称为杰出的成就,并有助于我们理解生命的基本属性,但这种方法依赖现有生命的存在。此外,该方法所依赖的生命形式是自生命起源以来数亿年演化的产物。抹去生命的进化历史就有可能得到关于生命性质的误导性描述。当前生命形式拥有的一些功能可能并非最简单和最基本的生命所必需的。
自下而上的方法试图找到生命的蓝图和定义生命的条件。学者们早已发现,寻找单独的充分且必要的用于定义生命的特征几乎是不可能的[4, 5]。我们需要的可能不是定义,而是关于生命本质特性的理论[6]。然而,通常从地球上的生命中观察到的属性构成了科学家研究的起点。一般来说,代谢/自复制/自维持,信息和区室/边界是最常被引用的属性,人们认为这些属性构成了生命的本质。因此,我们在表1(不完整但具有代表性的表格)中分类了各种有关生命属性的理论:
表1. 关于生命属性的各种理论。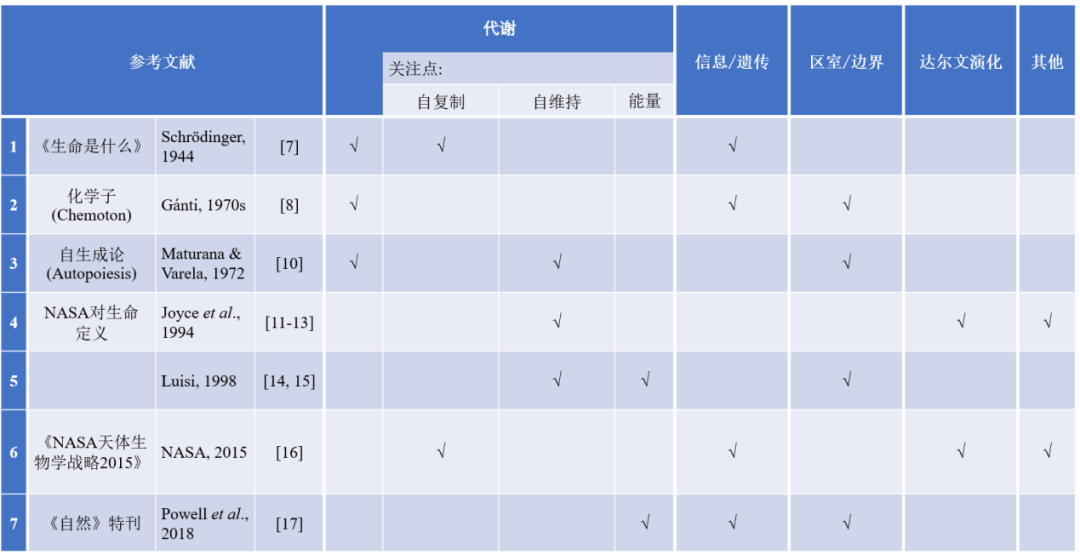
*“自复制”指的是生命系统能够复制自身的能力。“自维持”是指生命系统再生其所有组分、在时间上保持其存在和功能的能力(事实上,如果一个系统能够自复制,则在其他适当条件下它必须是自维持的,反之亦然,详见第3.1节)。“能量”指的是获得和利用能量以供代谢过程使用的能力。这三个特性都属于“代谢”。“信息/遗传”指的是拥有编码信息并可以传递给后代的遗传物质。“区室/边界”指的是拥有将系统与其环境分隔开来并允许其以个体方式存在的物理边界。“达尔文演化”指的是能够随着时间的推移而发生可遗传的变化,从而导致适应性增加,并增加生存和繁殖的机会的能力。最后,在第4行,“其他”特性指“化学系统”;在第6行,“其他”特性指“聚合物”(详见正文中的解释)。
1. 物理学家Erwin Schrödinger在他的著名书籍《生命是什么》中,从热力学角度阐述了生命的概念。生命以负熵为食,能够进行新陈代谢和自复制[7]。“熵”在热力学中是对一个体系混乱程度的度量,越有序,熵越低;“以负熵为食”则是一个隐喻:生命要以某种方式从外界获得“秩序”,即某种信息。
2.20世纪70年代,Tibor Gánti提出生命系统必须由三个子系统构成:有一种“细胞膜”来容纳细胞物质;能进行“新陈代谢”从而实现营养物质的补充及更新能力;并具有自己的“遗传物质”[8]。后两点跟Schrödinger的想法有重合,而第一点则强调了“细胞膜” (即边界) 的重要性:由于热力学第二定律的约束,任意封闭系统的熵都会自发增加,但生命的组织程度很高,因此,需要一个边界来将生命系统与非生命系统分开[9]。这个概念被称为“化学子(Chemoton)”。
3. 自创生论(Autopoiesis)由Humberto Maturana和Francisco Varela提出[10]。由于他们不满足于分子生物学这个主流的研究生命系统的方法,即认为基因信息是生命的本质。他们根据细胞这一生命基本单位的组织特性,提出了自创生论(Autopoiesis)。这个理论的主要关注点是个体的独立性以及与环境的互动,而不是信息。
4. 美国宇航局(NASA)对生命有一个定义,生命是一个可自维持的化学系统,并且能够进行达尔文演化[11-13]。这个定义通常归功于Gerald Joyce,他总结了NASA于1994年组成的一个委员会的讨论,旨在给出生命的定义。这个定义虽然很有用,但是说得比较笼统,比如什么是自维持化学系统、什么是达尔文演化,可能需要进一步澄清。这个定义特别强调了达尔文演化,所以我们在表1中也只强调了这一点。虽然我们理解许多研究人员认为的达尔文演化可能意味着自复制、信息/遗传(以及突变/变异),但我们没有标注这些含义。
5. Pier Luigi Luisi指出,生命应该是一个由半渗透的腔室结构所定义的系统,且该系统能够利用自身过程来转换外部能量和营养物质,从而自维持。其中强调了边界、能量、新陈代谢过程,特别是自维持[14, 15]。
6. 美国宇航局(NASA)发布的一份长达250页的《NASA星际生物学战略2015》指出,聚合物的出现对于生命的产生至关重要,这些聚合物必须能够复制、存储遗传信息,并能够进行达尔文演化[16]。这强调了特定类型大分子聚合物的重要性。
7. 《自然》杂志的专题”自下而上的生物学”总结认为,从头创造生命的三要素应该包括:区室化,能量和信息分子[17]。换句话说,这三个因素被认为是生命出现的必要条件。
尽管上述对生命属性的描述并不明显一致,但也并不相互排斥,而是各有侧重。原则上,如果我们可以将这些要素以某种方式组合起来,就可以在实验室创造出生命。这也正是一条努力的主流方向。一方面,可以从较复杂、成熟的生命形式中借鉴这些要素,通过某些方式降低它们的复杂度,以求达到一个最小细胞,这也就是自上而下(top-down)方法。另一方面,可以在实验室合成和创造这些要素,再以某种方式结合在一起,这也就是所谓的自下而上(bottom-up)方法(参见图1)。在第2.1节中,我们将讨论自上而下的方法,它实际上已经以某种方式取得了一些成功。虽然自下而上的方法没有真正实现创造人造生命的最终目标,但它已经在表1中显示的每个因素方面都取得了实质性的进展。我们将在第2.2节中分别详细讨论它们。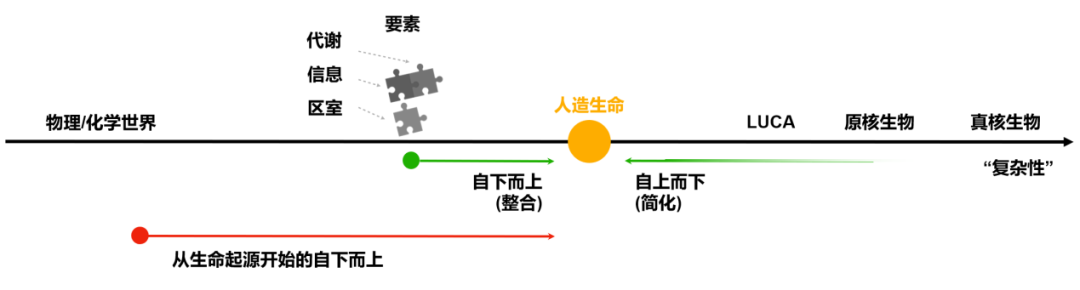 图1. “人造生命”的研究蓝图
图1. “人造生命”的研究蓝图
事实上,我们可以通过认识和回顾过去几十年在一个强相关的领域——“生命起源”领域所收集的丰富结果 (在这个领域中,我们关注使生命出现成为可能的基础机制,但并不关注生命在地球上究竟何时何地开始的历史问题) ,进一步推进自下而上的方法。如果这些概念、理论和工具可以被利用并用于人造生命的任务,这种方法仍然可以被称为“自下而上”的方法,只是我们将“底部”向下推到了非生命(即物理和化学)的世界,如图1中的红色箭头“从生命起源开始的自下而上”。实际上,生命起源研究和合成生物学这两个研究领域之间的交流并不多,可能是因为前者中提出的理论/概念与后者在尝试创造人造生命时必须面对的化学/生物世界之间存在着巨大的鸿沟。但由于合成生物学和相关理论工作的迅速发展,这个差距正在缩小。在第3节中,我们将讨论在生命起源领域的深刻理论和假设,即自催化集及其相关理论,也将讨论如果这个概念被用于人造生命时所面临的问题(第3.1节)。之后,我们将试图对这些问题给出我们当前的答案(第3.2节和第3.3节)。最后,在第4节中,我们将进行讨论和总结。
2. 已有的努力:自上而下和自下而上的方法
2.1 自上而下:简化复杂度
下面是几个比较著名的以自上而下(top-down)方式成功实现的人造生命的例子,它们以现有生命为蓝本,通过降低其复杂度的方法试图找到最小的同时仍能维持最基本的生命活动的系统。
- “黑进(Hack) ”细胞:从JCVI-syn1.0 到 JCVI-syn3A
2010年,Venter团队从头合成了支原体(Mycoplasma mycoides)基因组,将其植入一个事先除去了基因组的蕈状支原体细胞中。经过一段时间的生长、分裂,会出现正常细胞和只含有人造基因组的细胞,随后借助抗生素杀死正常细胞,便筛选出了“人造生命”,并被命名为JCVI-syn1.0或Synthia [18]。这个生命体可以独立地进行自复制。JCVI-syn1.0 的基因组是模仿现存基因组进行合成的。这个合成细胞的基因组含有超过1百万个碱基对,并不是最小基因组,显然有很多冗余或非必须基因。为了完成终极目标:设计最小基因组以找到对维持生命必须的基因组合。Venter团队开始了漫长的“设计-构建-测试”循环。终于在2016年,宣布合成了 JCVI-syn3.0 (JCVI-syn2.0 是一个中间版本)。这个生命体的基因组含有531,000个碱基对,473个基因,这是人类已知的可以独立复制的最小生命[2, 19]。然而,JCVI-syn3.0具有许多形态变异,例如丝状和囊泡形态,这些都是意料之外的,并且看起来是“异常”的。为了解决这个问题,2021年,Pelletier等设计了一种微流控恒温装置。这一装置可以协调微小细胞的运动,使其在光学显微镜下任意移动,并保持正常的生命活动。通过这一技术,研究人员对 5 年前创建的 JCVI-syn3.0 细胞进行了观察。在向 JCVI-syn3.0 细胞中加入了19个基因后,合成的新细胞 JCVI-syn3A,可以实现正常的分裂增殖[20]。但这添加的19个基因,仅有2个基因的功能是明确的,剩余基因的作用依旧不清楚。因此,通过了解每个基因的功能,以便开发出一个完整的人造细胞的愿景还需要进一步努力。
- “黑进(Hack) ”细胞:瞄准复杂真核生物
自从Venter的原核细胞成功合成,各国研究人员也将目光投向了较为复杂的真核生物。2018年,覃重军团队从含有16条染色体的单倍体酿酒酵母细胞出发,通过CRISPR-Cas9技术敲除酵母染色体多余的着丝粒和端粒,再通过连续地端对端染色体融合,历经4年,成功将其融合为1条。将染色体“16合1”后的酿酒酵母菌株命名为SY14。这是世界首例仅含单条染色体的真核细胞[21]。该项工作表明,天然复杂的生命体系可以通过人工干预而大幅改变,同时仍然可以执行复杂的生命功能。
- 从活细胞中重新组装组件
2022年,研究者通过在人工合成的囊泡中加入活的细菌,接着使用细胞壁水解酶对细菌进行原位裂解,再加入组蛋白和羧甲基葡聚糖提供营养和能量,在孵育了48小时之后,该细胞具有了正常的生命行为[3]。这相当于让活细胞分解,然后在囊泡中自动重组。这种将原核细胞作为结构和功能原料库,构建具有类真核细胞基本结构和功能的人工细胞构建方法,开辟了从“生命”到“生命”的另一种思路,为合成生命提供了更多的可能。但是,这个“细胞”还不是一个可自复制的细胞,这可能是由于人工合成囊泡的问题,说明细胞边界不仅仅用于隔离其与环境,也对于其分裂繁殖有重要作用,人们目前对这一部分的理解还很有限。
2.2 自下而上:整合定义明确的组件
2.2.1 代谢视角
早在前分子生物学时代,生命起源于代谢[22]还是遗传[23]的巨大辩论就曾伴随冷战两大阵营的对峙被公众熟知[24]。分子生物学时代,以Miller-Urey实验为开端的生命早期化学代谢进路与以DNA双螺旋发现及中心法则为核心的遗传进路又一次陷入数十年的争议,至今尚未平息。然而,由于双方关注的重点不同,且原始汤与RNA世界之间的巨大鸿沟难以填补,因此二者的分歧被Lazcano称为是“聋人间的对话” [24]。同时,如果我们试图把“代谢”和“遗传/信息”的概念完全分离,也会面临巨大的困难,因为二者是深度相连的。因此,我们决定放弃这种会导致模糊不清的分类方法,将遵循Miller-Urey实验(即从简单成分中形成复杂的化学反应网络)的研究线路称为“代谢线”,将从双螺旋结构和中心法则角度出发的研究线路称为“遗传/信息线”。
代谢(Metabolism),原本是讲生命体中各种物质的流入流出过程。但在前生命阶段,代谢的具体含义在不同语境下有些区别。目前较为公认的“代谢优先(motabolic-first)” 解释倾向于这个观点,即原始地球上出现的自催化“代谢”循环是遗传系统出现的一个基本先决条件。这一论断试图将物理学中根深蒂固的趋势外推到生物学中,并期望找到包括自组织、自组装在内的,能够解释生命等许多复杂系统的包容性规律[24]。
“代谢优先”的主要的理论包括 Kauffman的自催化集理论(autocatalytic sets)(第3节详述)[25]、Ganti的“化学子(Chemoton)”[8]、Maturana与Varela 的自创生(autopoiesis)理论[10]等。代谢优先理论包含以下一些要素的部分:1) 无酶的多步化学的自催化集;2) 能够固定CO2中的碳;3) 必不可少由封闭的膜作为个体边界;4) 能表现出增殖和形成复杂网络的能力[24]。
一直以来对“代谢优先”理论最大的攻击在于,缺乏实验证明代谢途径(自催化循环或网络)是化学可执行的[26],也没有证据表明这类系统确实可以进行复制、突变和进化[24]。事实上,有一些初步的实验证据支持“代谢为先”,包括:1) 实验验证了一系列天然存在的自催化反应。如醛缩糖反应、即甲醛聚合产生包括核糖在内的糖的复杂混合物[27],以及甲醛存在下由HCN单价生成HCN四聚体[28]等反应。2) 反式柠檬酸循环亦称为还原性三羧酸循环(rTCA)作为原始代谢的核心可以在非酶条件下成立,并可以连续增加的复杂性层级[29]。3) 实验证明,可以由CO和硫化氢为起点,在硫化铁和硫化镍作为催化剂的条件下,获得早期蛋白质[30-32]。此外,近年来的多项进展使得“代谢为先”相关领域热度越来越高,上述挑战也逐渐面临瓦解。Lazcano等提出了代谢途径的半酶起源假说,提出简单代谢样途径的起源产生了原始生物体所需的关键组分[33],这些途径最初由非酶或半酶催化,后来作为核酶介导的和基于蛋白质的酶代谢途径进行了微调[34]。Wołos等人利用一款名为Allchemy的软件进行的模拟,他们从水、氮气、硫化氢、氨气、氰化氢、甲烷这6种物质作为反应底物进行模拟,在第7代后发生了催化剂、可产生126%循环效率的自催化反应系统并发现了表面活性剂的涌现,后者可能成为构成边界膜的前体物质,他们还在化学实验中验证了其中一部分反应过程[35]。以上这些研究均部分回应了Orgel等关于自催化网络是否可以由真实的化学反应实现,以及反应网络可否变异、演化并被自然选择的质疑,而可能实现边界(膜)的脂类物质可由代谢反应网络实现也一再被证实。
2.2.2 遗传视角
自从生命过程由一系列自催化反应组成的观点被提出后,Troland认为生命当中应存在“具有遗传效力,同时可催化反应的分子(genetic enzymes)” [36],而这时RNA还尚未被发现。随后科学家均不同程度地将RNA与生命起源联系起来[37-39]。两类核酶的发现促使了RNA世界假说的提出。RNA世界假说是指地球早期存在一系列RNA,凭借自复制和催化能力衍生出其他重要生命元素[40]。中心法则中的DNA和蛋白质可能是早期RNA生命的后期发明。尽管最原始形式的生命不一定基于RNA,但是现在普遍认为地球上的生命起源于RNA世界[41]。
RNA世界在现有生物系统中的残留包括许多核苷酸衍生的辅酶、可自我加工的核酶、代谢物结合核糖体,以及由RNA核苷酸或其前体构成的现代信号分子和第二信使分子(如腺苷环磷酸(cAMP)、鸟苷四磷酸(ppGpp)、5-氨基-4-咪唑羧酰胺核苷酸三磷酸(ZTP)和环二核苷酸)等[42]。最为人熟知的便是RNA与蛋白联合构成的核糖体。核糖体的结构表明核糖体是一种核酶,因为在催化肽键形成的活性位点(18 Å以内的区域)中没有氨基酸侧链[43]。除此之外,新发现的氨基酰化能力也为RNA世界增加了一份可行性[44,45]。另一方面,现存基因组中也有不少RNA世界的“痕迹”。例如,在最后共同祖先(LUCA)中发现了大量RNA代谢相关的蛋白,包括用于RNA聚合,RNA修饰以及降解等的酶[46]。同样,细菌核心蛋白质组(core bacterial proteome)也是与RNA代谢、信息传递等密切相关[47]。
总的来说,RNA世界假说是一种描述地球早期生命可能存在的模型,其中RNA在生命进化过程中发挥了关键作用。虽然这个假说并不能完全解释地球上的生命起源,但它提供了一种有趣的解释,并且得到了许多独立的证据支持。RNA世界假说在其刚刚提出时就面临了两个主要问题:RNA能否真的能够独立执行所有生命功能,以及其是否可能在早期地球上形成。
对于第一个问题,在实验室合成了较短的RNA后,研究人员发现了具有催化自复制能力的RNA分子。其中一种核酶,能够通过模板的编码进行RNA复制中的聚合步骤[48],另一种则是可以将较小的RNA序列连接在一起,从而完成自复制。随后科研人员便发现了能够自维持复制的RNA系统,其中包括两个催化彼此合成的核酶,产物的复制周期约为1小时,并且在实验条件下受到自然选择的影响[49]。其中RNA可以通过自身编码的RNA复制酶所复制,这机制使得RNA水平的达尔文演化成为可能。在这个研究中,作为宿主RNA和寄生物的RNA通过240轮的一组转移实验,实现并维持了多重复制子网络[50]。
上述实验均未考虑RNA在地球上的起源问题(即RNA如何从无到有),即第二个问题。而很多基于解决核酸前生命合成的生命起源假说恰恰认为这是最重要的一步。对于合成核酸,诸多合成化学家青睐从氢氰酸(HCN)出发[51]。作为高能的生命前体分子,氢氰酸的生成条件很容易实现[52],并且可以以其为基础在地球条件下合成腺嘌呤[53]。此外,氢氰酸还被发现与还原性三羧酸循环有关,研究人员也因此提出了氢氰酸存在的自催化的潜力[54]。在此基础上,Saladino等人整理了氢氰酸的近亲,即甲酰胺的相关实验,论证从头合成RNA链以及代谢网络以及该图景的可行性[55, 56]。核酸的合成涉及碱基、五碳糖的合成与磷酸化、核糖核苷酸的聚合等多个复杂且条件不一的步骤[57],在前生命条件下很难保持这样理想的路径。
2.2.3 区室/边界视角
现有的研究中把一种在水环境中通过自组织形成的单层胶束或双层囊泡称为原细胞(protocell) [58, 59]。Chen和Szostak进一步证明,对于原细胞,其简单的物理化学特性就可以产生一些基本的细胞行为,包括原始形式的差异繁殖竞争和能量储存。膜与包裹物之间的这种合作式相互作用可以大大简化从简单复制分子到真正细胞的过渡[60]。在最近的一项研究中,科研人员通过探讨脂质如何通过内源催化过程进行化学修饰,从而引导了从胶束到囊泡以及从单体到生物聚合物的转化,扩展了人们对原细胞复合物化的理解[61]。
事实上,边界或区室的意义至少有以下几个方面。首先,区室/边界是定义个体的关键。Ganti在《化学子(Chemoton)理论》一书中指出,自催化化学反应循环可以带来目标物质在溶液中的累积,却不能产生“个体”,只有当催化反应循环被脂质微球包裹后,在化学计量方程式中,才可以以个体的增加来取代分子数的增加[8]。其次,拥有边界是热力学第二定律的要求,因为封闭系统的熵必然增加,而生命(具有组织程度高的特征)作为开放系统要对抗这种倾向,必须与环境隔开[7, 62]。
第三,作为边界的半透膜允许膜内的新陈代谢和小分子交换,并且防止大物质穿过膜,这样可以选择性地阻止和渗透物质,提高反应物浓度,可使得一些反应成为可能,或显著增加反应速率[63]。正如在Damer和Deamer关于生命起源的理论中,认为脂质在雨水形成的池塘里形成细胞膜的第一个阶段,此时的膜上有很多孔,这些孔会对进出的分子进行选择[64]。近期的工作指出合成细胞建模的核心原则可能是半渗透区室化的概念,因为它能够选择性地扩散,保留和释放结构组分、化学能、酶底物和信息输入/输出[65]。通过这种渗透化,细胞可以与其他细胞相互交流,这是生命系统的一个标志[66]。
最后,区室本身可能是“信息”首先出现的地方。最早的信息可能来自自维持的化学组装内分子的类型和数量,称为“组成信息(compositional genetics)”[67]。此后,更丰富的遗传信息可以涌现出来,如RNA或DNA等聚合物的出现[58]。这个猜想来自于以色列科学家Lancet及其合作者Segré等的“脂质世界假说(lipid world hypothesis)”,他们认为第一个能够自复制的有机体很可能是类脂质的,而且是单层胶束而非如现代生命般的双层磷脂层。因为单层胶束具有许多好处,包括对组成变化的更高敏感度,有利于形成自催化相互作用,化学多样性高,适应各种不同大小、几何形状和功能团的化学物质,增强了扩散相互作用等[61, 69, 70]。另外,DNA扩增被证明可以诱导脂质囊泡的生长和分裂,再次将信息物质的复制与区室/边界的复制联系起来[71]。
3. 从生命起源的研究中学习:建立复杂性
除了第2部分所说的路线(即自上而下通过简化现有的生命系统来降低其复杂性,而自下而上则通过整合定义明确的组件来构建系统),还有另外一条可能的进化路线:由于生命是从纯粹现代科学主流认为无生命的、物理化学系统演化而来,“模拟”其从无到有的过程是一条自然的、原始的路线:即从更加抽象的生命定义出发,构造极其简单的系统,这个系统可能一开始不满足生命的所有要素,是一个无生命的简单物理、化学系统,然后由此为起点构造并演化到“生命系统”。这种逻辑最早可以追溯到Miller-Uery实验,多种无机小分子(“非生命物质”)通过加热、电击、冷凝等方式生成了简单的氨基酸(“有生命物质”)。虽然其中的机制不是很清楚,但似乎我们只需要准备合适的物质(它们并不是很复杂)和合适的条件(它们并不是很复杂和微妙),转化就会自动发生。然后就有了这样的推测,即从这些小分子中导致生命的出现的“神奇事件”很可能发生,就像数十亿年前在地球上实际发生的那样。
虽然上述逻辑似乎很直接,但是应该添加怎样的物质、怎样的条件却是十分困难的事:这其中有一部分人走向了自下而上整合定义明确组件的道路,如上文所述;另一部分人则不再强调湿实验,转而从非化学系统入手,寻求自复制等生命的基本特性是怎么涌现出来的,并试图找到底层的数学规律。这其中有一部分分支发展成了现在称为“人工生命(artificial life)”(严格来讲,应该叫“软人工生命(soft artificial life)”)的领域,催生了诸如进化算法、人工生态学等工作和成果(跟本文的主题渐行渐远,其发展历程见方框1);另外还有一部分人仍然在研究生命的底层数学逻辑,其中最重要的工作是由Kauffman开启的自催化集(autocatalytic sets)及后续的相关研究为代表。
这部分研究跟人造生命非常相关,其中诸多成功可以在人造生命领域进行借鉴 (虽然距离根据这些理论创造出原细胞 (protocell) ,还存在巨大的的鸿沟),这条路我们姑且称为从生命起源开始的自下而上(bottom-up from the origin of life)进化,这条道路对于实验生物学家与合成生物学家来说,还是相对陌生的。相对于早期简单小分子的合成,我们可以从如何构建与耦合早期化学反应循环成为网络开始讨论。本章旨在从“自催化集”理论出发,介绍这条道路的优势以及其在创造人造生命中所面临的一些困难,比如关于边界和演化,之后对于这些困难尝试提出我们的2个解决方案。
方框1、“软人工生命”研究领域的简要历史(通常简称为“ALife”)
20世纪40年代末,John von Neumann发表了题为“自动机的一般和逻辑理论”的演讲。他将“自动机”定义为:通过结合环境信息和自身编程,可逻辑化地逐步执行行为动作的任何机器,并表示,最终人们会发现自然生物也遵循着类似的简单规则[72]。这可能是最早的机器上的“人工生命”的概念,尽管还没有这个名字。
随后,很多科学家转向这个领域,有很多有启发性的工作。20世纪60年代,剑桥大学教授John Horton Conway发明了最著名的元胞自动机,他称之为“生命游戏”[73]。康威生命游戏的世界是一个二维平面,由一个网格构成,每个网格称为一个细胞,每个细胞有两种状态,存活或死亡,每个细胞会根据周围八个细胞的状态来更新细胞的状态。就是这样一个非常简单的、根据局域规则更新、完全确定的系统展现出了极其丰富的动力学和涌现行为,而且被证明是图灵完备的,意味着它可以模拟任何其他图灵机。在70/80年代掀起了一股研究热。Christopher Gale Langton将John von Neumann最初的29个状态怪物简化为只有8个状态 (仍然基于元胞自动机),1979年10月,他仅用一台Apple II型台式电脑就成功地创造出了第一台能够自复制的计算机有机体[74]。1982年,计算机科学家Stephen Wolfram探索和分类了一维元胞自动机所显示的复杂性类型,并展示了它们如何应用于自然现象,如贝壳的模式和植物生长的性质,发表在一本著名的书籍《一种新科学》[75]。目前,元胞自动机模型不仅是展示涌现行为的很好的例子,也仍然在各个研究领域发挥着重要作用,比如模拟伤口愈合[76],模拟量子晶格气体[77],量子点细胞自动机[78]等。
1987年,美国圣塔菲研究所的科学家Christopher Langton正式提出了“人工生命”这一概念,它的英文为 Artificial Life (简称ALife)。他所给的定义可简单概括为:“研究在虚拟环境 (如计算机模拟) 中表现出生命特征的人工系统,以理解定义此类系统的基础信息处理机制。一般来说,ALife领域中发展的方法和技术包括元胞自动机 (如上所述),基于代理的 (或基于程序的) 模型,进化算法等。随着这个术语的提出和计算机技术的快速发展,ALife得到了重要的拓展和发展。值得一提的是,ALife不仅对科学有影响,而且对流行文化也有影响,包括许多视频游戏都遵循了ALife的思想和概念,如Creatures (1996年[80]),Spore (2008年),Darwinbots (2014年) 和Species (2018年)。
关于代理模型,Craig Reynolds于1987年创建了一个程序,成功地模拟了一群鸟的行为,其使用了三个简单的本地更新规则[81],从而开启了随后在群体动物行为方面的一系列研究,如著名的Viscek模型[82]。1991年,Tierra应运而生,用于模拟生物进化。它模拟了生物之间的竞争和合作以及它们适应环境变化的能力,每个生物都被称为“代理”,由计算机程序表示[83]。之后,Tierra启发了Charles Ofria、Chris Adami等人开发Avida,该程序目前仍在积极开发中。Avida中的代理能够通过执行基本的计算任务,使用其“基因组”获取资源并进行自复制,从而实现代理的基因组传递到下一代。通过这种机制,Avida能够模拟进化过程[84]。
进化算法是人工生命领域开发的一种有用和实用的工具,可为优化和搜索问题提供高质量的解决方案[85]。进化算法的基本思想是通过一组仿自然进化过程的规则(如选择、交叉和变异)在一系列世代中进化出一种解决方案(例如编码为整数向量)。其中最著名的进化算法之一是遗传算法,由John Holland首次提出。遗传算法已广泛应用于各种实际领域,包括工业设计、电路设计、生产调度等[86, 87]。
近年来,在人工生命领域,开源科学项目OpenWorm被开发出来,最终目标是建模和模拟秀丽线虫(Caenorhabditis elegans)的所有959个细胞及其全部行为。如果成功,这将是人们第一次完整地表征整个生物体的行为 [88]。另一个名为Neurokernel的项目,基于类似的思想,旨在建立一个开源平台,模拟果蝇(Drosophila melanogaster)的整个大脑功能。它包括一组库和工具,方便研究人员在多处理器系统上构建和执行大脑模型 [89]。
3.1 从生命起源开始的自下而上:自催化集
上文提到的多篇文献都主张代谢 (Metabolism) 是要实现人造生命的一个必要条件 (即使不是唯一必要组成),当然亦是生命本身的必要条件。从现代意义上讲,代谢(Metabolism) 是指生命有机体或细胞中用以维持生命的化学反应的总称;但是在生命起源之初(亦即我们构建人造生命的过程中),此时尚无生命有机体或细胞的概念,这时的代谢(Metabolism)只是指这个系统中所有化学反应的总称,这些反应共同发生作用,以实现类似生物体中繁殖(例如自复制,作为生命最重要的特征之一)等功能[90, 91]。所以,最关键的问题是,这个系统中需要哪些化学反应耦合在一起、怎样耦合在一起才能使得系统整体上实现类似生物体中自复制等功能。
1993年,Kauffman提出,如果一个化学体系中,任意一个反应的生成物都作为该系统中其他某个反应的催化剂(即一个闭合的系统),那么该系统中所有物质的量就能指数增加(如果原料充足的话),从系统层面上看,这就实现了自复制。他把这个体系称为自催化集(autocatalytic sets),并认为生命最初是作为自催化化学网络出现的[25, 95]。该理论经过Mike Steel和Hordijk等人的发展,成为一个数学上更为完备的自我维持化学反应网络(RAF)理论[96]。
更进一步的研究表明,自复制的条件并不像RAF理论中所要求的,系统中的每个反应都是催化反应,而是更为宽松[92],因为任何催化反应都可以在数学上被重写为一组耦合的非催化反应。在这里,我们总结了宽松的自复制条件:如果在一个化学反应系统中,
1) 对于每个反应,至少有一种反应物来自于其他反应的产物(称为自驱动(self-driven)的标准);
2) 存在某些类型的中间分子,它们出现在反应物的数量比产物的数量少(称为过量生产(overproduction)的标准);
3) 不存在任何类型的中间分子,其在反应物一侧出现的次数大于产物一侧出现的次数(称为无过量消耗(no-overtake)标准)。
如果一个系统满足前两个条件(自驱动(self-driven)和过量生产(overproduction)),则很有可能是自复制的(必要条件);如果满足所有3个条件(即在上述基础上满足无过量消耗(no-overtake))那么一定是自复制的。下面给出[92]中的几个例子(参考文献[92, 97]的补充信息还有其他例子):
图2. 能够自复制的化学反应系统的例子。该图经授权转载自文献[92],版权所有 © 2018年Liu和Sumpter。(a) 醛缩糖反应的简单表示。(b) 通过反应方程式表示的醛缩糖反应。(c) 另一个想象中的自复制反应网络。我们可以检查所有这些系统都满足上述三个条件。
图2a(和2b)中的系统是甲醛缩合反应的简单表示,是自复制反应网络的最简单示例,其中 代表甲醛,
代表甲醛, 代表甘油醛,
代表甘油醛,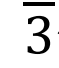 代表丙醛,
代表丙醛,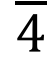 代表四糖。首先,第一反应中的
代表四糖。首先,第一反应中的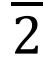 来自第三反应的产物,第二反应中的
来自第三反应的产物,第二反应中的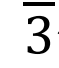 来自第一反应的产物,第三反应中的
来自第一反应的产物,第三反应中的 来自第二反应的产物,因此满足自驱动 (self-driven) 的标准。其次,
来自第二反应的产物,因此满足自驱动 (self-driven) 的标准。其次, 在产物侧出现了两次,在反应物侧出现了一次,因此满足过量生产 (overproduction) 的标准。最后,没有分子在反应物侧出现的次数超过了产物侧,因此满足无过量消耗 (no-overtake) 的标准。同样地,我们可以验证图2c中的系统也是自复制的。
在产物侧出现了两次,在反应物侧出现了一次,因此满足过量生产 (overproduction) 的标准。最后,没有分子在反应物侧出现的次数超过了产物侧,因此满足无过量消耗 (no-overtake) 的标准。同样地,我们可以验证图2c中的系统也是自复制的。
自复制从理论上来说是被很好定义的,但是实验上往往很难确定是否存在。因为这个定义依赖于对系统中所有化学反应的全面理解,而完全确定所有相关的反应通常很困难,甚至是不可能的[98]。所以我们在文献[98]中提出了一个能够在实验上判断是否自复制的标准(事实上,自复制和自维持的系统在化学网络拓扑的层面上是相同的,在不同的外界条件下是可以相互转化的,所以并不用特别纠结到底这个系统是自维持还是自复制的):如果一个化学系统在一个连续搅拌的槽反应器(CSTR)中,并受到一个输入流f的作用,则当且仅当满足以下两个标准时,它是自维持的(参见表2):在瞬态期之后,(1) 对于非零初始条件ξ,输出流f’与输入流f不同,(2) 对于零初始条件ξ0,输出流f’与输入流f相同。这里,“零初始条件”意味着在实验开始时反应器中没有任何物质 (否则是“非零初始条件”),详见文献[98]。根据这些标准,要实验性地确定系统是否是自维持的,我们只需要比较CSTR的输出流f’与输入流f即可。
表2. 化学系统成为自维持的条件。这里,f’代表输出流,f代表输入流。ξ表示非零初始条件,而ξ0表示零初始条件。请参见参考文献[98]以获取详细信息。经文献[98]授权转载。版权所有© 2020, The Author(s)。
除了自催化集和RAF理论之外,还有两个类似的概念。1994年Fontana提出一个类似的概念,称其为“λ演算法”,其核心思想是把一个分子看作是一个数学函数,把它与另一个分子碰撞时的相互作用看作是函数操作。把它们看作是“化学定律”的逻辑对应物。其化学的构造特征是分子的碰撞产生特定的新分子[99]。该理论在数学上更普适,更有野心,当然用起来也不容易;但在判定自复制的这件事上,跟自催化集理论和RAF理论得出的结论是类似的。另一个理论是由Dittrich和Banzhaf等人提出的化学组织理论(COT),作为研究生命起源的另一种形式框架[100]。它基于集合论,并将由一组封闭且自持续的组分(而不是像RAF理论中的网络一样)定义为化学组织。在COT中,它也不要求每个反应都是催化反应[101]。
对于上述的理论,实验上有很多证据。比如,在实验上确实观测到了比如大肠杆菌(E. coli)的代谢网络就是一个巨大的自催化集[102]。Semenov等人通过使用小的有机分子(一个硫酯,一个3-氨基硫醇的硫化物,一个动力学上快速和不可逆的硫醇抑制剂,以及一个缓慢和不可逆的硫醇抑制剂)建立了一个具有自催化、双稳态和振荡行为的有机反应网络,表明与细胞功能相关的复杂的调节动力学可能在细胞生命之前就已经自发产生了[103]。Otto教授发现了一种新的自复制机制。这种机制中存在一种含肽的小分子,称为基础块。基础块能够连接在一起形成3元环、4元环和6元环,其中6元环的产生是依赖于3元环和4元环的。6元环能够堆叠在一起,断裂后的残余部分可以继续堆叠。这样就形成了一个不依赖于外界的自复制系统[104, 105]。Nanda等人分析了β-片状肽网络中复杂的反应网络,发现该网络不仅具有结构稳定性、自催化和催化性,甚至具有可进化性、纠错性和手性选择性[106]。最近,Peng等人表明两个大规模反应网络具有分层组织的自催化特性[107]:一个包括与生命起源相关的所有反应,在KEGG反应数据库[108]中有记录,另一个包括放射性和地球化学反应[109]。
总之,对于人造生命这个任务,从自催化集理论、RAF理论和相关工作中可以得到的结论是:我们可以有目的地设计符合这些数学标准的系统,从而自动地获得“代谢(Metabolism)”的一个主要特征——自复制。这是实现生命体系的第一步(实际上,如前所述,已经有成功的案例[44, 45])。然而,虽然我们可以设计一定的化学反应网络,使某些分子能够复制,但我们不能称这种类型的分子或整个反应网络为生命,因为它面临至少两个主要问题:(1) 如何定义这些系统中的边界(或等效地,隔离系统与其环境的能力),(2) 能够具备进化的能力。在下面的两个小节中,我们将讨论这两个问题并试图提出潜在的解决方案。
3.2 关于区室/边界:碱性热液系统
自催化集理论 (或RAF理论) 并不强调区室或边界的概念。它认为组成边界的分子也应该通过这种自催化的化学反应网络而出现,跟能够复制的其他分子一样,所以没有必要单独讨论。然而在实际操作中,应该怎样设计这种自复制的边界系统,以及怎样将其耦合在整个“原细胞(protocell)”的代谢网络中,仍然是一个开放的问题。本节中,我们从另一个角度来探讨这个问题:最开始的边界是否可能仅仅是物理世界提供的,而后由于细胞慢慢“发明”了边界分子使得它能够像现代细胞一样自由移动。这就必须要从Günter Wächtershäuser的铁-硫世界到William Martin和Michael Russell的碱性热液喷口理论,本节我们首先回顾这些理论。
在1980年,Günter Wächtershäuser提出了“铁-硫世界”理论,这是一个关于前生物化学途径进化的理论,被视为生命进化的起点:生命可能首先形成于铁硫矿物表面[30]。该理论系统地将当前的生物化学过程追溯到原始反应,原始反应提供了从简单的气体化合物合成有机构件的替代途径。与经典的Miller实验依赖外部能量来源(模拟闪电、紫外线照射)不同,“Wächtershäuser系统”自带内置能量来源:铁的硫化物(如黄铁矿)和其他矿物[30]。这些金属硫化物的氧化还原反应所释放的能量可用于有机分子的合成,这种系统可能已经演化成自催化集,构成自复制、代谢活跃的实体,早于今天已知的生命形式[30, 110]。这个理论的前提是铁硫化物表面是带正电的,这可能通过反应物的吸附和正确排列来促进聚合[30]。
这个理论涵盖了几个关键点。首先,它提出生命起源的特定地点可能是在深海高压高温的火山热液喷口,最初被认为是黑烟囱[111, 112]。这些热液喷口不仅提供了源源不断的原材料和由氢和二氧化碳等物质之间的氧化还原反应释放的化学能,而且提供了陡峭的温度梯度,使得反应可以在最佳位置进行[113, 114]。其次,该理论认为生命的早期化学反应不是在三维溶液中发生,而是在如铁硫化物之类的二维矿物表面上发生。因此,它强调了表面的关键作用,包括但不限于:(1) 某些反应在表面上的速率比在溶液中的速率大得多,因为表面具有高选择性作用[30];(2) 这些热液喷口的表面可以提供许多过渡金属中心(主要是铁和镍)作为固碳反应的催化剂[115-118];(3) 这些固碳途径产生的小有机分子作为金属中心的有机配体,从无机气体逐渐形成自催化反应网络(例如,rTCA)[113, 114]。这一系列过程称为“表面代谢”[30]。虽然这个理论强调了二维边界的重要性,但它没有解释细胞能够自由生存所需的区室/边界是如何形成的。
后来,正如Martin和Russell所提出的[113, 114, 119],人们意识到黑烟囱型热液喷口可能过于炽热(约350°C),而地球上的第一种生命形式可能是在深海碱性热液喷口(约100°C)附近演化而来的(2000年首次发现的“失落城市”喷口系统就是这样的一个例子[120, 121])。这些碱性热液喷口包括大量微观孔洞,由薄膜状的金属硫化物壁覆盖。这个理论能够解决出现隔间的问题,而这在Wächtershäuser原始理论中是缺失的[113, 122]:
1. 尽管两性分子可以自发地自组成胶束或囊泡,但它们由一个分裂成两个仍然是具有挑战性的,可能需要一个非常复杂的机制。碱性热液喷口理论认为最早的细胞并不是自由生活的,而是有着类似于膜的物理边界,即微小的孔洞。进一步演化导致了自支撑的脂质膜和封闭的小室,从而离开微小的孔洞并变得自由生存。假设细菌(以及真核生物)和古菌具有完全不同类型的膜脂的事实支持这一假设,表明它们是独立演化的。
2. 微小的孔洞有助于浓缩新合成的分子,增加了形成寡聚体或聚合物的机会。
3. 细胞边界所组成的脂质合成,用于将细胞与外部环境隔离开来,直到几乎所有细胞功能都已准备就绪才变得不可或缺。
从某种意义上来说,碱性热液喷口中的区室可以被认为是“原细胞(protocell)”。实际上,如果不用持续浸泡在水环境中(例如在碱性热液喷口中),“原细胞(protocell)”的形成可能会更加容易,因为已有证明,湿润干燥循环和紫外线(UV)照射可以增强脂质形成膜,并导致更多种类的生物分子的合成[123-125]。另一种被广泛传播的“黏土假说”——黏土矿物是由晶体组成的水合铝硅酸盐(例如高岭石和蒙脱石)——在1980年代流行起来,而最初是由Graham Cairns-Smith于1960年代中期首次提出的[126]。这一假设甚至认为,粘土晶体本身,更像是边界,显然是非生命的,是首先被自然选择的个体。后来,在由粘土晶体表面催化的复杂有机分子执行了一次“遗传接管”之后,这些分子系统成为受选择的个体[127-129]。
总之,无论是铁硫世界假说还是Russell的理论,都非常强调物理表面的作用[113、114、119、126]。所以在人造生命、设计原细胞 (protocell) 的时候我们能不能也采用这种思路,即先不考虑自由边界,先在物理边界的基础上制造自催化集,然后期望自由边界慢慢演化出来?这条路线像一个关于生命起源的假说,用这种思路来进行人造生命的实验性工作并不是很多。不过有一些工作可能会有一些启发,比如:最近Peng和Baum等人在一系列原始汤和黄铁矿颗粒连续转移实验中,证明了自复制矿物质相关自催化系统的自发出现和富集的潜力[130,131]。在Wołos和Grzybowski等人开发的化学模拟系统中,发现可以通过醛类化学物的多次反应,自发形成表面活性分子 (例如,直链的饱和脂肪酸和α-羟基酸表面活性剂),意味着在早期地球环境中确实有可能从化学反应网络为起点,通过简单的反应步骤,自发形成原始的细胞-环境边界、甚至囊泡结构[35]。最近,研究人员创造了一种无机中空胶体 (替代脂质作为细胞区隔),其表面有定义良好的微孔,能够执行主动运输,因此是一种无机细胞类似体[132]。
3.3 如何从简单演化到复杂:梯径系统
正如前面提到的,从相对简单的由物理边界限制的自催化反应网络开始,随后的进化过程将允许形成复杂分子,包括例如可以作为独立边界成分的脂质。但是,这种逻辑是否得到任何理论的支持呢?事实上,上述许多研究已经暗含了这种逻辑,即从简单到复杂演化是自然趋势。例如,铁硫世界假说暗示,一旦在表面上建立原始的自催化网络,就会慢慢涌现出更复杂的自催化网络和复杂的有机化合物,甚至信息分子[30]。在RAF理论中,前一级的RAF集合将成为后一级RAF集合的原材料,从而促进越来越复杂的网络的形成[96]。在Peng和Baum提出的种子依赖性自催化系统(SDASs)理论中认为,早期地球上的开放环境可能通过自催化化学反应系统的连续激活而逐渐复杂化 (不是像RAF理论中所提出的通过原材料,而是通过“种子”触发) [107]。尽管如此,上述这些论述是否有更普遍的理论基础?一个系统需要符合哪些条件,才能实现从简单到复杂的演化?毕竟不是所有的系统都能有这种特性。
首先, 需要讨论一下什么是“复杂”,特别是演化中的复杂,通常它是模棱两可的[133,134]。让我们看一个有冲突的例子:我们通常会说生命(比如蛋白质分子)的复杂度比理想气体分子系统的复杂度高,因为前者更有序;但是当我们问晶体和蛋白质分子谁更复杂的时候,直觉告诉我们似乎还是生命更复杂。但此时我们就面临一个看起来的悖论,即前面我们说更有序意味着更复杂,但这里显然晶体比蛋白质分子更有序,为什么我们还是认为蛋白质分子更复杂呢?最近我们提出的梯径理论(Ladderpath theory)认为,我们通常直觉上说的“复杂”其实有两根轴,一个叫梯径度(λ),定量表示描述构造目标系统的代价和困难,另一个叫有序度(ω),表示目标系统是怎样有序地、以层级的方式构造起来,定量描述这种有序的程度是多少。这两者的结合才是复杂度(比如可以取两者的乘积) [135, 136]。如下图3所示,理想气体分子系统和晶体是两种极端情况,前者靠近x轴,表示梯径度(λ)很高有序度(ω)很低,对应地,后者靠近y轴,表示梯径度(λ)很低有序度(ω)很高 (参见文献[136]) 。而生命系统应该是两者都很高,因而复杂度很高。 图3. 梯径理论提出的复杂性的两个方面的概念示意图。请参阅正文。
图3. 梯径理论提出的复杂性的两个方面的概念示意图。请参阅正文。
梯径理论进一步提出了“梯径系统”的定义,一个系统如果满足以下两个条件,则称为梯径系统:
1. 能够生成新组件 (“成核”)
2. 部分或所有组件能够复制 (“复制”)
并且梯径系统的复杂度可以自然而然地增加。为了说明这一点,我们考虑一个高度简化的理想实验。如图4a所示,假设该系统有能力生成新的基础构件或结构(用黑色和灰色的点表示),但这些结构无法复制(因此这个系统不是梯径系统)。最初,有许多基础结构,即单个点。结构[A]可以通过五个连续的组合生成。接下来,考虑图4b中的另一个系统,假设它也可以生成新的结构,但这些结构可以复制(即一个梯径系统)。我们可以看到,结构[B]可以在五个步骤内生成。根据梯径理论,我们可以计算[A]和[B]的复杂性:(1) [A]和[B]的梯径度(λ)都是5,因为它们都需要五步;(2) 基于有序度ω = S – λ 的定义(其中S是大小指数,即在这种情况下构成黑点总数),[B]的有序度(ω)是24-5=19,而[A]的ω是6-5=1;(3) 因此,[B]的复杂度是19×5,而[A]的复杂度是1×5,比前者小。这个结论表明[B]比[A]更复杂,与我们的直觉一致。因此,通常可以说,在梯径系统中,生成更大、更复杂的结构更容易且需要更少的步骤,而在其他系统中则不是这样。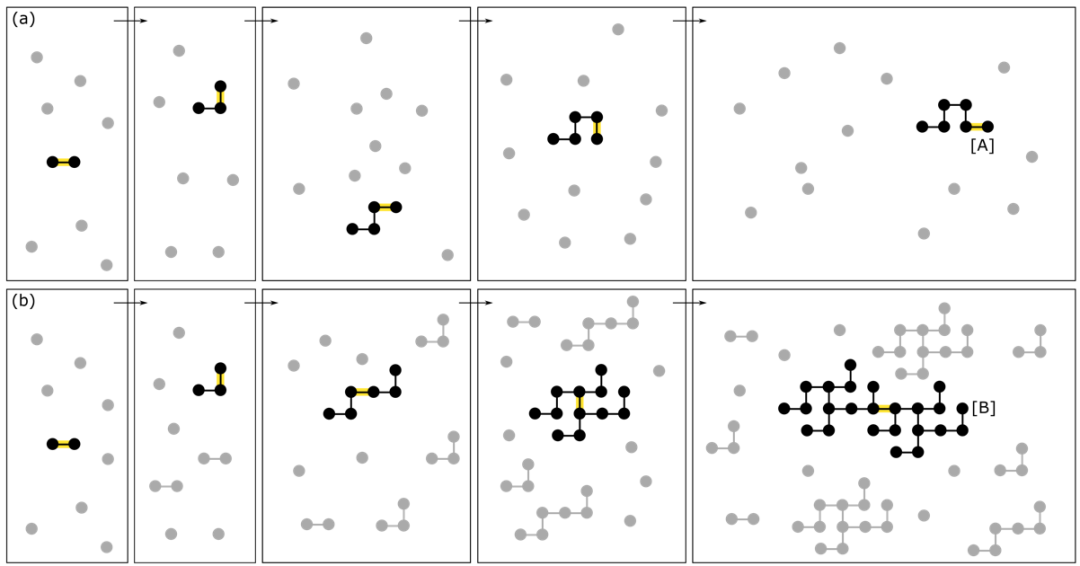
图4. (a) 非梯径系统的演化。(b) 梯径系统的演化。方框代表系统(其大小无关紧要)。系统从左到右演化。每个新的组合(即产生新结构的方式)可以被看作是在两个黄色的原子之间形成一个新的化学键。新产生的结构用黑色表示,而已经存在的结构用灰色表示(仅易于读者识别,与动力学无关)。
因此,我们的预期是有依据的。因为(1)自催化集在广义上具有信息(即载体和内容未分开);(2)自催化集满足梯径系统的两个条件,所以其复杂度会有自然增加的倾向,其所蕴含的信息也就会慢慢积累而扩大。当然具体的信息是如何增加,取决于在导向演化的过程中,实验上如何去引导。只要每一次迭代都要求自复制系统的出现,那么从最开始的原初广义信息、到生成越来越复杂的网络和分子、最后生成某种可以记录信息的分子,即信息载体和内容分开,即狭义信息,就是可以期待的。上文提到的3个期待复杂性增加的例子 (铁硫世界假说,RAF理论,种子依赖性自催化系统(SDASs)理论中) 其实也可以用梯径理论给予支撑,虽然它们本身并没有提到过,只是觉得这是个理所应当的过程,这是因为那3个例子所涉及到的系统其实都满足成核和复制这两个条件,都是梯径系统。
最后我们想强调,我们这里虽然说是从自催化集(代谢)开始,然后期望遗传和边界会自然涌现,以组成我们目前的生命形式。事实上,无论谁优先,只要是能够将三个组分和最终整体进行连通的线路,都是可能线路。在实际的地球历史中,这些连通有可能多次发生,也可能只发生一次,也许具有偶然性。也许再给一次机会,在同样的物理化学环境中,也可能走通另一条线。这时,就会点亮“生命的多重可实现”的理论框架,而我们仍可以在多条路径中找到一条最短、或者最可能、或者最通用的路径。因此,合成生命的路径不需要等同于历史路径,因为历史事实受条件限制并具有一定偶然性。在合成生物学的设计中,我们可以将通用路径用于指导新的类生命合成过程,当真实的设计需求与通用路径相比较后,结合现实可及的手段,就有很大可能帮助实验者找到可执行性最高的方案。
4. 讨论和结论
在总结我们在本文中所述的内容之前,我们用两段话来讨论并强调人造生命研究的目标,以更好地激励这项工作。在理论层面上, 人造生命的最终问题实际上要归结到“信息”是如何“从无到有”出现的,并且复现这个过程。不过首先, 这里我们必须明确“信息”到底是指的什么。其中一种观点是比较宽泛的、广义的,即任何方式使得不确定性的减小 (对应于某些时间概率分布的变化),都可以称为“信息”,这种观点大多是物理学家所持有的,即所谓的信息熵,从信息论中而来。比如在一个自催化反应系统中,一个种子分子可以触发一整个反应,从而改变系统的组分,这又会影响后续系统的发展,这即是改变了事件发生的概率分布,在这个意义上,自催化系统具有信息。因此,已经提出的自催化集有“初步遗传性”[98]。此外,在“脂质世界”假说中,脂质的分子组成也被定义为“组成信息”[67]。关于信息的另一个思想流派更具体,认为信息载体和内容必须分离和独立[8]。例如,小说书中所编码的信息是故事和情节等,与信息载体 (例如,书上的纸张类型和使用的墨水) 无关。在地球上的生命中,这基本就是指,生物的遗传信息编码在线性聚合物如 DNA 和 RNA中,它们的侧链以离散的化学形式出现,通常遵循中心法则的流向来传递[137]。对于人造生命,信息载体可能不同于DNA / RNA,只是我们现在还不知道有什么更好的实现方式。
事实上,人造生命是想解决“无信息 -> 广义信息 -> 狭义信息”,并且主要是后面那个箭头,因为广义信息的出现是比较容易的,就像大量存在的自催化集。Szathmáry曾经提出,生命起源具有三个阶段,从整体复制体(Holistic) -> 具有有限遗传信息的复制体(limited hereditary) -> 具有无限遗传信息的复制体(unlimited information)。这里,“整体”指的是广义信息,而后两者指的是狭义信息(有限遗传指RNA,无限遗传指DNA)[138]。无论如何,人造生命这个问题要从广义信息涌现出狭义信息的时候才算达到目标。只有这样,才算明白了生命的基本法则即其多重可实现性,才能回答生命起源的机制问题(这不仅仅是指地球生命起源的历史问题)。
所以,在这篇文章中,我们首先回顾了目前合成生物学中最常用的两条路径:自上而下的方法(在第2.1节中简要描述),该方法通过简化现有生物系统的复杂性来实现,以及自下而上的方法(在第2.2节中详细介绍),该方法通过整合定义明确的组件来实现。我们可以看到,它们都不能足够地解决该领域中的挑战,因为它们都预设了独立信息载体(狭义信息形式)的存在。因此,在第3节中,我们提出了另一种可能性,也可以归类为“自下而上”的方法,但是从更原始的层面开始,即生命起源:首先构建利用物理边界作为初始区室的自催化化学反应网络,然后设计定向进化系统,预期最终独立区室的出现,使得系统变得自由生存(这种预期是由一个新近发展的理论,即梯径理论支持的,该理论在第3.3节中概述)。这实际上像在模拟生命起源的过程。在经过生命起源这几十年的发展,有很多定量数学模型等方法工具,但是目前这些理论工具还未引入到人造生命的脉络中来。通过这篇文章,我们希望引起合成生物学家和实验科学家对更理论的视角的关注,并促进生命起源研究者与人造生命研究者之间的交流。
此外,值得一提的是,有一些技术具有创造人造生命的巨大潜力。例如,微流控技术被用于整合明确定义的组件[139]:Deshpande等人使用微流控芯片将他们选择的蛋白质注入脂质囊泡中,这种技术被命名为辛醇辅助脂质体组装 [140];Weiss等人使用微流控将ATP合酶“上传”到囊泡中,形成能够在囊泡内制造ATP的原始线粒体[141]。对于软件方面,已经开发出“CatlyNet”程序,可以从任何反应和食物集合中计算RAFs(粗略地说,是自催化集)和其他集合[108,142]。与反向合成的想法不同 (尽管反向合成包括许多软件,例如Chematica和RetroPath2.0[143,144]) ,并有“Allchemy”平台已被开发出来,用于探索合成通路,从用户定义的起始成分中设计可能的从简单分子到复杂大分子的前生物进化过程,使研究人员能够在不断尝试创造人造生命时设计可能的前生物进化通路[35]。不仅可以在软件中识别通路,而且最近发展的自动化化学可以帮助研究人员合成各种分子,在创造人造生命需求的不同分子的不断尝试中可以节省大量的精力,例如Burger等人开发了一台自动合成机器人,在八天内执行了688个连续反应,发现了一种从水中产生氢的光催化剂[145],以及Chemputer系统,在几天内自动合成了三种药物化合物,其收率可与之前的文献报道相媲美[146]。此外,还可以运用定向进化的思想,应用突变-选择-积累循环和串行转移技术。最近的发展包括机器学习,可以从失败的试验中筛选和重新选择废弃的信息,从而提高效率[147,148]。最后,需要注意的是,相关技术不仅限于上述提到的技术。
合成生物学对基因回路的设计及利用现有材料进行膜、遗传物质与代谢的整合尝试,也许并不能完全解释地球生命如何起源;反之,如果生命起源研究没有解决从广义信息到狭义信息的转变如何发生,那么对构建“人造生命”或“生命的多重实现”所具有的帮助也会很有限。短期来看,分离的信息 (即翻译,或酶参与的复制)如何从自催化网络起源,是这两个学科向前发展均要面对的重要问题。长远来看,对于生命起源问题:可能需要从原始地球分子和早期自催化循环开始,逐步实现一个个新功能,直到完成前细胞构建才能被认为是真正解析和重构了生命起源,并真正证明了细胞可以从物理化学世界经过演化自然产生。这一过程的重构,对于星际生物学中识别外星生命、以及在可能的不同于地球的环境 (甚至于数字虚拟的环境中) 重现这一起源和早期演化过程,(即证实了生命的多重可实现性)是充分且必要的。反过来,在这一连续过程被重构之后,地球生命从元件、到组织方式、到整合步骤,才会有完全意义上的理论指导和可能性分析,从合成生物学的角度来说,我们才拥有了完全认识、设计、再创生命的能力。这可能并不是一个具体的时间点,而是通过漫长却越来越令人兴奋的逐步努力来实现的。
致谢
我们感谢集智俱乐部组织的生命自生成结构读书会以及集智科学研究中心的生物起源知识图谱项目的支持。
参考文献
1. Miller, S. L. (1953) A production of amino acids under possible primitive earth conditions. Science, 117, 528-529
2. Hutchison, C. A., Chuang, R. Y., Noskov, V. N., Assad-Garcia, N., Deerinck, T. J., Ellisman, M. H., Gill, J., Kannan, K., Karas, B. J., Ma, L., et al. (2016) Design and synthesis of a minimal bacterial genome. Science, 351, aad6253
3. Xu, C., Martin, N., Li, M., and Mann, S. (2022) Living material assembly of bacteriogenic protocells. Nature, 609, 1029-1037
4. Koshland, D. E. (2002) The seven pillars of life. Science, 295, 2215-2216
5. Smith, K. C., and Mariscal, C. (1970) Social and conceptual issues in astrobiology, Cambridge University Press
6. Cleland, C. E., and Chyba, C. F. (2007) Planets and life: The emerging science of astrobiology, Cambridge University Press
7. Schrodinger, E. (1944) What is life? And mind and matter, Cambridge University Press
8. Ganti, T. (2003) The principles of life, Oxford University Press UK
9. Shapiro, R. (2007) A simpler origin for life. Sci. Am., 296, 46–53
10. Maturana, H. R., and Varela, F. J. (1991) Autopoiesis and cognition: The realization of the living, Springer Science & Business Media
11. Joyce, C. M., and Steitz, T. A. (1994) Function and structure relationships in DNA pol ymerases. Annu. Rev. Biochem., 63, 777-822
12. Szostak, J. W., Bartel, D. P., and Luisi, P. L. (2001) Synthesizing life. Nature, 409, 387–390
13. Benner, S. A. (2010) Defining life. Astrobiology, 10, 1021-1230
14. Luisi, P. L. (1998) About various definitions of life. Biosphere, 28, 613–622
15. Damiano, L., and Luisi, P. L. (2010) Towards an autopoietic redefinition of life. Orig. Life Evol. Biosph., 40, 145-149
16. Allwood, A., Amend, J., Anbar, A., Billings, L., Blankenship, R., Boss, A., Braakman, R., Cavanaugh, C., Copley, S., Driscoll, P., et al. (2015) NASA astrobiology strategy, NASA
17. Powell, K. (2018) Biology from scratch: Built from the bottom up, synthetic cells could reveal the boundaries of life. Nature, 563, 172-175
18. Gibson, D. G., Glass, J. I., Lartigue, C., Noskov, V. N., Chuang, R. Y., Algire, M. A., Benders, G. A., Montague, M. G., Ma, L., Moodie, M. M., et al. (2010) Creation of a bacterial cell controlled by a chemically synthesized genome. Science, 329, 52-56
19. Thornburg, Z. R., Bianchi, D. M., Brier, T. A., Gilbert, B. R., Earnest, T. M., Melo, M. C. R., Safronova, N., Saenz, J. P., Cook, A. T., Wise, K. S., et al. (2022) Fundamental behaviors emerge from simulations of a living minimal cell. Cell, 185, 345-360
20. Pelletier, J. F., Sun, L., Wise, K. S., Assad-Garcia, N., Karas, B. J., Deerinck, T. J., Ellisman, M. H., Mershin, A., Gershenfeld, N., Chuang, R. Y., et al. (2021) Genetic requirements for cell division in a genomically minimal cell. Cell, 184, 2430-2440
21. Shao, Y., Lu, N., Wu, Z., Cai, C., Wang, S., Zhang, L. L., Zhou, F., Xiao, S., Liu, L., Zeng, X., et al. (2018) Creating a functional single-chromosome yeast. Nature, 560, 331-335
22. Oparin, A. I. (1938) Organisms and the earth. Nature, 142, 412-413
23. Muller, H. J. (1947) The gene. Proc. R. Soc. Med., 134, 1-37
24. Lazcano, A. (2010) Which way to life? Orig. Life Evol. Biosph., 40, 161-167
25. Kauffman, S. A. (1993) The origins of order: Self-organization and selection in evolution, Oxford University Press.
26. Orgel, L. E. (2008) The implausibility of metabolic cycles on the prebiotic earth. PLoS Biol., 6, e18
27. Mizuno, T., and Weiss, A. H. (1974) Synthesis and utilization of formose sugars. Adv. Carbohydr. Chem. Biochem., 29, 173-227
28. Schwartz, A. W., and Goverde, M. (1982) Acceleration of hcn oligomerization by formaldehyde and related compounds: Implications for prebiotic syntheses J. Mol. Evol. , 18, 351-353
29. Morowitz, H. J. (1999) A theory of biochemical organization, metabolic pathways, and evolution. Complexity, 4, 39-53
30. Wächtershäuser, G. (1988) Before enzymes and templates: Theory of surface metabolism. Clin. Microbiol. Rev., 52, 452–484
31. Wächtershäuser, G. (1990) Evolution of the first metabolic cycles. Proc. Natl. Acad. Sci. USA, 87, 200-204
32. Wächtershäuser, G. (2000) Life as we don’t know it. Science, 289, 1307-1308
33. Lazcano, A., and Miller, S. L. (1999) On the origin of metabolic pathways. J. Mol. Evol., 49, 424–431
34. Delaye, L., Becerra, A., and Lazcano, A. (2005) The last common ancestor: What’s in a name? Orig. Life Evol. Biosph., 35, 537-554
35. Wolos, A., Roszak, R., Zadlo-Dobrowolska, A., Beker, W., Mikulak-Klucznik, B., Spolnik, G., Dygas, M., Szymkuc, S., and Grzybowski, B. A. (2020) Synthetic connectivity, emergence, and self-regeneration in the network of prebiotic chemistry. Science, 369, 1584
36. Troland, L. T. (1917) Biological enigmas and the theory of enzyme action. Am. Nat., 51, 321-350
37. Woese, C. R., Dugre, D. H., Saxinger, W. C., and Dugre, S. A. (1966) The molecular basis for the genetic code. Proc. Natl. Acad. Sci. USA, 55, 966–974
38. Crick, P. H. (1968) The origin of the genetic code J. Mol. Biol., 38, 367-379
39. Orgel, L. E. (1968) Evolution of the genetic apparatus. J. Mol. Biol., 38, 381-393
40. Gilbert, W. (1986) The RNA world. Nature, 319, 618
41. Copley, S. D., Smith, E., and Morowitz, H. J. (2007) The origin of the RNA world: Co-evolution of genes and metabolism. Bioorg. Chem., 35, 430-443
42. Nelson, J. W., and Breaker, R. R. (2017) The lost language of the RNA world. Sci. Signal., 10, eaam8812
43. Nissen, P., Hansen, J., Ban, N., Moore, P. B., and Steitz, T. A. (2000) The structural basis of ribosome activity in peptide bond synthesis. Science, 289, 920-930
44. Turk, R. M., Chumachenko, N. V., and Yarus, M. (2010) Multiple translational products from a five-nucleotide ribozyme. Proc. Natl. Acad. Sci. USA, 107, 4585-4589
45. Muller, F., Escobar, L., Xu, F., Wegrzyn, E., Nainyte, M., Amatov, T., Chan, C. Y., Pichler, A., and Carell, T. (2022) A prebiotically plausible scenario of an RNA-peptide world. Nature, 605, 279-284
46. Aravind, L., Anantharaman, V., and Koonin, E. V. (2002) Monophyly of class I aminoacyl tRNA synthetase, USPA, ETFP, photolyase, and PP-ATPase nucleotide-binding domains: Implications for protein evolution in the RNA. Proteins, 48, 1-14
47. Danchin, A., Fang, G., and Noria, S. (2007) The extant core bacterial proteome is an archive of the origin of life. Proteomics, 7, 875-889
48. Johnston, W. K., Unrau, P. J., Lawrence, M. S., Glasner, M. E., and Bartel, D. P. (2001) RNA-catalyzed RNA polymerization: Accurate and general RNA-templated primer extension. Science, 292, 1319-1325
49. Joyce, G. F. (2009) Evolution in an RNA world. Cold Spring Harb. Sym., 74, 17–23
50. Mizuuchi, R., Furubayashi, T., and Ichihashi, N. (2022) Evolutionary transition from a single RNA replicator to a multiple replicator network. Nat. Commun., 13, 1460
51. Oró, J. (1961) Mechanism of synthesis of adenine from hydrogen cyanide under possible primitive earth conditions. Nature, 191, 1193–1194
52. Abelson, P. H. (1966) Chemical events on the primitive earth. Proc. Natl. Acad. Sci. USA, 55, 1365-1372
53. Roy, D., Najafian, K., and Schleyer, P. v. R. (2007) Chemical evolution: The mechanism of the formation
of adenine under prebiotic conditions. Proc. Natl. Acad. Sci. USA, 104, 17272–11727
54. Eschenmoser, A. (2007) On a hypothetical generational relationship between hcn and constituents of the reductive citric acid cycle. Chem. Biodivers., 4, 554-573
55. Saladino, R., Botta, G., Pino, S., Costanzo, G., and Di Mauro, E. (2012) Genetics first or metabolism first? The formamide clue. Chem. Soc. Rev., 41, 5526-5565
56. Saladino, R., Crestini, C., Pino, S., Costanzo, G., and Di Mauro, E. (2012) Formamide and the origin of life. Phys. Life Rev., 9, 84-104
57. Muchowska, K. B., Varma, S. J., and Moran, J. (2020) Nonenzymatic metabolic reactions and life’s origins. Chem. Rev., 120, 7708-7744
58. Chen, I. A., and Walde, P. (2010) From self-assembled vesicles to protocells. CSH Perspect. Biol., 2, a002170
59. Cornell, C. E., Black, R. A., Xue, M., Litz, H. E., Ramsay, A., Gordon, M., Mileant, A., Cohen, Z. R., Williams, J. A., Lee, K. K., et al. (2019) Prebiotic amino acids bind to and stabilize prebiotic fatty acid membranes. Proc. Natl. Acad. Sci. USA, 116, 17239-17244
60. Chen, I. A. (2006) The emergence of cells during the origin of life. Science, 314, 1558–1559
61. Kahana, A., and Lancet, D. (2021) Self-reproducing catalytic micelles as nanoscopic protocell precursors. Nat. Rev. Chem., 5, 870-878
62. Shapiro, R. (2007) The sudden appearance of a large self-copying molecule such as RNA was exceedingly improbable. Energy-driven networks of small molecules afford better odds as the initiators of life. Sci. Am., 296, 46–53
63. Chang, T. (2007) 50th anniversary of artificial cells: Their role in biotechnology, nanomedicine, regenerative medicine, blood substitutes, bioencapsulation, cell/stem cell therapy and nanorobotics. Artif. Cell Blood Sub. Biotech., 35, 545-554
64. Damer, B., and Deamer, D. (2015) Coupled phases and combinatorial selection in fluctuating hydrothermal pools: A scenario to guide experimental approaches to the origin of cellular life. Life, 5, 872-887
65. Mukwaya, V., Mann, S., and Dou, H. (2021) Chemical communication at the synthetic cell/living cell interface. Commun. Chem., 4, 161
66. Mukwaya, V., Zhang, P., Liu, L., Dang-i, A. Y., Li, M., Mann, S., and Dou, H. (2021) Programmable membrane-mediated attachment of synthetic virus-like nanoparticles on artificial protocells for enhanced immunogenicity. Cell Rep. Phys. Sci., 2, 100291
67. Segré, D., and Lancett, D. (2000) Composing life. EMBO Rep., 1, 217–222
68. Segré, D., Deamer, D. B.-E. D. W., and Lancet, D. (2001) The lipid world. Origins Life Evol. B., 31, 119-145
69. Serrano-Luginbühl, S., Ruiz-Mirazo, K., Ostaszewski, R., Gallou, F., and Walde, P. (2018) Soft and dispersed interface-rich aqueous systems that promote and guide chemical reactions. Nat. Rev. Chem., 2, 306-327
70. Lancet, D., Segre, D., and Kahana, A. (2019) Twenty years of “lipid world”: A fertile partnership with david deamer. Life, 9, 77
71. Kurihara, K., Tamura, M., Shohda, K., Toyota, T., Suzuki, K., and Sugawara, T. (2011) Self-reproduction of supramolecular giant vesicles combined with the amplification of encapsulated DNA. Nat. Chem., 3, 775-781
72. Neumann, J. V. (1948) The general an logical theory of automata, Papers of John Von Neumann on Computing & Computer Theory
73. Gardner, M. (1970) The fantastic combinations of john conway’s new solitaire game “life”. Sci. Am., 223, 120-123
74. Langton, C. G. (1986) Studying artificial life with cellular automata. Physica D, 22, 120-149
75. Wolfram, S. (2002) A new kind of science, Wolfram Media
76. Weihs, D., Gefen, A., and Vermolen, F. J. (2016) Review on experiment-based two- and three-dimensional models for wound healing. Interface Focus, 6, 20160038
77. Shakeel, A., and Love, P. J. (2013) When is a quantum cellular automaton (QCA) a quantum lattice gas automaton (QLGA)? J. Math. Phys., 54,
78. Cole, T., and Lusth, J. C. (2001) Quantum-dot cellular automata. Prog. Quantum. Electron., 25, 165–189
79. Langton, C. G. (1989) Artificial life, The MIT Press
80. Pilat, M. L., and Jacob, C. (2008) Creature academy: A system for virtual creature evolution. IEEE Trans. Evol. Comput., 3289-3297
81. Reynolds, C. W. (1987) Flocks, herds, and schools: A distributed behavioral model. Comput. Graph., 21, 25-34
82. Vicsek, T., Czirok, A., Ben-Jacob, E., Cohen, I. I., and Shochet, O. (1995) Novel type of phase transition in a system of self-driven particles. Phys. Rev. Lett., 75, 1226-1229
83. Hickinbotham, S., and Stepney, S. (2015) Environmental bias forces parasitism in Tierra. Proc. Eur. Conf. Artif. Life, 294-301
84. Ofria, C., and Wilke, C. O. (2004) Avida: A software platform for research in computational evolutionary biology. Artif. Life, 10, 191–229
85. Illman, S. (2001) Hilbert’s fifth problem: Review. J. Math. Sci., 105, 1843-1847
86. Holland, J. H. (1975) Adaptation in natural and artificial systems: An introductory analysis with applications to biology, control, and artificial intelligence, The MIT Press
87. Deb, K., Member, A., IEEE, Pratap, A., Agarwal, S., and Meyarivan, T. (2002) A fast and elitist multiobjective genetic algorithm: NSGA-II. IEEE Trans. Evol. Comput., 6, 182-197
88. Szigeti, B., Gleeson, P., Vella, M., Khayrulin, S., Palyanov, A., Hokanson, J., Currie, M., Cantarelli, M., Idili, G., and Larson, S. (2014) Openworm: An open-science approach to modeling caenorhabditis elegans. Front. Comput. Neurosci., 8, 137
89. Givon, L. E., and Lazar, A. A. (2016) Neurokernel: An open source platform for emulating the fruit fly brain. PLoS One, 11, e0146581
90. Xu, C., Hu, S., and Chen, X. (2016) Artificial cells: From basic science to applications. Mater Today, 19, 516-532
91. Gomez-Marquez, J. (2021) What is life? Mol. Biol. Rep., 48, 6223-6230
92. Liu, Y., and Sumpter, D. J. T. (2018) Mathematical modeling reveals spontaneous emergence of self-replication in chemical reaction systems. J. Biol. Chem., 293, 18854-18863
93. Stewart, J. E. (2018) The origins of life: The managed-metabolism hypothesis. Found. Sci., 24, 171-195
94. Ameta, S., Matsubara, Y. J., Chakraborty, N., Krishna, S., and Thutupalli, S. (2021) Self-reproduction and darwinian evolution in autocatalytic chemical reaction systems. Life, 11, 308
95. Kauffman, S. (1995) At home in the universe: The search for laws of self-organization and complexity, Oxford University Press
96. Hordijk, W., and Steel, M. (2012) Autocatalytic sets extended: Dynamics, inhibition, and a generalization. J. Syst. Chem., 3, 5
97. Liu, Y., Hjerpe, D., and Lundh, T. (2020) Side reactions do not completely disrupt linear self-replicating chemical reaction systems. Artif. Life, 26, 327-337
98. Liu, Y. (2020) On the definition of a self-sustaining chemical reaction system and its role in heredity. Biol. Direct., 15, 15
99. Fontana, W., and Buss, L. W. (1994) “The arrival of the fittest”: Toward a theory of biological organization. Bull. Math. Biol., 56, 1–64
100. Dittrich, P., and di Fenizio, P. S. (2007) Chemical organisation theory. Bull. Math. Biol., 69, 1199-1231
101. Hordijk, W., Steel, M., and Dittrich, P. (2018) Autocatalytic sets and chemical organizations: Modeling self-sustaining reaction networks at the origin of life. New J. Phys., 20, 015011
102. Sousa, F. L., Hordijk, W., Steel, M., and Martin, W. F. (2015) Autocatalytic sets in E. coli metabolism. J. Syst. Chem., 6, 4
103. Semenov, S. N., Kraft, L. J., Ainla, A., Zhao, M., Baghbanzadeh, M., Campbell, V. E., Kang, K., Fox, J. M., and Whitesides, G. M. (2016) Autocatalytic, bistable, oscillatory networks of biologically relevant organic reactions. Nature, 537, 656-660
104. Sadownik, J. W., Mattia, E., Nowak, P., and Otto, S. (2016) Diversification of self-replicating molecules. Nat. Chem., 8, 264-269
105. Monreal Santiago, G., Liu, K., Browne, W. R., and Otto, S. (2020) Emergence of light-driven protometabolism on recruitment of a photocatalytic cofactor by a self-replicator. Nat. Chem., 12, 603-607
106. Nanda, J., Rubinov, B., Ivnitski, D., Mukherjee, R., Shtelman, E., Motro, Y., Miller, Y., Wagner, N., Cohen-Luria, R., and Ashkenasy, G. (2017) Emergence of native peptide sequences in prebiotic replication networks. Nat. Commun., 8, 434
107. Peng, Z., Linderoth, J., and Baum, D. A. (2022) The hierarchical organization of autocatalytic reaction networks and its relevance to the origin of life. PLoS Comput. Biol., 18, e1010498
108. Xavier, J. C., Hordijk, W., Kauffman, S., Steel, M., and Martin, W. F. (2020) Autocatalytic chemical networks at the origin of metabolism. Proc. Biol. Sci., 287, 20192377
109. Adam, Z. R., Fahrenbach, A. C., Jacobson, S. M., Kacar, B., and Zubarev, D. Y. (2021) Radiolysis generates a complex organosynthetic chemical network. Sci. Rep., 11, 1743
110. Keller, M. A., Turchyn, A. V., and Ralser, M. (2014) Non-enzymatic glycolysis and pentose phosphate pathway-like reactions in a plausible archean ocean. Mol. Syst. Biol., 10, 725
111. Pedersen, R. B., Rapp, H. T., Thorseth, I. H., Lilley, M. D., Barriga, F. J., Baumberger, T., Flesland, K., Fonseca, R., Fruh-Green, G. L., and Jorgensen, S. L. (2010) Discovery of a black smoker vent field and vent fauna at the arctic mid-ocean ridge. Nat. Commun., 1, 126
112. Georgieva, M. N., Little, C. T. S., Maslennikov, V. V., Glover, A. G., Ayupova, N. R., and Herrington, R. J. (2021) The history of life at hydrothermal vents. Earth-Sci. Rev., 217, 103602
113. Martin, W., and Russell, M. J. (2003) On the origins of cells: A hypothesis for the evolutionary transitions from abiotic geochemistry to chemoautotrophic prokaryotes, and from prokaryotes to nucleated cells. Philos. Trans. R. Soc. Lond. B Biol. Sci., 358, 59-83
114. Martin, W., and Russell, M. J. (2007) On the origin of biochemistry at an alkaline hydrothermal vent. Philos. Trans. R. Soc. Lond. B Biol. Sci., 362, 1887-1925
115. Roldan, A., Hollingsworth, N., Roffey, A., Islam, H. U., Goodall, J. B., Catlow, C. R., Darr, J. A., Bras, W., Sankar, G., Holt, K. B., et al. (2015) Bio-inspired CO2 conversion by iron sulfide catalysts under sustainable conditions. Chem. Commun., 51, 7501-7504
116. Camprubi, E., Jordan, S. F., Vasiliadou, R., and Lane, N. (2017) Iron catalysis at the origin of life. IUBMB Life, 69, 373-381
117. Belthle, K. S., and Tüysüz, H. (2022) Linking catalysis in biochemical and geochemical CO2 fixation at the emergence of life. ChemCatChem, e202201462
118. Varma, S. J., Muchowska, K. B., Chatelain, P., and Moran, J. (2018) Native iron reduces CO2 to intermediates and end-products of the acetyl-CoA pathway. Nat. Ecol. Evol., 2, 1019-1024
119. Weiss, M. C., Sousa, F. L., Mrnjavac, N., Neukirchen, S., Roettger, M., Nelson-Sathi, S., and Martin, W. F. (2016) The physiology and habitat of the last universal common ancestor. Nat. Microbiol., 1, 16116
120. Kelley, D. S., Karson, J. A., Fruh-Green, G. L., Yoerger, D. R., Shank, T. M., Butterfield, D. A., Hayes, J. M., Schrenk, M. O., Olson, E. J., Proskurowski, G., et al. (2005) A serpentinite-hosted ecosystem: The lost city hydrothermal field. Science, 307, 1428-1434
121. Kitadai, N., and Maruyama, S. (2018) Origins of building blocks of life: A review. Geosci. Front., 9, 1117-1153
122. Koonin, E. V., and Martin, W. (2005) On the origin of genomes and cells within inorganic compartments. Trends Genet., 21, 647-654
123. Mulkidjanian, A. Y., Bychkov, A. Y., Dibrova, D. V., Galperin, M. Y., and Koonin, E. V. (2012) Origin of first cells at terrestrial, anoxic geothermal fields. Proc. Natl. Acad. Sci. USA, 109, E821-E830
124. Patel, B. H., Percivalle, C., Ritson, D. J., Duffy, C. D., and Sutherland, J. D. (2015) Common origins of RNA, protein and lipid precursors in a cyanosulfidic protometabolism. Nat. Chem., 7, 301-307
125. Deamer, D., Damer, B., and Kompanichenko, V. (2019) Hydrothermal chemistry and the origin of cellular life. Astrobiology, 19, 1523-1537
126. Cairns-Smith, A. G. (1987) Genetic takeover: And the mineral origins of life, Cambridge University Press
127. Zimmer, C. (2004) What came before DNA? Discover.
128. Huang, W., and Ferris, J. P. (2006) One-step, regioselective synthesis of up to 50-mers of RNA oligomers by montmorillonite catalysis. J. Am. Chem. Soc., 128, 8914-8919
129. Kloprogge, J. T. T., and Hartman, H. (2022) Clays and the origin of life: The experiments. Life, 12, 259
130. Baum, D. A. (2018) The origin and early evolution of life in chemical composition space. J. Theor. Biol., 456, 295-304
131. Vincent, L., Berg, M., Krismer, M., Saghafi, S. S., Cosby, J., Sankari, T., Vetsigian, K., Ii, H. J. C., and Baum, D. A. (2019) Chemical ecosystem selection on mineral surfaces reveals long-term dynamics consistent with the spontaneous emergence of mutual catalysis. Life, 9, 80
132. Xu, Z., Hueckel, T., Irvine, W. T. M., and Sacanna, S. (2021) Transmembrane transport in inorganic colloidal cell-mimics. Nature, 597, 220-224
133. Thoren, H., and Gerlee, P. (2010) Weak emergence and complexity, MIT Press
134. Kolchinsky, A., and Wolpert, D. H. (2018) Semantic information, autonomous agency and non-equilibrium statistical physics. Interface Focus, 8, 20180041
135. Liu, Y., Mathis, C., Bajczyk, M. D., Marshall, S. M., Wilbraham, L., and Cronin, L. (2021) Exploring and mapping chemical space with molecular assembly trees. Sci. Adv., 7, eabj2465
136. Liu, Y., Di, Z., and Gerlee, P. (2022) Ladderpath approach: How tinkering and reuse increase complexity and information. Entropy, 24, 1082
137. Baum, D. A., and Lehman, N. (2017) Life’s late digital revolution and why it matters for the study of the origins of life. Life, 7, 34
138. Szathmary, E. (2000) The evolution of replicators. Philos. Trans. R. Soc. Lond. B Biol. Sci., 355, 1669-1676
139. Powell, K. (2018) Biology from scratch built: From the bottom up, synthetic cells could reveal the boundaries of life. Nature, 563, 172-175
140. Deshpande, S., Caspi, Y., Meijering, A. E., and Dekker, C. (2016) Octanol-assisted liposome assembly on chip. Nat. Commun., 7, 10447
141. Weiss, M., Frohnmayer, J. P., Benk, L. T., Haller, B., Janiesch, J. W., Heitkamp, T., Borsch, M., Lira, R. B., Dimova, R., Lipowsky, R., et al. (2018) Sequential bottom-up assembly of mechanically stabilized synthetic cells by microfluidics. Nat. Mater., 17, 89-96
142. Steel, M., Xavier, J. C., and Huson, D. H. (2020) The structure of autocatalytic networks, with application to early biochemistry. J. R. Soc. Interface, 17, 20200488
143. Lin, G.-M., Warden-Rothman, R., and Voigt, C. A. (2019) Retrosynthetic design of metabolic pathways to chemicals not found in nature. Curr. Opin.Sys. Biol., 14, 82-107
144. Mikulak-Klucznik, B., Golebiowska, P., Bayly, A. A., Popik, O., Klucznik, T., Szymkuc, S., Gajewska, E. P., Dittwald, P., Staszewska-Krajewska, O., Beker, W., et al. (2020) Computational planning of the synthesis of complex natural products. Nature, 588, 83-88
145. Burger, B., Maffettone, P. M., Gusev, V. V., Aitchison, C. M., Bai, Y., Wang, X., Li, X., Alston, B. M., Li, B., Clowes, R., et al. (2020) A mobile robotic chemist. Nature, 583, 237-241
146. Steiner, S., Wolf, J., Glatzel, S., Andreou, A., Granda, J. M., Keenan, G., Hinkley, T., Aragon-Camarasa, G., Kitson, P. J., Angelone, D., et al. (2019) Organic synthesis in a modular robotic system driven by a chemical programming language. Science, 363, 144
147. Wu, Z., Kan, S. B. J., Lewis, R. D., Wittmann, B. J., and Arnold, F. H. (2019) Machine learning-assisted directed protein evolution with combinatorial libraries. Proc. Natl. Acad. Sci. USA, 116, 8852-8858
148. Yang, K. K., Wu, Z., and Arnold, F. H. (2019) Machine-learning-guided directed evolution for protein engineering. Nat. Methods, 16, 687-694
(参考文献可上下滑动查看)
自生成结构读书会
生命是有心(意识)的,但当你一层层剥开生命的组织,却只有心脏而没有一颗“心灵”;打开大脑皮层看到一个个神经元,却看不到“意识”。然而,生命与意识都具有“自我生成“的能力,生命自发从非生命中生成,意识自发从生命中生成。更惊人的是,生命与意识的自我生成结构似乎很相似。如果这个假设成立,那么最可能的备选结构会是什么呢?
为了更深入地认识复杂活系统的自生成结构,集智科学家小木球(仇玮祎)联合周理乾、王东、董达、刘宇、苑明理、傅渥成、章彦博等科学哲学、计算机科学、物理学和生物化学等学科的一线研究者共同发起组织《自生成结构系列读书会》。其中第一季《共识——自生成结构与自复制自动机的研究背景》已经完结,读书会所形成的自生成结构社群集结了相关领域的教授、硕博及产业界人士。现在报名读书会即可解锁第一季读书会所有录播视频并入群,欢迎从事相关研究、对相关领域有浓厚兴趣的探索者报名参与交流。
详情请见:
自生成结构读书会启动:剖解生命、意识与智能的核心结构
推荐阅读
1. 外星生命或智能摆在面前,你能认出来吗?——“梯径”提供的思路 2. 生命起源与复杂性 | 刘宇访谈3. 人工细胞:自上而下创造生命 | 集智百科4. 生命起源百年探索:进化论、信息论、热力学谁是主角?5. 生命起源研究的新里程碑:Science刊文揭示前生命化学自催化网络6. 生命形式曾经多次发生?多重视角探寻生命起源的理论结构7. 张江:第三代人工智能技术基础——从可微分编程到因果推理 | 集智学园全新课程8. 成为集智VIP,解锁全站课程/读书会9. 加入集智,一起复杂!
点击“阅读原文”,报名读书会





 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง





