编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Ali Madani团队的一篇论文。基于注意力机制的模型在蛋白质序列的分类和生成任务中展现出了惊人的成功,这些任务对于人工智能驱动的蛋白质设计至关重要。然而,我们对于大规模模型和数据在有效蛋白质模型开发中所扮演角色的理解仍然不足。作者引入了一套名为ProGen2的蛋白质语言模型,模型的规模扩大到了64亿参数,并且在从基因组、宏基因组和免疫库数据库中提取的超过十亿蛋白质的不同序列数据集上训练。ProGen2模型在捕捉观察到的进化序列分布、生成新的可行序列,以及在不需要额外微调的情况下预测蛋白质适应性方面展示了最先进的性能。

蛋白质是生命的“打工人”, 它们在维持我们的健康和环境方面扮演着非常重要和多功能的角色。通过技术手段改造蛋白质,可以让它们在制药、农业、特殊化学品和燃料等多个行业中发挥作用。现在用于蛋白质改造的工具还很有限,所以科学家们主要依靠一种叫做“定向进化”的方法。这个方法通过随机改变一个蛋白质的初始序列,测试每个改变后的版本,并不断重复这个过程,直到蛋白质的功能达到预期的改善效果。自然界中的蛋白质有着丰富而复杂的多样性。现在,随着DNA测序技术的飞速发展,我们能以前所未有的速度收集到这些自然界中的蛋白质序列。同时,我们也看到机器学习模型在理解图像和自然语言的数据分布方面做得非常好。特别是一种叫做Transformer的模型,它被证明是一个强大的语言模型,能够处理多种类型的数据。理解蛋白质序列变化带来的功能效果对于精准设计蛋白质非常关键。
在这项工作中,作者对非常大规模模型和数据的影响进行了研究。作者训练了一系列模型,范围从1.51亿到64亿参数(单一蛋白质transformer中已发布的最大之一),在不同数据集上,总计10亿蛋白质序列,来自基因组、宏基因组和免疫库数据库。作者通过预测的结构和生物物理属性分析了通用和特定于家族的模型的生成。
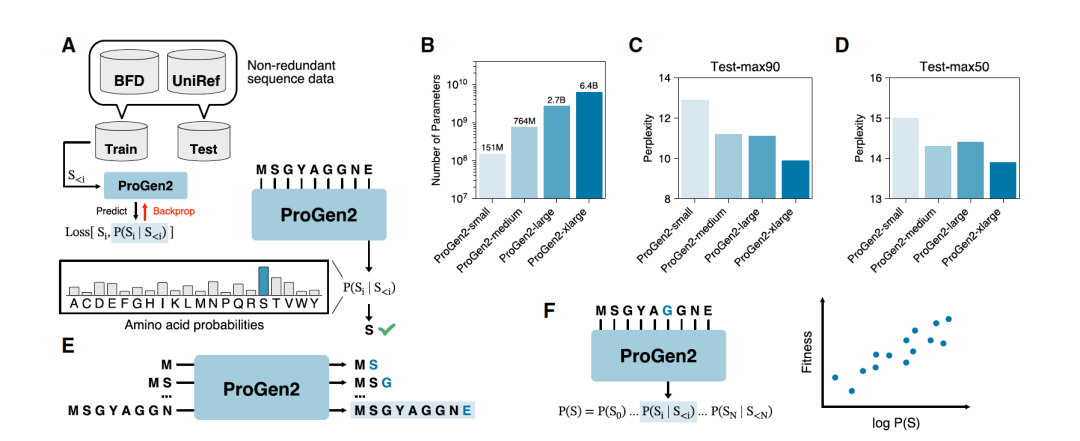
图 1
将生成性蛋白质语言模型扩展
自回归语言模型已被证明对于多种蛋白质工程任务非常有用,包括功能性序列生成和蛋白质适应性预测。这类模型起源于自然语言处理领域,最近的趋势显示,更大的模型性能越来越好,并且随着规模的扩大可以获得新的能力。为了进一步评估大规模PLM(预测语言模型)的行为,作者训练了一系列的模型,参数范围从1.51亿到64亿(图1A和1B),称为ProGen2。ProGen2模型主要训练了来自基因组和宏基因组来源的通用蛋白质集。这些序列来自于非冗余的UniProtKB子集,按90%的序列相似性聚类(UniRef90),以及按30%相似性聚类的BFD宏基因组数据库(BFD30)。作者还考虑了两种替代的数据分布用于模型训练。首先降低了对宏基因组序列的严格要求,并在UniRef90和BFD90(BFD按90%序列相似性聚类)的组合上训练ProGen2-BFD90。其次,作者探索了使用来自抗体空间(OAS)数据库的免疫库序列(即抗体)进行模型训练。OAS中的序列按85%的序列相似性聚类,并用来训练ProGen2-OAS。为了评估ProGen2模型,作者首先考虑日益增长的模型捕捉蛋白质序列分布的能力。然后探索了自回归PLM的应用,重点是序列生成(图1E)和适应性排名(图1F)。通过分析,作者强调了在训练和应用PLM时数据分布的作用和重要性。
作者首先评估了ProGen2模型在捕捉自然界中蛋白质序列的分布方面的能力。具体来说,作者关注的是它预测那些还未被观测到的自然序列的能力,并通过计算一个专门测试集上的“困惑度”(一种衡量模型性能的指标)来量化这种能力。更大的模型在这方面表现得更好,困惑度更低,这表明即使模型很大,它们也还远未到达过度拟合的程度。但是我们需要注意的是,这些结果只能反映模型在学习训练数据时表现出的能力。这些数据是从特定的分布中抽取出来的,作者称之为p0。但这并不一定能反映出分子适应性,也就是蛋白质在生物体内发挥作用的效能。更精确地说,让我们假设pN是蛋白质在进化过程中达到的一个稳定分布,这个分布与蛋白质的适应性有关。但是,由于数据集中可能存在的种种不平衡,实际上学习的数据分布p0可能与pN不一致。因此,即使能够准确估计出训练数据的分布p0,这并不意味着模型也能准确估计出pN或蛋白质的适应性。
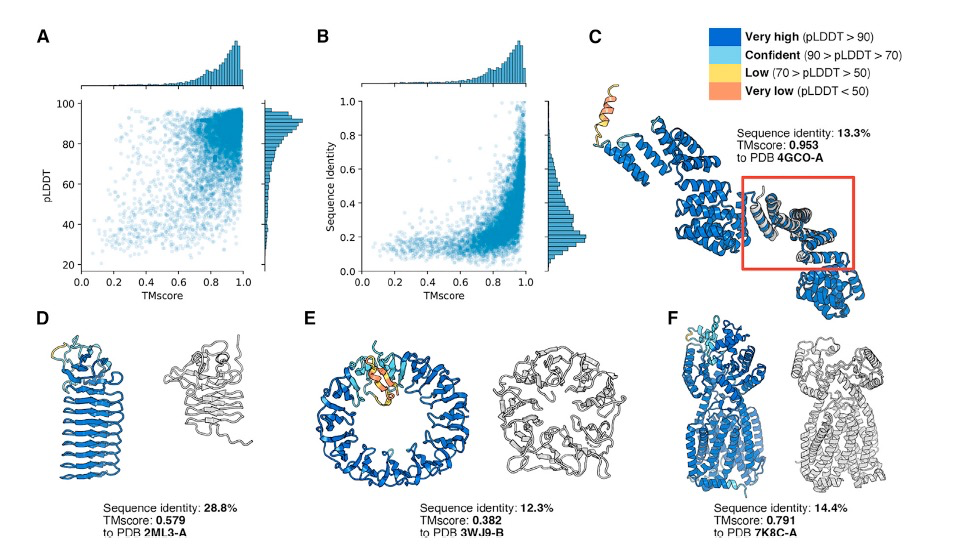
图 2
蛋白质序列生成
先前的研究已经展示了,通过预测语言模型(PLM)生成的蛋白质序列能够形成多种多样的结构,这些结构在序列上经常与已观察到的蛋白质有相当大的差异。为了评估ProGen2模型的生成能力,作者使用ProGen2-xlarge模型生成了6757个序列。每个序列的三维结构都是使用ESMFold预测的。对于每个结构,作者使用Foldseek找到了PDB(蛋白质数据库)中最结构相似的自然蛋白质。在图2A中,作者展示了结构与自然蛋白质相似性(TMscore)与ESMFold预测置信度(pLDDT)之间的关系。大多数结构都被自信地预测了(中位pLDDT为85.2)并且在PDB中有结构类似的同源物(中位TMscore为0.89)。尽管生成的序列经常采用之前观察到的折叠方式,但它们与自然蛋白质的序列相似度很低(图2B)。在图2C中,作者展示了一个采用超螺旋折叠的生成序列。PDB中最接近的结构同源物是一个应激诱导蛋白的中央域(PDB ID 3GCO-A),它与预测结构的一部分对齐。尽管采用几乎相同的折叠方式,生成的蛋白质和自然蛋白质之间的序列相似度仅为13.3%。对于另一个预测为β卷结构的生成序列(图2D),最接近的结构匹配是一个具有截短的β折叠的异构酶(TMscore 0.579)。在图2E中,作者展示了一个具有环状结构的生成蛋白质。这种蛋白质包含与真核生物起始因子(PDB ID:3WJ9)相似的结构元素,但形成了直径更大的三级结构。总的来说,这些例子展示了ProGen2生成的序列的一些独特属性。
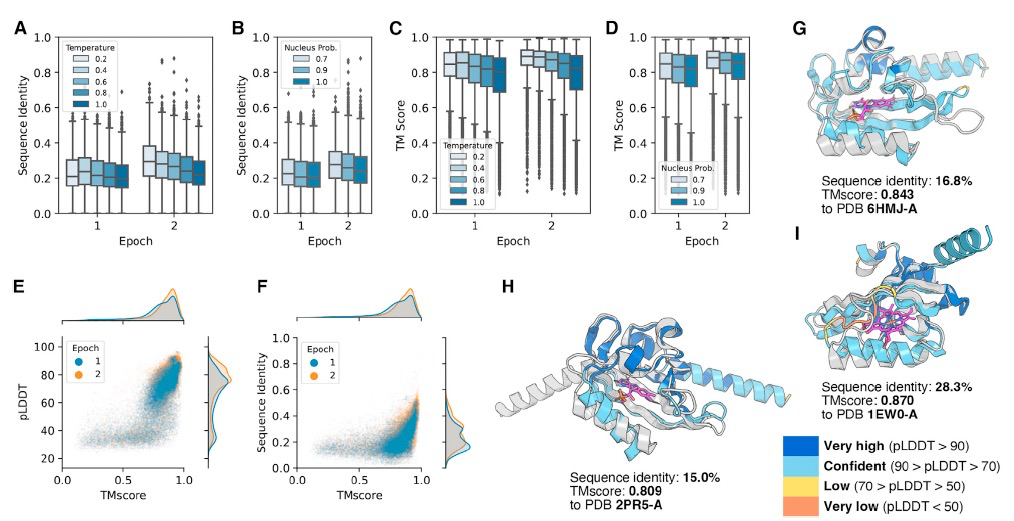
图 3
接下来,作者考虑了从在具有共同结构架构的蛋白质序列上微调的模型中生成序列。为了评估采样参数对共同架构内结构多样性的影响,作者使用ESMFold预测了所有60,000个序列的结构,并使用Foldseek计算了与PDB中最相似结构的TMscores和序列。作者选择验证PDB中的序列和结构相似性,以评估ProGen2生成采用自然折叠的多样化序列的能力。对于这两个指标,可以观察到随着微调的延长,与自然蛋白质的相似性增加(图3A-3D)。在相同模型检查点生成的序列中,采样参数与序列新颖性(即较高的采样温度或核心概率产生较低的序列身份)强烈相关,如图3A和3B所示。当比较结构多样性时,出现了类似的趋势,更严格的采样参数通常产生更接近自然蛋白质的结构(图3C和3D)。接下来,作者使用Foldseek分析了根据序列和与PDB中最接近结构的TMscore生成的双层夹心蛋白质的多样性。绝大多数序列被自信地预测为采用类似于自然蛋白质的结构(中位TMscore为0.85,图3E)。与预训练模型生成的序列一样,尽管序列有相当大的不同,这些序列仍然折叠成这些结构(图3F)。在更多新颖结构中,多样性的主要来源是配体结合区域,而非结合区域则类似于自然蛋白质(图3G-3I)。
零样本适应性预测
零样本适应性预测是一种没有额外训练的模型预测方法。理想情况下,蛋白质序列设计的模型应该能学习到与我们期望的功能特性相匹配的特征。通常,实验室里的实验可以帮助我们收集一些蛋白质样本,这些样本将特定的序列与其功能(比如“适应性”)联系起来。作者研究了这些实验测量的适应性与模型生成可能性之间的关系。简单来说,这种方法不需要在监督学习环境中进行额外的微调。
作者发现,当模型用于预测适应性时,即使模型规模更大,也并不一定能在所谓的“狭窄的适应性景观”(即只考虑很少变化的环境)上有更好的预测表现。为了比较,作者首先看了一些专注于单一替换的深度突变扫描实验的研究,这些实验是用来评估模型在未经特殊训练时的表现。作者发现,即使是规模较小的ProGen2模型,也展示了在这些零样本任务上更高的平均表现,这强调了预训练数据分布的重要性。与此相反,ProGen2模型的训练数据是一个混合体,包括来自不同来源的序列。作者还比较了两个在不同程度宏基因组序列冗余性上训练的ProGen2模型。结果显示,包含更多宏基因组序列的模型在狭窄景观的适应性预测性能上略有提高。作者的最佳ProGen2模型在各种不同建模策略的基线上表现更好或相当。这突显了了解用于训练模型的序列集合的重要性。最终,通过平均不同ProGen2模型的分数,形成的模型组合比任何单个模型都更有效。作者发现,随着模型容量的增加,零样本适应性预测的性能在某个点达到峰值,然后随着模型规模的进一步增大而下降。这与模型困惑度(一种衡量模型性能的指标)的趋势相反,后者随着模型规模的增大而持续改善。这表明,选择合适大小的模型而不是完全匹配数据分布可能更有助于改善预测适应性的表现。这种效应不仅适用于单一蛋白质家族数据集,也适用于包含进化上多样蛋白质的大型数据集。
虽然更大的模型不一定总是在无需额外训练的适应性预测方面表现更好,但在某些特定情况下它们可能还是有优势的。作者比较的大多数适应性测试主要集中在研究较多的蛋白质上,这些蛋白质有很多进化上相近的序列。直观地说,在蛋白质序列中那些比较罕见的分布区域,用较小的模型可能很难准确描述这部分的生成概率;所以,在这些区域,预测适应性和生成新蛋白质序列可能都不太准确。实际上,作者发现一些证据显示,在更广泛的适应性范围中,大型模型在预测适应性方面比小型模型表现得更好,特别是当新生成的蛋白质序列与自然界中的任何序列相差较远时。
参考资料
Nijkamp, E., Ruffolo, J.A., Weinstein, E.N., Naik, N. and Madani, A., 2023. ProGen2: exploring the boundaries of protein language models. Cell Systems, 14(11), pp.968-978.




 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง





