编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Jeffrey J. Gray团队的一篇论文。用于治疗应用的单克隆抗体的发现和优化依赖于大型序列库,但是由于可开发性问题而受到阻碍。基于数百万蛋白质序列训练的生成式语言模型,是按需生成现实且多样化序列的强大工具。作者提出了免疫球蛋白语言模型(IgLM),这是一种用于创建合成抗体库的深度生成式语言模型。与之前利用单向上下文进行序列生成的方法相比,IgLM基于自然语言中的文本填充来构建抗体设计,允许它使用双向上下文重新设计抗体序列中的可变长度区域。

由于多样性和高特异性地结合抗原的能力,抗体已成为治疗学上的热门选择。传统上,单克隆抗体(mAbs)是通过杂交瘤技术获得的,这需要动物的免疫接种,或是通过转基因动物系统获得,这涉及将人类免疫位点整合到其他物种(例如,小鼠)中。1985年,噬菌体展示技术的开发允许从大型抗体库中体外筛选特异性高亲和力的mAbs。尽管有这些进展 mAbs仍面临可开发性问题,如表达差、溶解度低、热稳定性差等问题。目前技术依赖于作为起点的高质量和多样化的抗体库,以分离出更易于开发的高亲和力抗体。合成抗体库是通过将合成DNA引入定义互补决定区(CDRs)的抗体序列区域而准备的,允许制造人造抗原结合位点。为了发现高亲和力抗体,必须构建达到10^10至10^11变体的大规模合成库。然而,可能的合成抗体序列空间非常大(多样化CDR的10个位置产生20^10至10^13的可能变体),这意味着这些方法仍然大大低估了序列的可能空间。此外,随机化库中的序列通常包含大量非功能性抗体。通过限制库仅包含类似自然抗体的序列,因此更有可能成为可行的治疗药物,可以减少这些负面影响。在这里,作者介绍了免疫球蛋白语言模型(IgLM),一种利用双向上下文设计不同长度抗体序列跨度的生成式语言模型,同时在大规模自然抗体数据集上训练。作者展示了IgLM可以根据链类型和起源物种生成完整长度的抗体序列。此外,IgLM可以在样本抗体上多样化生成,以生成表现出有利的预测生物物理特性同时类似于人类抗体的高质量库。IgLM是抗体发现和优化的强大工具。
IgLM模型部分
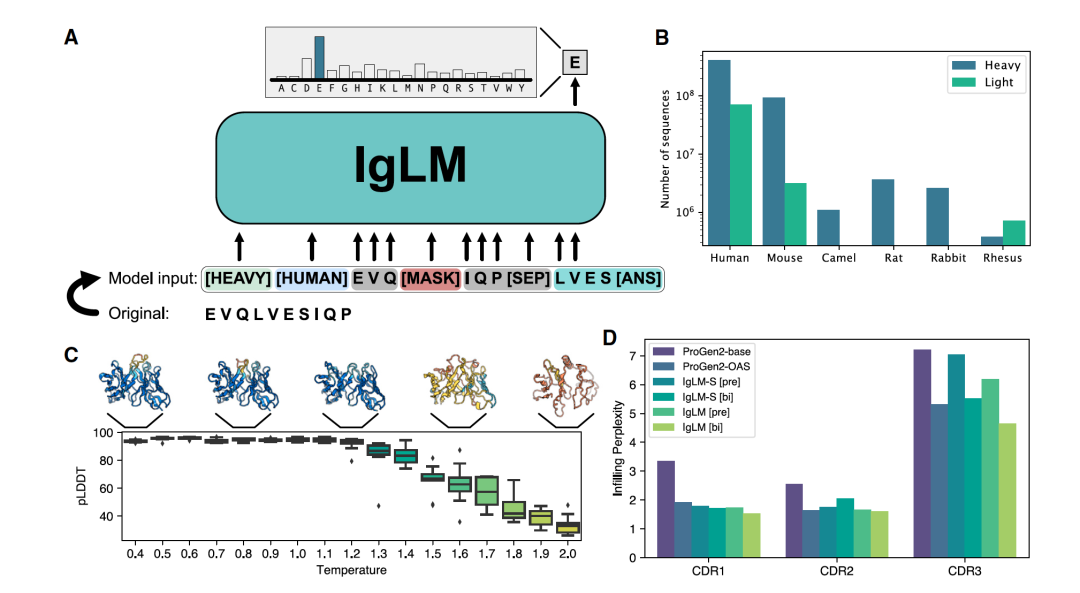
图 1
抗体库的设计通常侧重于多样化互补决定区(CDR)环序列,以便结合到多种抗原上。通过传统的多样化技术,可以产生许多假定的抗体序列,并经过实验筛选,从而发现或优化特定抗体。然而,这样的技术通常产生大量非活性或表现不佳的序列,因为它们没有限制在自然抗体序列的范围内。蛋白质序列的生成模型,例如语言模型,提供了一种高效采样自然蛋白质空间以产生大量序列库的替代方法。然而,现有的蛋白质序列生成方法(包括抗体)通常采用从左到右的解码策略。尽管这些模型在生成多样且功能性序列方面已被证明有效,但它们不适合重新设计蛋白质内的特定段落。为了解决这个局限性,作者开发了IgLM,一种用于免疫球蛋白序列的填充式语言模型。IgLM使用基于GPT-2的标准从左到右解码器仅变换器架构,但它经过填充式重排序列的训练。具体来说,作者采用了自然语言处理中的填充式语言模型公式,即在训练期间将任意长度的序列段(跨度)掩盖,并附加到序列的末尾。通过在这些重排序列上的训练,模型学会在周围序列上下文的条件下预测掩盖的跨度。为了训练IgLM,作者从OAS数据库中收集了抗体序列。OAS数据库包含来自六个物种(人类、小鼠、大鼠、兔子、恒河猴和骆驼)的自然抗体序列。在训练期间,作者随机掩盖抗体序列内的十到二十个残基,以便在推理过程中实现任意跨度的多样化。此外,作者根据链类型(重链或轻链)和起源物种对序列进行了条件化。提供这种上下文使得能够控制地生成特定物种的抗体序列。训练数据构建的一个示例如图1A所示。
IgLM生成可折叠的抗体序列
作为对IgLM抗体序列生成能力的初步验证,作者进行了小规模的调查。具体来说,作者研究了采样温度参数对生成序列多样性调节的影响。高于1的采样温度值有效地使每个生成步骤中的氨基酸分布平坦化,导致序列更加多样化,而低于1的温度则使每个位置的分布更加尖锐,类似于贪婪解码策略。我们在0.4到2.0的温度范围内生成了一组全长序列,为模型提供了人类重链和轻链的条件标签。由于IgLM是针对序列填充训练的,生成的序列包含不连续的序列段,作者简单地重新排序以产生全长抗体。重链和轻链序列是独立生成的,因为IgLM只考虑单链。然后根据采样温度将序列配对,并使用AlphaFold-Multimer预测它们的结构。一般来说,IgLM在较低温度下(最高到1.2)生成结构预测相对有信心的序列,在更高温度时开始降低质量(图1C)。对于后续实验,作者设置采样时最高温度为1.2,以保持在可折叠抗体空间内,并使用更快的IgFold模型进行高通量结构预测。
基于条件的抗体设计
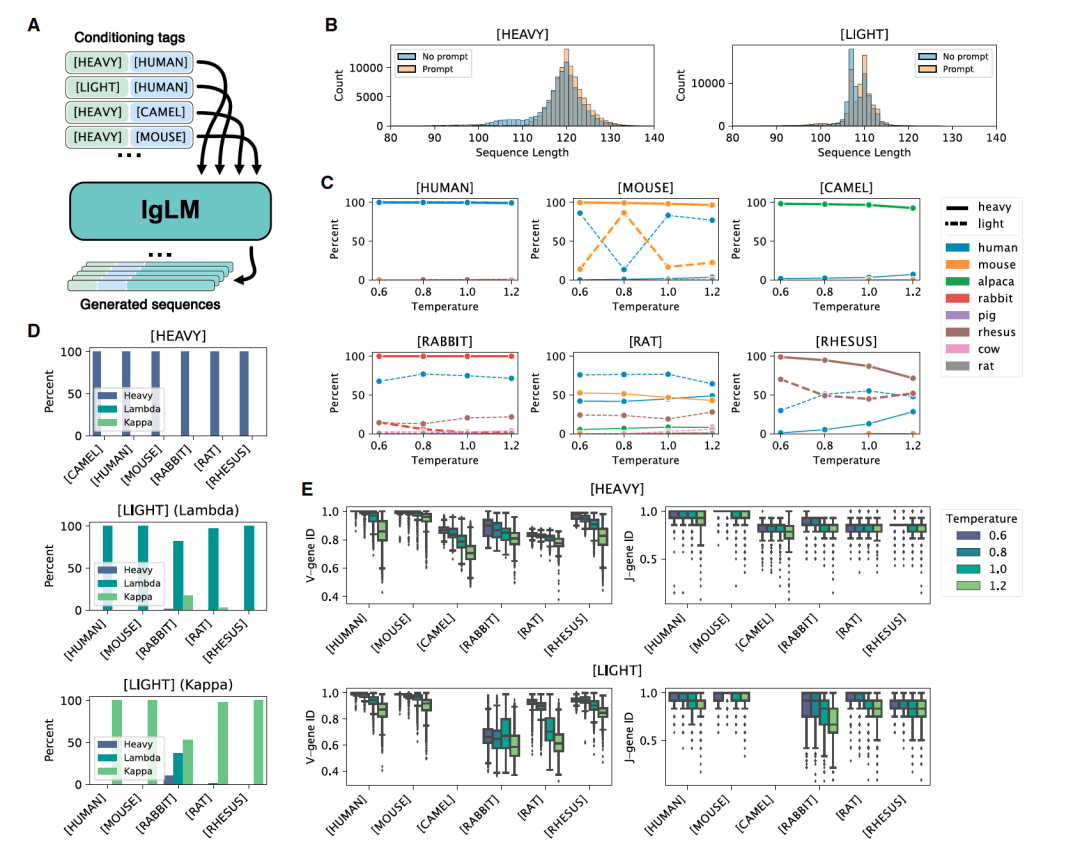
图 2
为了评估IgLM的可控性,作者使用所有可行的条件标签组合以及一系列采样温度,生成了220,000个全长序列(图2A)。对于每个物种(除了骆驼),作者都使用重链和轻链的条件标签进行了采样。对于骆驼序列的生成只采样重链,因为它们不产生轻链。为了产生一组多样化的序列进行分析,作者使用了一系列温度进行采样(T ∈{0.6; 0.8; 1.0; 1.2})。在这些条件下的采样产生了一组多样的抗体序列。为了评估可控生成的有效性,作者考虑了提供的条件标签与IgLM生成的序列之间的一致性。对于每个生成的序列,作者使用ANARCI对物种和链类型进行了分类。在图2C中,作者根据采样温度展示了每个物种条件标签的序列构成。在每个图中,按物种分类的重链和轻链序列的百分比分别用实线和虚线表示。对于大多数物种(人类、小鼠、骆驼、兔子和恒河猴),IgLM能够在每个温度下成功生成重链序列。唯一的例外是大鼠序列,模型无法生成ANARCI分类为预期物种的任何序列。生成序列的能力并不直接由训练数据集中的普遍性解释,因为该模型在训练时接触到的大鼠重链序列比恒河猴多一个数量级。IgLM在生成大多数物种的轻链序列方面通常效果较差。除了人类轻链外,所有物种都有大量被分类为非预期物种的序列。接下来,作者评估了IgLM生成序列对链类型条件标签的遵守程度。在图2D中,作者展示了ANARCI分类为重链或轻链的序列百分比。轻链进一步分为λ和κ类。当条件化为重链生成时,IgLM有效地为所有物种产生重链。对于轻链观察到类似的趋势,IgLM为所有物种主要产生轻链序列。
生成抗体的多样化
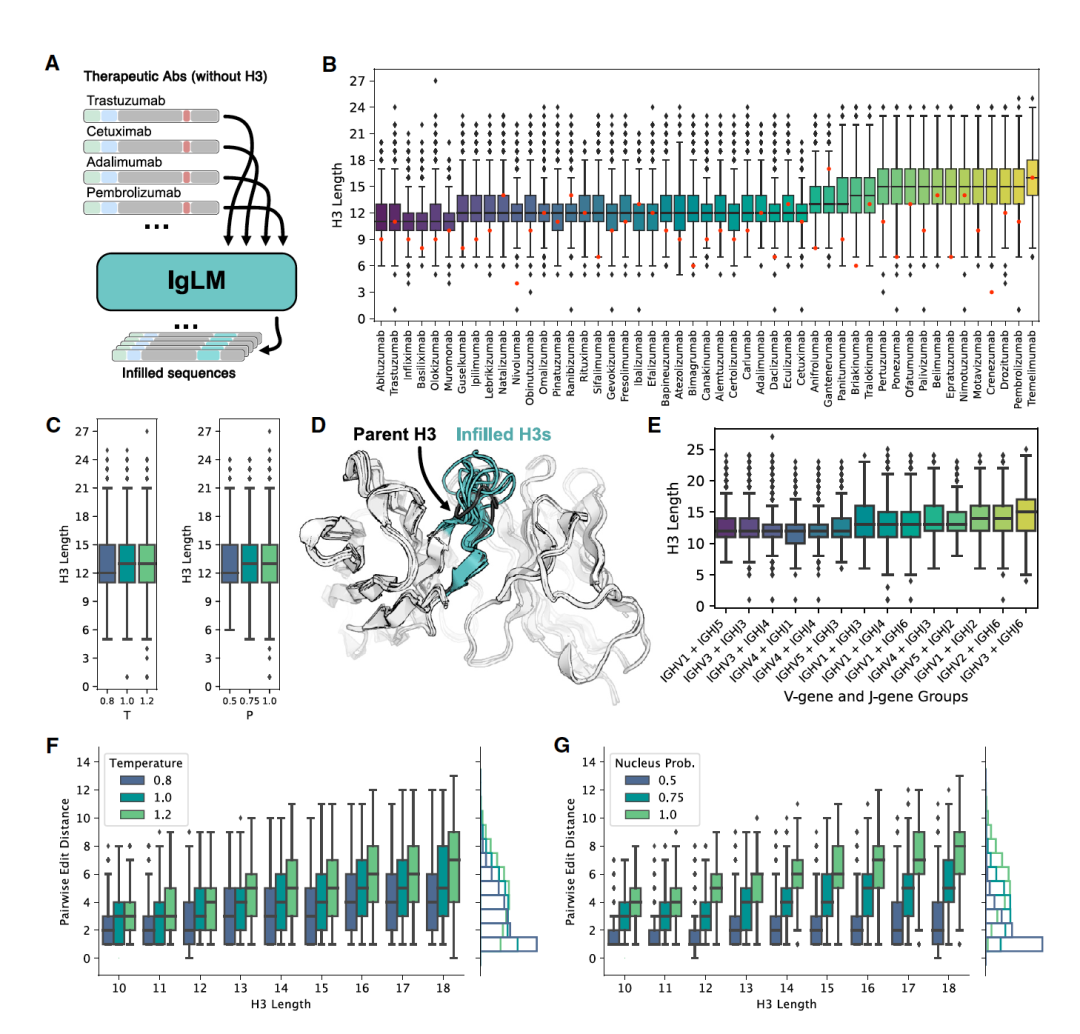
图 3
为了评估IgLM用于多样化抗体序列的填充方法的实用性,作者为来自Thera-SAbDab数据集的49种治疗性抗体创建了填充式库。这些抗体被选中是因为它们具有实验确定的结构,并且之前已经进行了可开发性筛选。对于每种抗体,作者根据Chothia定义移除了CDR H3环,并使用IgLM生成了一系列填充序列(图3A)。为了产生多样化的序列,作者使用了一组采样温度(T ∈{0.8; 1.0; 1.2})和核采样概率(P ∈ {0.5; 0.75; 1.0})。核采样有效地在采样过程中的每个位置剪辑概率分布,使得只有最可能的氨基酸被考虑。对于这49种治疗性抗体中的每一种,作者为每个T和P的组合生成了1,000个填充序列,每个亲本抗体总共有9,000个变体。在图3D中,作者展示了一部分来自trastuzumab抗体的十个填充环的预测结构(使用IgFold)。填充环在长度上变化并采取不同的结构构型。在填充库中,作者发现了不同亲本抗体的多种CDR H3环长度(图3B)。跨越抗体的填充环的中位长度从11到16个残基不等。作者观察到在变化采样温度和核概率时,填充环长度上的影响很小(图3C)。填充环长度的分布在这49种治疗性抗体上变化显著。因为IgLM是在自然抗体序列上训练的,作者假设模型可能在进行某种匹配,即具有相似基因的序列导致相似的环长度分布。为了测试这一点,作者使用ANARCI为每种抗体确定了最接近的始祖基因。然后根据共有的V-和J-基因组将亲本抗体分组,并比较了每组填充环长度的分布(图3E)。尽管可能存在相似的V-和J-基因导致相似填充环长度分布的趋势,但作者观察到了相当大的变化。这表明IgLM不是纯粹进行始祖匹配,而是考虑了亲本抗体的其他属性。
为了发现或优化针对抗原靶标的序列,多样化的环库是必不可少的。作者根据环长度和采样参数的选择比较了序列的多样性(图3F和3G)。通常可以观察到生成的环在更长的长度上更加多样化,这符合预期,因为随着更多残基的添加,可用的组合复杂性增加。增加采样温度和核概率会导致序列多样性的增加。实际上,随着核概率的增加,成对距离的边际分布产生了更大的变化(图3G),这比温度的变化更显著(图3F)。在实践中,采样参数的组合可能适合产生高可能性(低温度和低核概率)和多样化序列的平衡。
参考资料
Shuai, R. W., Ruffolo, J. A., & Gray, J. J. (2023). IgLM: Infilling language modeling for antibody sequence design. Cell Systems.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


