
点击蓝字 关注我们

微末生物
Nanomega BioAI
2021年,来自DeepMind的AlphaFold以其精准到“原子级”的蛋白质三维结构预测结果,震惊了生物界。一时间,这个人工智能算法被誉为结构生物学领域的“无人驾驶飞机”,有望在不久的将来完成所有已知蛋白质的三维结构数据库。
然而,就在AlphaFold引发热潮的同时,科学界也开始反思和审视这个“尚未完工”的结构预测工具。AlphaFold的预测质量究竟如何?它的优劣势体现在哪里?实验工作者该如何正确使用AlphaFold的预测结果?一系列疑问逐渐浮出水面。
为回答这些关键性问题,最近《Nature Methods》刊登了一项针对AlphaFold预测质量的重要评估研究。该项工作系统校验了AlphaFold与实验结果之间的差异,呈现了这款炒作已久的算法更为立体和全面的画像,为结构生物学领域的实验工作者正确解析和应用AlphaFold预测提供了宝贵建议。
惊艳水准的蛋白质结构预测工具
近年来,伴随着计算机技术和算法不断进步,蛋白质三维结构的计算机预测也日益精确。尤其是2021年AlphaFold诞生后,预报的蛋白质主链原子坐标误差中值降至了1.1Å,侧链原子也接近1.5Å,已达到了实验X射线晶体学最高分辨率的程度。
相比之下,此前主流的评估平台CASP中报告的最好结果误差中值也只有2-3Å。可以说,AlphaFold的出现标志着蛋白质结构预测进入了一个崭新的发展阶段。它基于蛋白序列本身特征以及进化共变关系,高效预测三维空间折叠,被普遍认为是一大突破性进展。
尽管预测误差不到2Å听起来十分微小,但这在生物大分子中却可能导致功能和相互作用发生巨大变化。因此,准确评估AlphaFold的预测质量至为重要。很多研究仅仅将AlphaFold预测与已有的蛋白质数据库(PDB)结构进行比较。但是这些实验模型本身也存在误差或局限性。
这项研究的创新之处在于,研究团队直接采用了X射线衍射实验本身的原始电子密度图作为“金标准”,检查AlphaFold的预测结构是否与这些实验数据吻合。这种基于实验事实的校验,使得他们得出的关于AlphaFold的结论也更具有说服力。
结构预测高度吻合,但“漏洞百出”
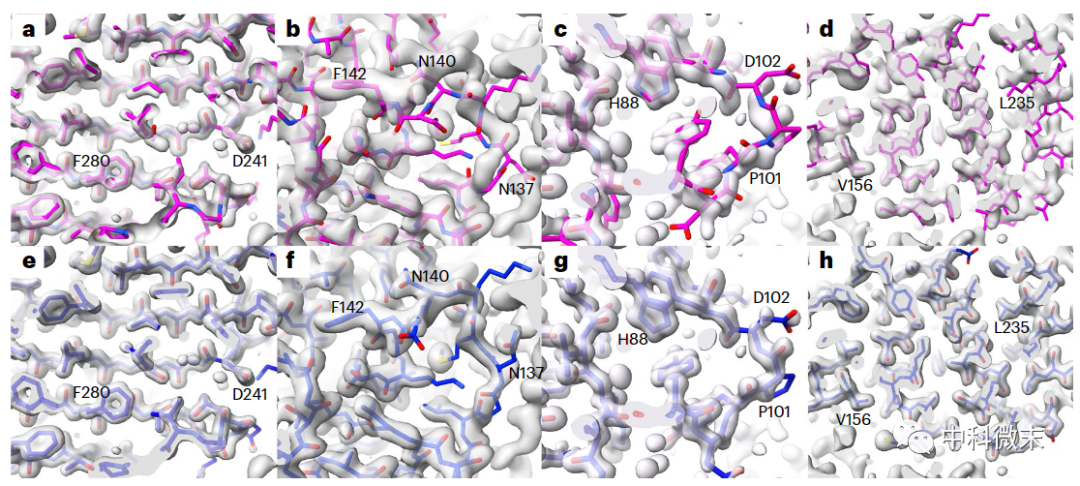
图1 部分Alphafold预测结果与实验所得密度图“驴唇不对马嘴”
研究团队分析了102个高分辨率的蛋白质晶体衍射电子密度图,与相应的AlphaFold预测进行对比。结果发现,AlphaFold对许多蛋白质结构的预测高度准确,与实验电子密度图吻合非常紧密,就像“量体裁衣”。
但他们也观察到了不少案例,AlphaFold预测在整体结构上存在失真现象,或是局部氨基酸主链和侧链取向与实验结果明显不符,尽管这些部位的预测置信度很高。总的来说,AlphaFold预测与对应PDB实验模型之间的Cα原子坐标差异的中位数约为1Å;而相同组分但在不同空间群晶体结构中获得的高分辨率结构模型之间的Cα原子坐标差异中位数仅为0.6Å。以统计中位数为标准,从整体主链原子的吻合程度判断,AlphaFold的预测不如多个实验模型之间的互相匹配,预测结果与实验事实仍存在一定差距。
具体到局部结构,研究人员通过一种“移植”操作,单独检查AlphaFold预测的侧链取向是否符合实验电子密度分布。结果发现,20%的高置信度侧链与实验结构差异较大,其中1/3与实验数据完全不兼容。AlphaFold预测结果与实验模型之间的差异,比相同蛋白质但在不同晶体空间群条件下获得的实验结构之间的差异要大得多。具体而言:对于相同蛋白质的不同晶体结构之间,只有6%侧链有较大差异,2%不兼容。由此可以看出,AlphaFold预测结果与实验模型之间的差异远超过实验本身的误差范围,不能简单归因于蛋白质的内在弹性或各向同性。
值得一提的是,研究人员还特别分析了文献报道的关键功能位点预测情况,结果与所有位点的整体水平类似,7%与实验数据不符,未见明显的提升或下降。
综上所述,尽管AlphaFold实现了前所未有的结构预测突破,但距离“完美”还存在一定差距,许多预测细节仍有待进一步优化和实验验证,不能简单等同于实验结果。预测与实验结果之间的差异部分,很可能反映了AlphaFold本身还未考虑到的生物学因素,如蛋白质的相互作用、配体结合和翻译后修饰等情况下的构象变化。
置信度评分预测 实验验证仍不可或缺
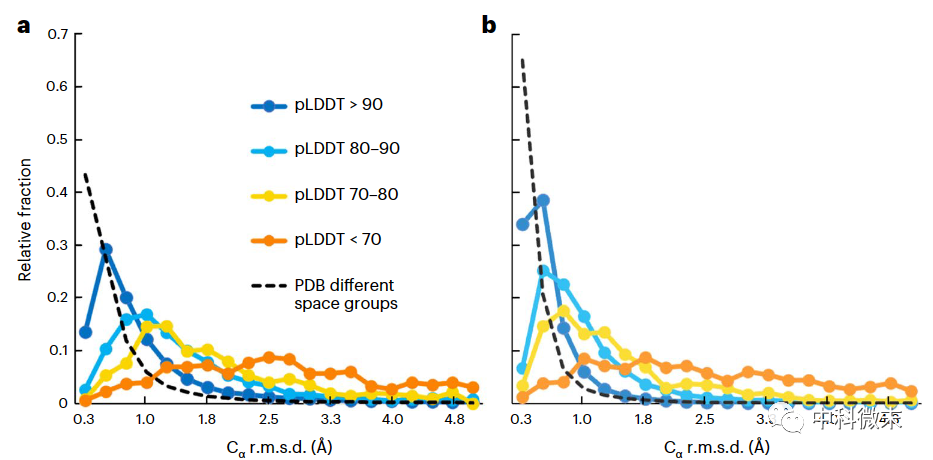
图2 理解AlphaFold预测的准确性限制。pLDDT越大,准确度越高,但高置信区间内也有10%的结果误差很大;进行预测结果调整后再比较(b图)中位数误差有所下降,但仍高于实验本身误差(虚线)。总的而言,pLDDT能反映误差总体水平,单个情况下仍可能存在很大偏差,置信度高也不意味完全准确。
鉴于AlphaFold预测的准确性参差不齐,如何评估单个结构预测的置信水平就尤为关键。幸运的是,AlphaFold算法本身提供了每一个氨基酸残基的预测置信度评分“pLDDT”。DeepMind团队对此指标进行了独立验证,证明其是对实际模型准确度的无偏估计。
研究团队根据这个评分,统计了不同置信区间内AlphaFold预测误差的中值和分布。他们发现,当pLDDT大于90时,表示预测非常可靠,Cα原子坐标误差中值为0.6Å,约90%的情况下小于2Å;而pLDDT在80-90之间,误差中值约为1.1Å;70-80之间则是1.5Å。这证实了pLDDT可以作为一个有效的指示器,为实验科学家快速评估预测质量提供参考。当然,由于分布非常不对称,中值并不能代表全部情况,用户在应用时也应注意个例中可能出现的极大偏差。
总的来说,AlphaFold预测包括了许多高度精确的局部特征,是很有价值的结构假说,能加速后续的实验验证和结构优化。但对于功能关键位点或小分子与蛋白质复合物的具体相互作用来说,直接应用预测结果仍有一定风险。这些情况下,传统的实验手段例如冷冻电镜,X射线晶体学等仍然发挥必要的补充作用。
中科微末,作为深耕于结构生物学和AI领域的企业,已充分认识到提供优质结构解析服务的重要意义。中科微末依托自主研发的冷冻电镜技术,专注为生物医药企业提供结构生物学研究服务,实现目标蛋白原子级高分辨率的结构解析。团队成员累积发表400+顶级SCI期刊论文,团队手握多项专利软著,在冷冻电镜技术工作流每个环节累积了大量独家算法和专利,可以提供“高难度结构有解决方案,低难度结构更迅速价格更便宜”的技术服务。展望未来,中科微末将继续致力于冷冻电子显微技术的研发和创新,持续推出更多切合客户需求的结构解析解决方案。
微末生物
Nanomega BioAI

请关注微末生物
期待与您交流



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


