
12 月 14 日,美国临床阶段生物公司 Deep Apple Therapeutics ,宣布从美国生命科学风投机构 Apple Tree Partners (ATP) 获得 5200 万美元A轮融资。
Deep Apple 由 ATP 创建和孵化,通过人工智能生成的虚拟库的虚拟筛选,快速发现针对高价值靶标的新型小分子疗法。
Deep Apple 的成立是为了开发一种发现引擎,该引擎结合了整体冷冻电镜、深度学习和超大型文库的分子对接屏幕,可以在不到 12 个月的时间内从目标识别到先导化合物优化(仅占行业标准时间的一小部分),并且可以实现传统发现方法无法达到的生物靶标信号传导。 发现引擎
发现引擎
Deep Apple 的发现引擎广泛适用于疾病领域,特别适合针对整合膜蛋白的快速命中发现。该公司目前正在推进多个专注于 GPCR 调节剂的项目,这是一种经过验证的目标类别,可应用于代谢紊乱、炎症、免疫学和内分泌疾病。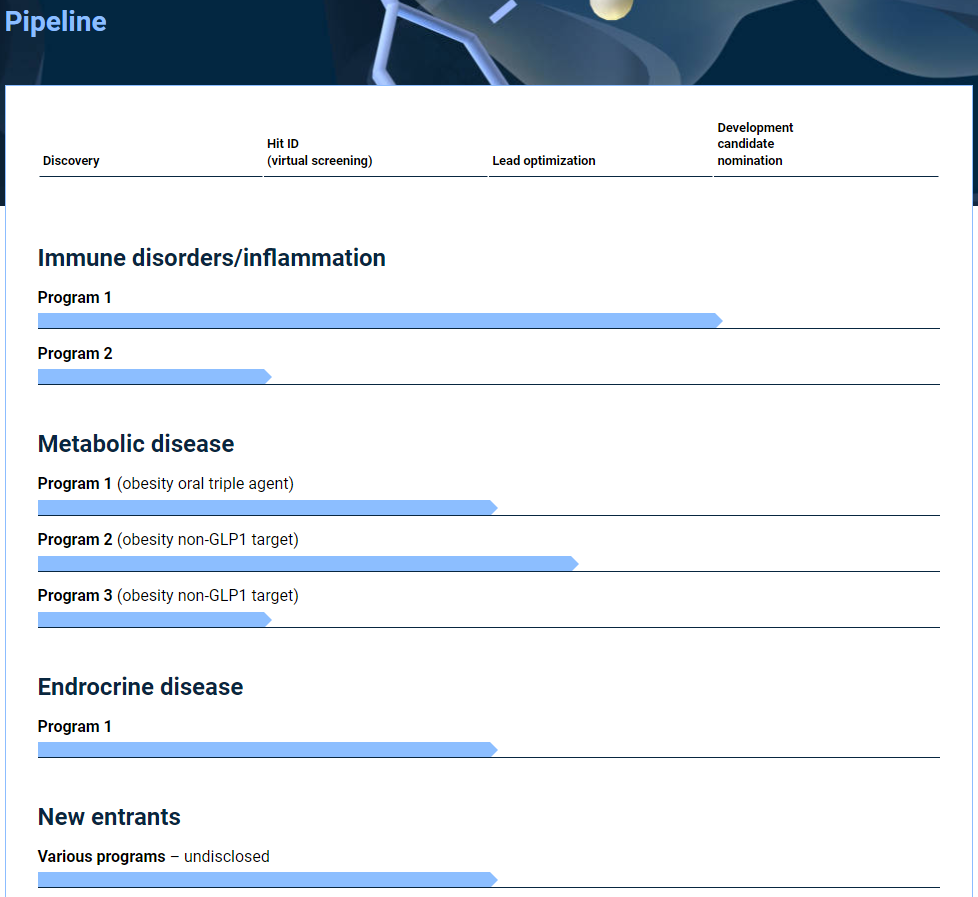
Deep Apple 的药物发现引擎建立在其学术联合创始人的领先专业知识和技术之上:
Georgios Skiniotis 博士,斯坦福大学,冷冻电镜和 GPCR 结构生物学领域的世界领先者;Brian Shoichet 博士,来自加州大学圣何塞分校 Francisco (UCSF),虚拟放映先驱;John Irwin 博士,加州大学旧金山分校 (UCSF) 的计算图书馆权威,他创建了广泛使用的 ZINC free包含超过 100 亿种可合成化合物的虚拟库。
Deep Apple 的团队由 20 多人组成,其中包括一群具有分子动力学和模拟经验的计算化学家和机器学习工程师。药物发现负责人是一个例外,他是一名药物化学家。他们还正在建立一支生物学家团队,利用内部设计的检测方法进行体外药理学。 冷冻电镜
冷冻电镜
Deep Apple 创始人兼 ATP 风险合伙人 Spiros Liras 表示,原始的二维冷冻电镜通常会被人眼过滤,导致大部分数据被丢弃。Deep Apple正在利用深度机器学习来分析所有原始的二维冷冻EM数据,以加速创建质量更好的3D地图,并提取与构象状态有关的动力学,例如它们是稳定的还是短暂的。
在这些数据的帮助下,他们可以通过他们的内部虚拟文库Orchard.ai来识别似乎稳定所需蛋白质的神秘或短暂的口袋,该文库主要由他们针对GPCR亚家族开发的模型衍生的新化合物组成。为了选择和优先考虑化合物,Deep Apple创建了一种专有的基于对接的大规模评分算法。
Orchard.ai™ 是 Deep Apple 的虚拟库扩展工具,用于机器学习,可加速命中发现。Orchard.ai 生成项目特定的专有化学库,这些库>针对 Zinc22 的 95% 新颖性,显着提高了针对感兴趣靶标的码头评分,并提高了具有高合成易处理性的命中的药物相似性。
Liras说,创建Orchard.ai有几个原因。一个原因是,预先存在的虚拟库,如ZINC虚拟库——一个用于虚拟筛选的商业可用化合物的免费数据库——在寻找生物活性分子类似物的过程中,可以作为快速、合理价格合成和测试的模型。Deep Apple只有在其计算方法使他们相信它具有某种内在的生物学价值时才能生产出一种化合物,而不是生产所有的化合物进行物理筛选。
尽管应用广泛,但Deep Apple的平台最适合靶向任何完整的膜蛋白,包括受体、转运蛋白和离子通道。Deep Apple将其平台专注于GPCR,部分原因是其创始人的专业知识,以及有很多机会。
该公司正在推进多个专注于GPCR调节剂的项目,这是一个已被证实的目标类别,应用于代谢紊乱、炎症、免疫学和内分泌疾病。
但该团队仍处于药物发现过程的早期阶段,共有七个靶点。Liras表示,他们希望在2024年第二季度推出首个针对炎症靶点的临床候选药物。Deep Apple希望在2025年初增加更多的临床候选药物,并在与减肥管理有关的代谢疾病方面有几个目标。






 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


