使用AlphaFold、RoseTTAFold和相关方法预测蛋白质结构的准确性大大优于前几代预测算法,这使人们对大分子的三维结构的理解更接近于实际。随之而来的一个问题是,这些预测能够在多大程度上替代实验结构测定?
2023年11月30日,美国Los Alamos国家实验室的Thomas C. Terwilliger等人在Nat Methods杂志发表文章AlphaFold predictions are valuable hypotheses and accelerate but do not replace experimental structure determination,从多个角度考察了AlphaFold预测结果的准确性。

评估蛋白质结构预测结果准确性的方法
基于人工智能的蛋白质结构预测方法已经彻底改变了结构生物学。然而事实上,无论是实验确定的蛋白质结构还是预测的模型,都有相应的局限性。
蛋白质是柔性和动态的,其构象分布取决于温度、溶液条件和配体或其他蛋白质的结合(包括晶体学中的晶体接触)。高分辨率的晶体结构可以准确地表示特定环境中晶体中存在的主要构象,但有时可能存在问题(如结晶假象问题,参见The advantages and limitations of protein crystal structures. Trends Pharmacol. Sci. 26, 10–14, 2005)。
在许多情况下,基于人工智能的模型可以非常准确。然而,它们尚未考虑配体、共价修饰或环境因素的存在,并以有限的方式考虑蛋白质-蛋白质相互作用和多种构象。
评估预测结果准确性的标准,通常是看预测结果与蛋白质数据库(PDB)中相同序列结构的匹配程度。使用注重局部准确性的比较方法,AlphaFold预测结果被评估为具有”原子级别的准确性”,其准确性可与”最佳实验结果”相媲美,其质量可与实验晶体结构媲美。
实际情况到底如何?
本文通过将预测结果直接与实验晶体图进行比较,来评估AlphaFold预测在描述蛋白质结构方面的效果。
本文发现,在许多情况下,AlphaFold预测结果与实验图非常吻合。而在其他情况下,即使是置信度非常高的预测结果,也会因失真(distortion)和结构域定向而在全局范围内与实验图存在差异,并在骨架和侧链构象的局部范围内与实验图存在差异。
作者建议将AlphaFold预测视为非常有用的假设。另外,在解释AlphaFold预测时,必须考虑预测的置信度,并进行实验结构测定以验证结构。
将AlphaFold预测与密度图进行比较
作者首先使用一组晶体学电子密度图作为评估AlphaFold预测的标准。这些密度图是通过迭代AlphaFold预测和利用保存在PDB中的X射线晶体学数据重建模型得到的。
AlphaFold预测结果的残基特定的置信度(pLDDT,局部距离差检验的预测值)大于90的残基被认为具有极高的预测置信度,而pLDDT值大于或等于70的残基则具有中高置信度。
图1比较了AlphaFold预测结果、实验密度图和相应的蛋白质数据库中结构模型(预测结果叠加)。图1显示的所有残基的预测置信度都非常高(pLDDT > 90),密度图的分辨率从1.1 Å到1.6 Å不等。
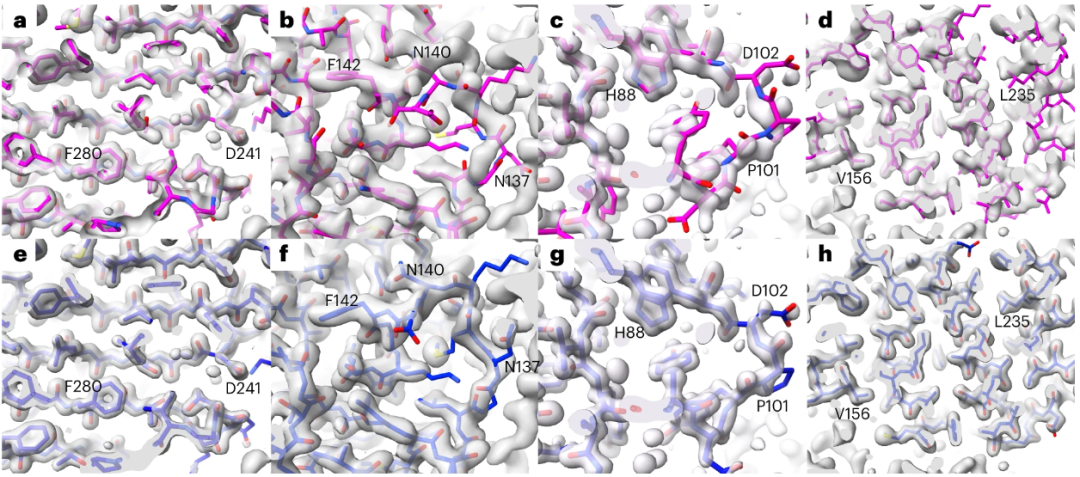
图 1:AlphaFold预测的细节与密度图的比较
图1a显示了AlphaFold预测与相应密度图紧密叠加的一个例子。图1b显示了PDB条目7s5L的预测结果,其中包含了与密度图不匹配的高置信度区域。图1c显示了一个预测结果与密度图不匹配的例子,但它可能仍然代表了分子的一种合理构象。图1d展示了AlphaFold预测构象相对于密度图(PDB条目7naz)发生失真的情况。
考察AlphaFold预测中的失真和域移动
图 1d 表明,相对于实际结构,AlphaFold预测可能会有些失真(Distortion)。为了确定AlphaFold预测是否都会出现这种情况,作者对每个AlphaFold预测进行了“变形”,使其与蛋白质数据库中结构模型更相似(此过程可减少由链内域的失真或交替位置引起的预测模型和蛋白质数据库中结构模型之间的差异)。
对每个预测模型进行变形后,预测结果与电子密度图更接近(图2)。
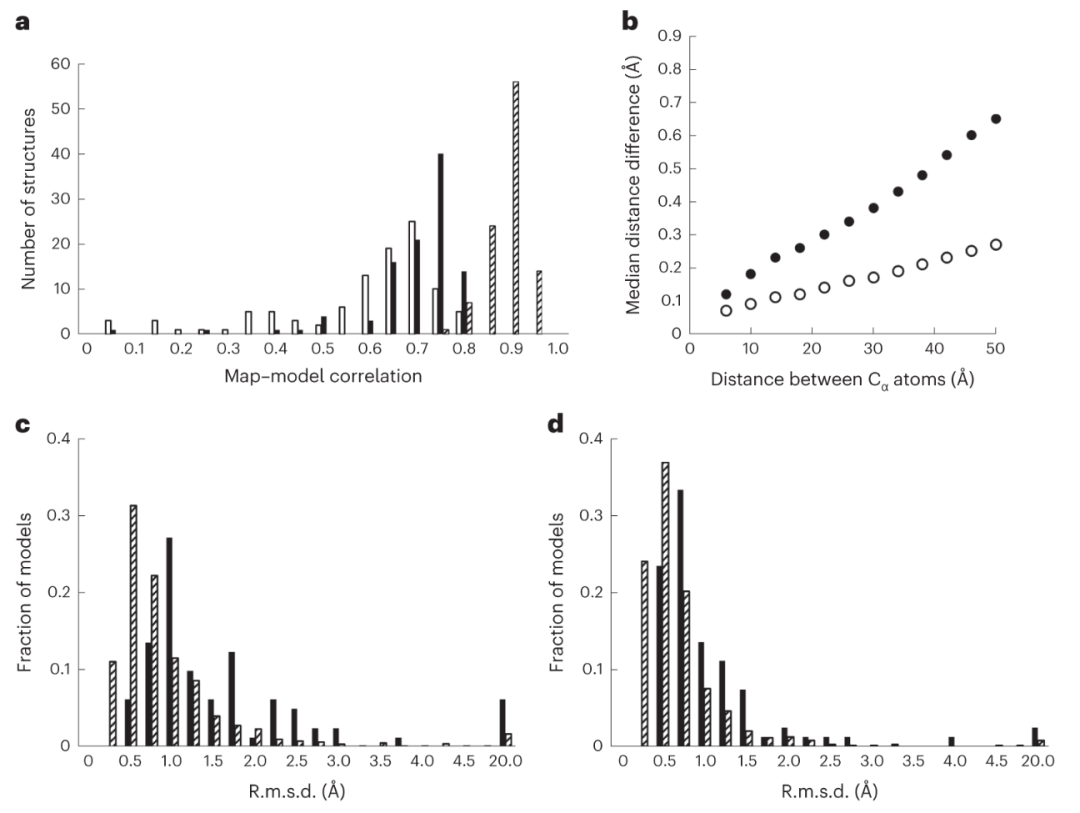
图2:AlphaFold预测与密度图和蛋白质数据库中结构模型的总体比较
图 2a显示了102个AlphaFold预测结果与相应密度图的整体兼容性,这是通过图-模型相关性来衡量的。
图2b显示,对于距离较近的原子对(相距4 Å至8 Å),蛋白质数据库中结构模型与AlphaFold预测的中高置信度部分之间的原子间距偏差中值约为0.1 Å,而对于距离较远的原子对(48 Å至52 Å),这一偏差中值则增加到0.7 Å,这表明在这一距离范围内,原子间距的典型失真约为0.5-1 Å。
图2c显示了AlphaFold预测的Cα r.m.s.d.值分布,r.m.s.d.的中值为1.0 Å。对于PDB中不同空间群结晶的匹配结构对,Cα r.m.s.d.的中位数仅为0.6 Å,而通过应用变形场(应用的r.m.s.变形中位数为 0.2 Å)可将其减小到0.4 Å。
总体而言,AlphaFold预测的Cα坐标与PDB条目的差异要远远大于同一分子在不同空间群中的高分辨率结构蛋白质数据库中结构之间的差异(中位数r.m.s.d.为1.0 Å,而PDB条目为0.6 Å),这种差异的很大一部分是由长程变形造成的。
AlphaFold侧链预测与实验图的比较
如图1所示,AlphaFold预测结果通常至少包含一些与蛋白质数据库中结构相似的区域,但即使在这些区域中,许多细节也往往存在差异。接下来,作者使用上述电子密度图和蛋白质数据库中结构模型来评估侧链构象(侧链中原子相对于其所连接的主链原子的位置)。
为了分析局部侧链结构并消除结构域偏移或扭曲的干扰效应,作者将AlphaFold预测中每个残基的侧链嫁接到蛋白质数据库中结构模型中相应的主链原子残基上。这样就得到了一个复合模型,其中包含了已存模型的主链坐标和与AlphaFold预测相对应的侧链构象。
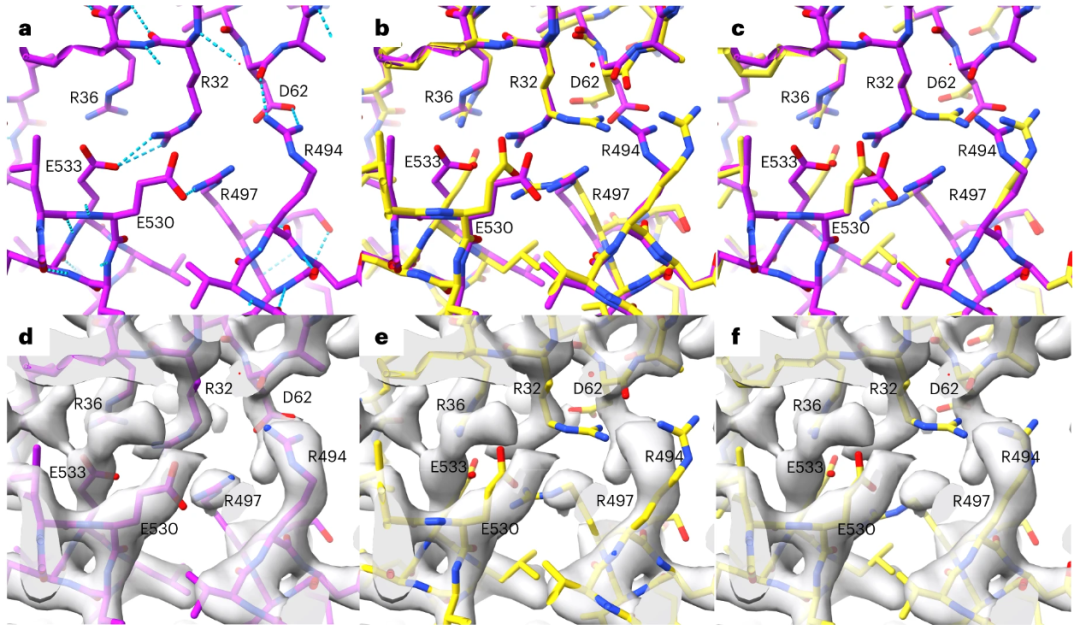
图3:AlphaFold侧链预测结果与PDB条目7vgm的密度图的比较
图3a显示了PDB条目7vgm的局部,图3b显示了AlphaFold预测与蛋白质数据库中结构模型的叠加。图3c显示了带有嫁接侧链和复合模型的同一区域。AlphaFold模型中几条侧链的位置(例如 R32、D62、E530、E533、R494)与蛋白质数据库中结构模型中的位置不同。图3d显示了7vgm的蛋白质数据库中结构模型和PDB条目7vgm的密度图。图3e显示了AlphaFold模型与同一密度图的叠加。
尽管密度图是通过AlphaFold预测获得的,并没有参考蛋白质数据库中结构模型,但蛋白质数据库中结构模型中的所有侧链都与密度图非常吻合。相反,AlphaFold预测中与蛋白质数据库中结构模型中不同的侧链在嫁接前(图3e)和嫁接后(图3f)都与密度图不匹配,这表明这些侧链构象很可能是不正确的。
使用置信度估计AlphaFold预测中的误差
由于AlphaFold预测可能与相应的实验模型有很大不同,因此估计这些预测的坐标不确定性的简单方法将很有用。作者将AlphaFold预测叠加在相应的蛋白质数据库中结构模型上,并确定C之间的距离α预测模型和蛋白质数据库中结构模型中的原子,以及预测C的置信度(pLDDT)α原子。
图4a显示了不同置信度范围内预测误差的分布。
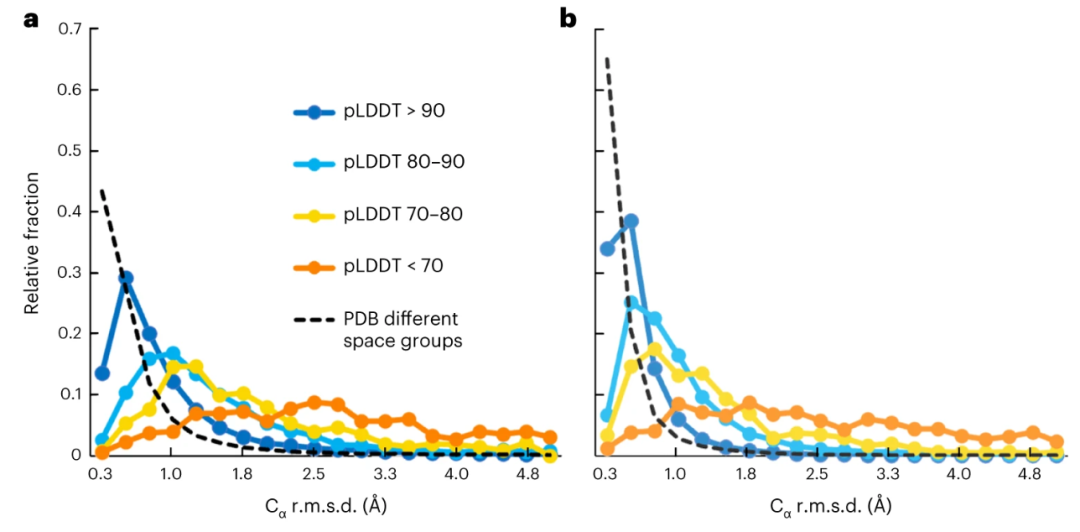
图 4:AlphaFold预测置信度范围内的预测误差分布
为便于比较,图4a中的虚线显示了含有相同成分但在不同空间群结晶的成对结构中匹配 Cα 原子间差异的分布。高置信度(pLDDT > 90)残基的预测误差中值为0.6 Å,而pLDDT在80和90之间的残基的预测误差中值为1.1 Å,pLDDT在70和80之间的残基的预测误差中值为1.5 Å(表 1)。
表 1 按 AlphaFold 置信度计算的预测误差中值和预测误差超过2 Å的百分比
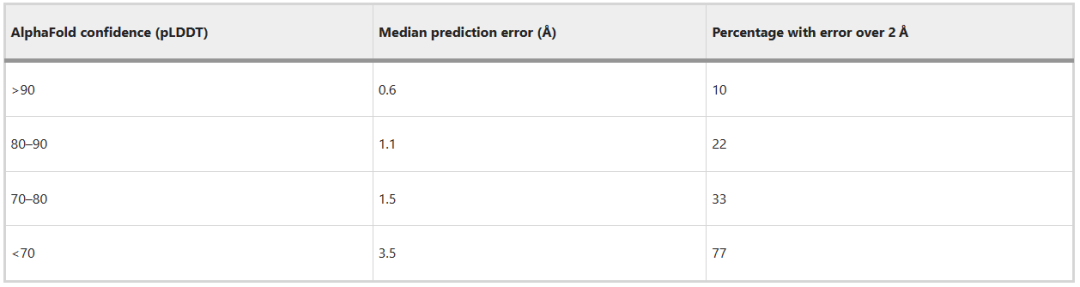
相比之下,不同空间群中成对结构的Cα原子匹配中值相差0.3 Å。图4b显示,按上述方法对每对结构中的一个成员进行变形可减少所有置信度范围内的差异,但PDB中成对结构之间的匹配差异也同样减少。
总的来说,这里发现的AlphaFold预测与实验数据之间的一致程度,与DeepMind在开发AlphaFold时进行的不确定性量化结果一致。
分析表明,估计模型准确度(pLDDT)是实际模型准确度(LDDT)的无偏预测指标,pLDDT估计值与实际LDDT之间的相关性约为0.76。不确定性量化进一步估计,7%(pLDDT > 90 时)到 30%(70 < pLDDT < 90 时)的侧链的 χ1 角度偏差至少为40°。在作者的分析结果中,平均pLDDT为94,其中12%的残基的pLDDT在70到90之间。
因此,AlphaFold开发过程中估计的误差与作者的观察结果基本一致,即7%到20%的pLDDT为70或以上的侧链与实验数据不符。
结论
虽然AlphaFold预测通常具有惊人的准确性,但作者发现AlphaFold预测的许多部分与相应晶体结构的实验数据不一致。作者的研究结果表明:
(1)AlphaFold预测未必能比PDB中保存的模型更好地描述晶体结构(在预测模型和PDB中模型出现差别的地方,PDB中模型与实验数据的吻合度更高)。
(2)即使是置信度非常高的AlphaFold预测结果,其与PDB中保存的相应模型之间的差异也是PDB中不同空间群结晶的高分辨率结构对的两倍左右,这表明AlphaFold预测结果的误差超出了由于灵活性而产生的预期误差。
(3)由于AlphaFold预测没有考虑配体、离子、共价修饰或环境条件的存在,因此不能指望它能正确反映蛋白质结构的许多细节。
每个AlphaFold预测都会产生一个置信度指标(pLDDT)。DeepMind 团队对这一置信度指标进行了详细研究,结果表明它是无偏的(过低或过高的可能性相同),并且与它所估计的 LDDT 指标具有良好的相关性(皮尔逊相关性为 0.76)。因此,该置信度指标可以作为预测准确性的一个非常有用的残基特定指标。在本文研究的结构中,AlphaFold预测中置信度非常高的部分(pLDDT > 90,占分析残基的86%)一般都相当准确(与蛋白质数据库中结构模型的 Cα 坐标差异中位数为 0.6 Å)。不过,值得注意的是,在可信度非常高的残基中,约有10%与蛋白质数据库中结构模型的差异超过2 Å(表1)。
那么AlphaFold预测的价值何在?
作者认为,尽管存在局限性,AlphaFold预测已经在改变蛋白质结构假设的生成和检验方式。事实上,尽管AlphaFold预测的所有部分都不准确,但它们提供了可信的假设,可以提示作用机制,并允许设计具有特定预期结果的实验。将这些预测作为起始假设还能大大加快实验结构确定的过程。AlphaFold预测通常具有很好的立体化学特征,使其成为局部结构特征的极佳假设。
所有这些功能很可能只是人工智能方法在结构生物学中日益广泛应用的开端。人工智能方法必将从蛋白质扩展到核酸、配体、共价修饰、环境条件、所有这些实体之间的相互作用以及多种结构状态。
随着更多因素的加入以及序列和结构信息数据库的扩大,这些预测的准确性以及与之相关的不确定性很可能会不断提高。预测结果将成为越来越有用的结构假说,为生物系统的实验和理论分析奠定坚实的基础。
参考资料
Terwilliger, T.C., Liebschner, D., Croll, T.I. et al. AlphaFold predictions are valuable hypotheses and accelerate but do not replace experimental structure determination. Nat Methods (2023).
https://doi.org/10.1038/s41592-023-02087-4
——— End ———



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


