protein language model》的研究论文(图1)。张贵军教授课题组的研究团队在之前开展的基于超快形状识别(USR)蛋白质模型评估方法DeepUMQA(Bioinformatics, 2021)、基于序列和结构知识增强的蛋白质模型质量评估方法DeepUMQA2(Briefings in Bioinformatics,2022)、蛋白质复合物模型质量评估服务器DeepUMQA3(Bioinformatics, 2023)、基于等变图神经网络的蛋白质结构全局打分模型方法GraphGPSM(Briefings in Bioinformatics,2023)等工作基础上,进一步提出了基于深度图耦合网络语言模型的蛋白质模型质量评估方法GraphCPLMQA。该工作融合从蛋白质语言模型中学习到的潜在序列信息与模型结构知识,推断出蛋白质复合物残基级的模型质量,建立序列、结构与质量的关联,达到模型的准确性精度评估的目的。浙江工业大学信息工程学院张贵军教授为该论文通讯作者,张贵军教授课题组的刘栋博士生、张彪博士为论文共同第一作者。
图1.《Briefings in Bioinformatics》发表论文
一、研究思路
GraphCPLMQA的研究方案如图2所示。GraphCPLMQA包括使用多序列比对信息GraphCPLMQA-MSA和单序列信息GraphCPLMQA-Single两个版本,它们都是由图编码模块和基于Transformer的卷积解码模块组成。对于输入的模型结构,首先基于序列信息利用ESM蛋白质语言模型得到高维序列嵌入,并提取出模型结构的高、低维几何特征和理化能量特征。通过编码模块将序列信息与结构信息相融合获得几何约束表示,然后输入解码模块中预测距离偏差图和距离阈值图,并最终推断出蛋白质模型每个残基的质量。
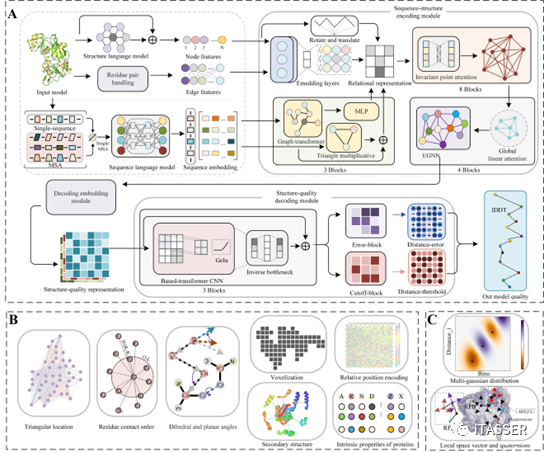
图2. GraphCPLMQA流程图
二、实验结果
在CASP15复合物的模型接口评估中,作者从CASP15复合物数据集中收集了9108个蛋白质模型,并与CASP15复合物界面接触残基精度评估赛道中排名第一的方法GuijunLab-RocketX(即DeepUMQA3,张贵军教授课题组开发)以及目前最主流的复合物模型局部接口评估方法ModFOLDdockR等进行了比较(图3)。测试集上的评估结果表明,GraphCPLMQA-Single与 ModFOLDdockR相比在Pearson指标上提高了23.6%,并相对于 GuijunLab-RocketX提升6%,并在其它指标上,依然取得相对最高的水平。进一步,作者分析了在不同多聚体类型(同源寡聚物和异源寡聚物)上的表现。有趣的是,对于同源寡聚物和异源寡聚物该方法的精度评估结果基本一致,这说明多聚体的类型并不会影响该方法的性能。 GraphCPLMQA-Single可以更为准确的评估复合物局部接口的质量,这为复合物界面残基质量提供一种可靠的置信度评估方法。
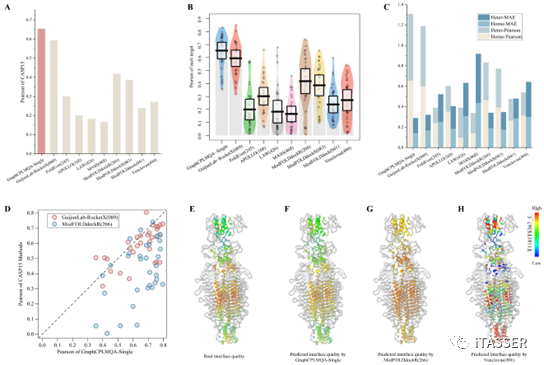
图3.GraphCPLMQA与最主流复合物模型接口评估方法的比较
为了进一步验证方法的性能,该工作以ZJUT-GraphCPLMQA(浙江工业大学– GraphCPLMQA服务器)参加了CAMEO模型质量盲测评估(CAMEO-QE)。在服务器参赛周期中,总共评估了超过3134 个的蛋白质结构模型。测试结果表明,除了在MSE上仅次于DeepUMQA2,GraphCPLMQA在各项指标上精度均超出其他参赛服务器,并在CAMEO盲测中连续6个月(2023年5月19日-2023年11月11日)获得总排名第一的成绩。此外,对于CASP13和CASP14的单体蛋白质测试集,GraphCPLMQA在全局和局部评估指标上均实现了最高精度超越了其他同类方法,其中QMEANDisCo和 DeepAccNet-MSA 分别是 CASP13 和 CASP14 中表现最好的局部模型质量评估方法之一。
表1. ZJUT-GraphCPLMQA在CAMEO盲测试集上的比较结果
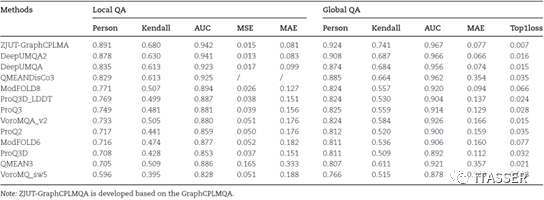
图4显示了GraphCPLMQA在测试集上与AlphaFold2比较评估的案例。作者分析了GraphCPLMQA对于AlphaFold2中高质量的蛋白质模型的评估结果。分析表明,AlphaFold2局部结构预测的准确性与评估结果密切相关。在某种程度上,AlphaFold2的pLDDT可能无法精确反映局部结构的质量。当AlphaFold2预测的局部结构区域与天然结构存在较大差距时,该区域所对应pLDDT局部质量评估并不精确,甚至得到相反的评估趋势。而对于高质量的AlphaFold2模型的pLDDT基本都高于真实模型质量。而GraphCPLMQA预测局部质量的分布非常接近真实的分布,这有助于弥补AlphaFold2的局部pLDDT存在的缺陷。在未来的研究中,GraphCPLMQA也可能为AlphaFoldDB中未解析出天然结构的模型提供有价值的评估参考。
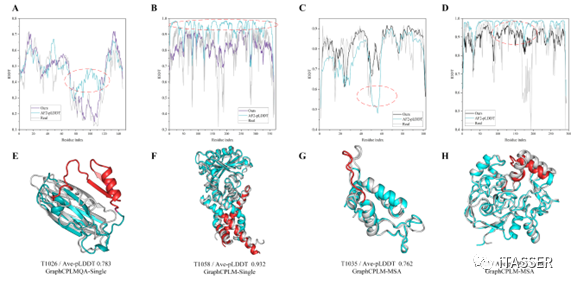
图4. GraphCPLMQA与AlphaFold2比较的评估案例
三、结论
GraphCPLMQA算法基于图耦合网络,在编码模块中融合从蛋白质语言模型中学习到的潜在序列信息与结构特征知识,并通过解码模块推断出残基级的模型质量。这表明通过序列、结构和质量相互联系的方式,可以深入挖掘序列和几何空间结构的映射关系,为模型结构和质量的推断提供一种可靠的指导,进一步提升模型准确性评估的精度。该工作尽管使用单体模型结构的数据来训练网络模型,但依然可以较好的评估复合物局部模型结构。这在一定程度上表明,单体内与单体间的局部空间结构有相似之处。当然,复合物结构模式可能更为复杂多样,需要进一步深入的研究和探索。目前,在复合物模型质量评估中依然存在许多挑战,期望借助最新深度学习技术,辅以复合物模型特征工程探索,将模型准确性评估引领到一个新的高度,推动复合物结构预测技术的发展。
原文网址:
https://academic.oup.com/bib/article/25/1/bbad420/7450272
服务器网址:
http://zhanglab-bioinf.com/GraphCPLMQA




 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


