今天为大家介绍的是来自Jiawei Luo和Kelin Xia团队的一篇论文。分子表征学习在分子性质预测中扮演重要角色。现有的分子性质预测模型基于共价键的分子图,用以在原子层面上表示分子拓扑结构,并且完全忽略了分子内部的非共价相互作用。在这项研究中,作者提出了一种分子几何深度学习模型,旨在预测分子的性质,该模型致力于综合考虑分子的共价和非共价相互作用信息。

基于人工智能(AI)的模型已在预测各种分子性质方面展现出巨大的力量和潜力。特别是,在AI驱动的药物设计方面,已在药物虚拟筛选的各个步骤中取得了巨大成功,并有潜力彻底改变药品行业。然而,即使有这样的进步,设计高效的分子表征和特征化仍然是一个巨大的挑战。通常,所有基于AI的分子模型可以分为两种类型,即基于分子描述符的机器学习模型和端到端的深度学习模型。
第一种类型使用分子描述符或指纹作为机器学习模型的输入特征。描述符的生成被称为特征化或特征工程。除了物理、化学和生物属性,如原子偏电荷、疏水性、电子性质、空间性质等,大多数分子特征来自分子结构属性。事实上目前已经开发了5000多种分子结构描述符,可以概括为一维(1D)、二维(2D)、三维(3D)和四维(4D)特征。1D分子描述符包括原子计数、键计数、分子重量等和其他总结的一般属性。2D分子描述符包括图属性、组合属性等。3D分子描述符包括分子表面属性等。生成3D分子描述符通常需要较高的计算成本。4D化学描述符则表征了动态过程中的构型变化。第二种类型是端到端几何深度学习(GDL)模型。在这些GDL中,分子被表示为分子图、密度函数或分子表面,可以使用各种深度学习模型,如三维卷积神经网络(3D CNNs)、图神经网络(GNNs)、递归神经网络(RNNs)等,来自动学习分子性质。在这些不同的分子表征中,分子图是最受欢迎的一种。特别是,基于共价键的分子图成为了在原子层面表示分子拓扑结构的通用标准。在这里,作者首次展示了仅包含非共价相互作用的分子表征可以在分子性质预测中达到与共价键基模型相同甚至更好的效果。更具体地说,作者系统地在几个最常用的基准数据集上比较了使用两种类型的分子表征的GDL模型的性能,即共价相互作用图和非共价相互作用图。
基于共价键的图是黄金标准吗?
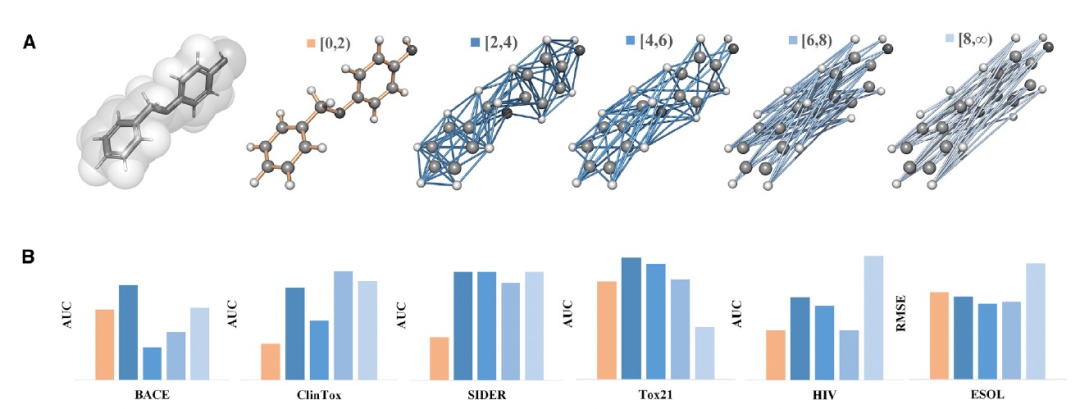
图 1
作者展示了在分子性质预测中,仅由非共价相互作用构建的分子图的GDL可以达到与共价键基模型相似甚至更好的效果。在此考虑了五种不同的分子图表征及其在六个最常用数据集(包括BACE、ClinTox、SIDER、Tox21、HIV和ESOL)上的GDL性能,如图1所示。在这五种不同的分子图中,一种是基于共价键模型,其他四种都是仅使用非共价相互作用构建的。换句话说,这四个分子图中的所有边都仅代表非共价信息,没有一个是由共价键生成的。(这四个非共价分子图是通过仅在一定预定义的欧几里得距离内的原子之间定义边来构建的。作者为每个图指定了一个特定域I,即这个图中的边仅在相应的两个原子之间的距离位于域I内时存在,具体设置如图1A所示。)从图1中可以看出,基于共价键的分子图的GDL并不具有最佳性能。相比之下,使用非共价分子图的GDL不仅可以取得可比的结果,甚至还可以超越基于共价键的模型。
模型部分(Mol-GDL)
对于一个有N个原子,原子坐标分别为r1, r2,…, rN的分子,其分子图表征可以表示为G(I) = (V, E(I))。这里,I是用来定义图表征的特定相互作用区域。在分子图表征中,每个原子被模型化为一个单独的节点。Mol-GDL不仅仅使用一个分子图,而是通过选择不同的区域I来系统地生成一系列的图。从几何学角度来看,对于特定的图,只有当两个原子之间的欧几里得距离位于一定区域内时,才会在它们之间形成一条边。通过这种方式可以灵活地构建不同的分子图。在Mol-GDL中另一个重要的设置是与距离相关的节点特征。与传统的节点特征不同,作者设置的节点特征仅包含原子类型和距离信息。
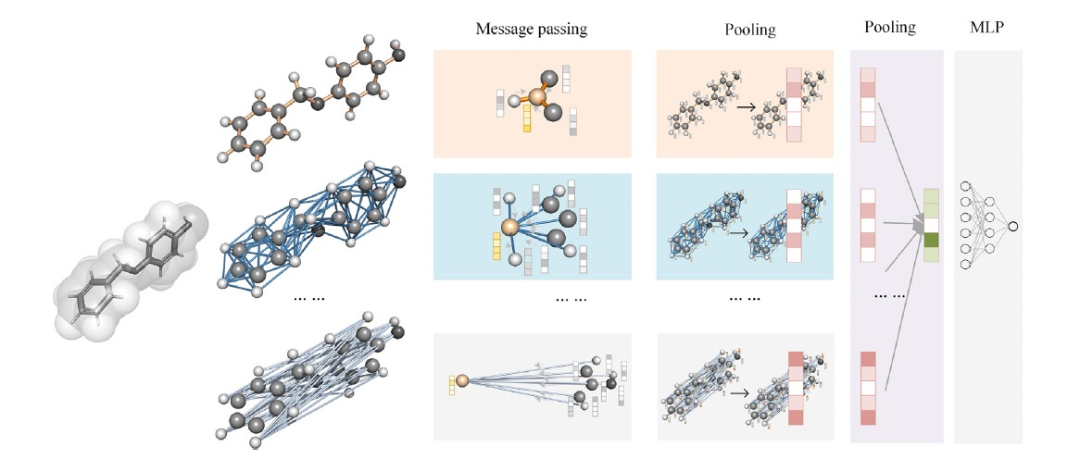
图 2
Mol-GDL提供了一个能够全面学习分子内多尺度信息的GDL架构。图2展示了Mol-GDL的流程图。与之前所有的GDL不同,该模型系统地构建了一系列关注不同尺度相互作用的分子图。在每个单独的分子图上都采用了一个共同的消息传递(MP)模块。之后,进行了两次池化操作。第一次池化在原子层面进行。对于每个分子图,节点特征被聚合到一个单一的分子特征向量中。第二次池化在分子图层面进行。来自第一次池化的每个分子特征向量通过单层感知机,然后被连接成一个单一的特征向量。最后,包含所有分子图信息的单一特征向量通过多层感知机(MLP)生成最终预测。
实验部分
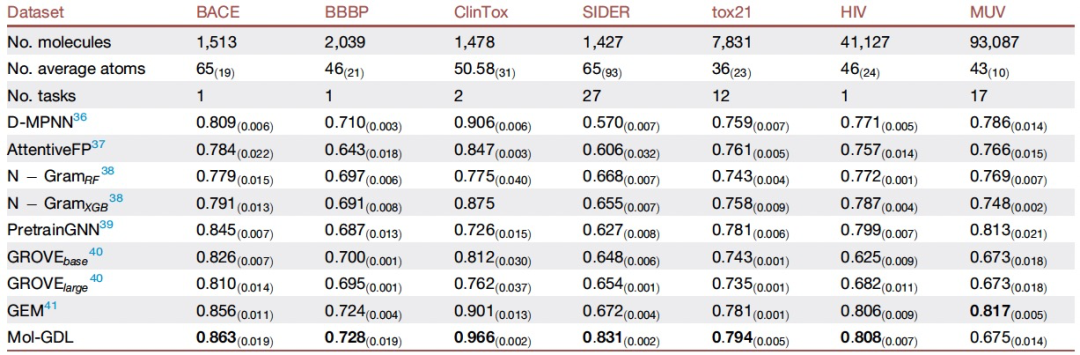
表 1
在分类任务中,作者考虑了七个常用数据集。这些数据集大致可以分为两类:一类是生物物理性质(BACE、HIV和MUV),另一类是生理性质(BBBP、Tox21、SIDER和ClinTox)。Mol-GDL在分类基准测试的比较显示在表1中。可以看到,Mol-GDL在除了MUV的一个任务外的所有任务中均取得了最佳结果,并且持续超越SOTAs。Mol-GDL在BACE上的AUC为0.863,优于之前最佳的结果(AUC = 0.856)。Mol-GDL模型在HIV和MUV两个数据集上的AUC结果分别为0.808和0.675。Mol-GDL在BBBP和SIDER上的结果显著优于SOTAs。
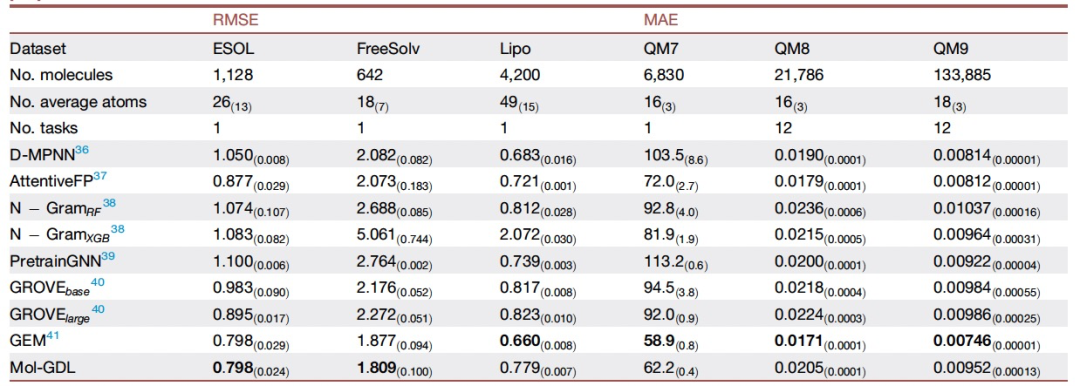
表 2
随后作者在回归任务上对模型进行了检测,如表2所示。用于分子性质回归任务的常用数据集主要分为两类。一类是预测分子的物理和化学性质,包括ESOL、FreeSolv和Lipo。另一类是量子化学领域,包括QM7、QM8和QM9。总的来说Mol-GDL可以达到与SOTA相似甚至更好的结果。特别是,Mol-GDL在ESOL和FreeSolv任务上拥有最小的RMSE值。在QM7数据集上,Mol-GDL模型的MAE值为62.2,仅次于GEM。
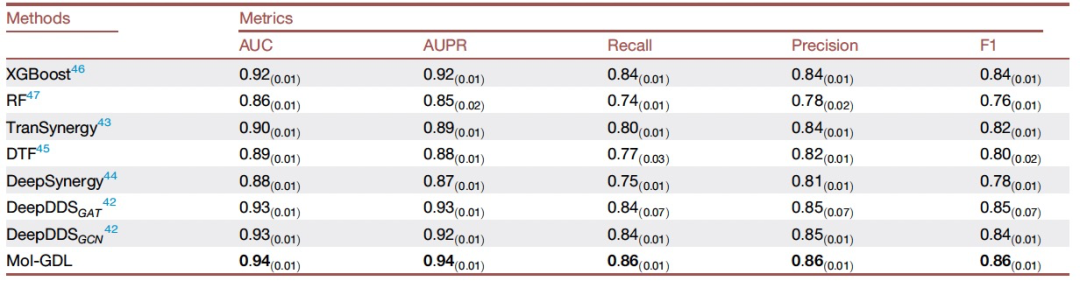
表 3
作者还在药物协同作用任务上对模型进行了分析。作者使用了来自DeepDDS的数据集。由于这个测试涉及到不同细胞系中两种药物间的药物组合预测,因此使用了略有不同的架构。作为基准,使用了五种基于深度学习的药物协同作用预测方法,包括TranSynergy、DeepSynergy等,以及两种机器学习方法。表3展示了Mol-GDL模型在预测协同药物组合方面的整体性能。可以看出,Mol-GDL模型在五个验证指标上都优于其他所有方法。
编译 | 曾全晨
审稿 | 王建民
参考资料
Shen, C., Luo, J., & Xia, K. (2023). Molecular geometric deep learning. Cell Reports Methods.




 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง





