
作者提出了一种基于多视图的图神经网络和变分自编码器生成药物分子骨架(Scaffold Generation based on multi-view GNN and Variational AutoEncoder, ScaffoldGVAE)。该模型集成了以节点为中心和以边缘为中心的信息传递、侧链嵌入和骨架的高斯混合分布等重要组成部分。结果表明,ScaffoldGVAE可以探索未知的化学空间,并产生不同于已知化合物的新分子。作者通过对模型生成的分子进行验证,证明了ScaffoldGVAE生成新化合物的有效性。这种新方法也可以应用于各种疾病的其他蛋白质靶点,从而有助于未来新药的开发。
背景
近年来,由于深度学习技术的应用,药物设计发生了革命性的变化,而分子生成是这一转变的关键方面。然而,目前大多数深度学习方法在进行分子生成时,没有明确考虑和应用骨架跳跃策略。
方法
ScaffoldGVAE的模型架构是基于变分自编码器(VAE)的概念。然而,与传统的基于VAE的分子生成方法不同,ScaffoldGVAE重点是在分子设计中生成骨架以促进骨架的跳跃。为了实现这一目标,作者提出了一种新的变分自编码器,专门设计用于骨架生成。该编码器(图1A)利用多视图的图神经网络分别对分子的边(键)和节点(原子)进行编码,即分别以节点和边为中心进行消息传递。在读出阶段,将节点和边的嵌入连接在一起,得到整个分子嵌入。根据分子骨架的不同,分子嵌入可进一步分为侧链嵌入和骨架嵌入两部分。
解码器(图1B)采用递归神经网络(RNN)模型将骨架嵌入与侧链嵌入连接为初始隐式向量,从而实现骨架SMILES的重建。该骨架生成过程考虑了原分子的骨架信息和侧链信息。图1C和图1D分别描述了骨架跳跃策略:与参考分子侧链结合的新型骨架取样;以及描述模型的训练、采样和骨架跳跃的流程图。
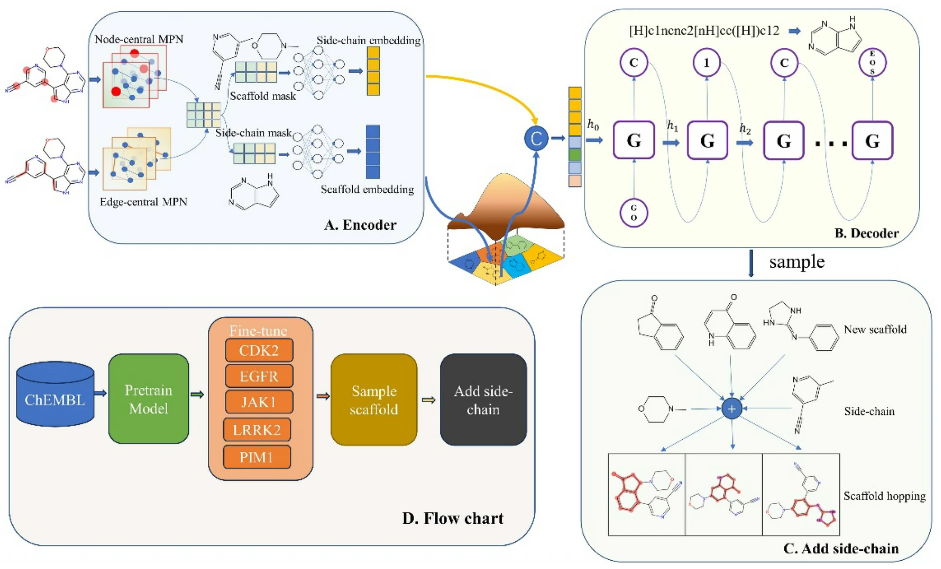 图1 ScaffoldGVAE结构图
图1 ScaffoldGVAE结构图
模型中的编码器采用图消息传递神经网络对分子图进行有效编码,如图1A所示。图中的每个节点都与一个节点特征向量相关联,该特征向量捕获原子的基本属性,如原子类型、原子价和其他相关特征。类似地,图中的每条边都由一个封装键类型的特征向量表示。
为了引入骨架跳跃,区分分子内的侧链和骨架部分是至关重要的。这需要为属于骨架的节点赋值1,为侧链中的节点赋值0。这种区分使得人们能够有针对性地关注骨架的特性,从而可以利用骨架跳跃技术产生新的分子的定长图表示,并执行读出操作。读出函数利用图自注意力机制,得到适合后续骨架跳跃任务的定长表示。
在训练阶段,提取归一化的骨架SMILES并将其编码为独热向量,作为重建骨架SMILES的目标。为了方便将单个标记转换为有意义的向量表示,使用了一个包含128个单元的嵌入层。该层将每个标记转换为128维向量。模型的GRU组件包括三层,每层包含512个神经元。这些层有效地捕获SMILE序列中的依赖关系和模式。最后,GRU层的输出被馈送到一个密集的连接层,该连接层具有与单词总数相同数量的神经元,包括指示SMILES字符串开始和结束的附加标记。
该模型基于从训练集中学习到的分子语法和语义,以及分子和骨架的空间分布进行骨架采样。给定参考分子及其相应的骨架,该模型产生新的骨架,可以取代原来的骨架。利用基于图的神经网络对分子骨架和侧链进行编码,实现信息传递。当侧链保持不变时,骨架嵌入从隐藏空间重新采样。这两个嵌入作为初始隐藏向量输入到RNN中。然后使用RNN模型的自回归特性对骨架进行采样。随后,参考分子的侧链被组装到新采样的骨架上。这一过程产生了具有新型骨架的分子。侧链的剪接如图1C所示。导致骨架跳跃的骨架采样和拼接的整个过程如图1D所示。通过利用分子语法和空间信息,该模型生成了多种骨架替代品,从而为药物设计和发现探索新的化学空间。
解码器输出骨架后,需要将侧链添加回骨架中,以获得完整的生成分子,如图1C所示。在这里,使用RDKit工具将骨架与侧链结合起来,遵循以下简单的原则:(1)将采样的骨架与原始骨架进行比较,列举所有可能的侧链安装在采样的骨架上;(2)计算添加侧链后的分子与原分子拓扑指纹的相似度;(3)采用添加侧链的方式,使生成的分子尽可能与原分子相似;(4)检查生成的分子的有效性,如果存在价键错误或侧链无法连接等情况,则认为该分子无效。遵循这些原则,该模型最终可以产生具有跳跃骨架和不变侧链的新分子。
结果
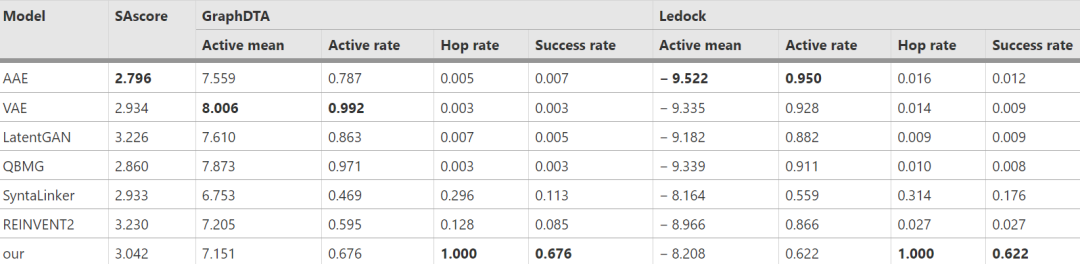
作者将ScaffoldGVAE与一些具有代表性的方法进行了比较。比较方法包括:(1)比较生成分子的可合成性分数(SAScore),SAScore范围在1-10之间,越低表示越容易合成。(2)将生成分子分别用GraphDTA和Ledock这两种分子对接预测工具,预测在不同的给定靶标上,分子与靶标的结合活性均值,活性率,骨架跳跃率(可反映生成分子的新颖性),结合成功率。如表1所示。以CDK2靶标蛋白为例(其他靶标上的结果详见原文),ScaffoldGVAE在所生成分子的合成难度与其他方法相近的情况下,具有较高的骨架跳跃率(新颖性)和对接成功率。
表1 与其他方法对比
作者设计了模型消融实验。ScaffoldGVAE是基于多视图的图神经网络,即结合了节点中心消息传递网络和边缘中心消息传递网络,使信息从边缘和节点两个角度传播。分别采用去除节点中心网络(模型1)和去除边缘中心网络(模型2)作为消融实验。此外,模型中使用变分自编码器来编码分子和解码骨架,这与一般的分子到分子生成方法不同。
ScaffoldGVAE采用这种策略,而没有选择骨架到骨架或分子到分子的编码-解码策略,有两个原因。首先,骨架对骨架的编解码策略会丢失侧链的信息,难以保证生成适合侧链的骨架。其次,分子间编码-解码策略不能同时保证骨架跳跃和侧链保留。作为比较,在消融实验中也测试了这两种策略,分别对应不侧链嵌入(模型3)和不侧链添加(模型4)。最后,模型中涉及的高斯混合分布对骨架跳跃非常重要。模型5为不含高斯混合分布的消融模型,即一般的基于图的VAE。模型6包含了所有这些组件。
本研究所进行的消融实验总结如表2所示。结果表明,缺失节点中心(模型1)、边缘中心(模型2)或侧链嵌入(模型3)的模型的性能略低于完整模型(模型6)。此外,没有侧链添加策略的直接分子到分子生成模型(模型4)的性能低于包含侧链添加步骤的分子到骨架模型(模型6)。模型5的消融效果比其他所有消融模型都差,成功率只有10%左右,与VAE、AAE等基线模型相当。这是因为模型5本质上是一个图VAE模型。这些发现为模型中每个成分的重要性提供了有价值的见解,并强调了在分子中加入侧链添加步骤对生成过程的重要性。
表2 消融实验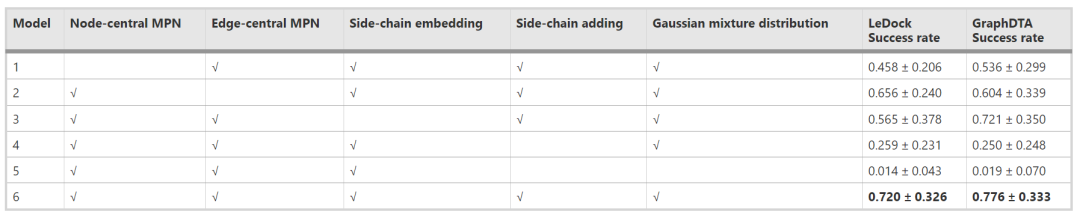
作者还进行了案例分析。
从图2中可以看出,大多数生成的化合物具有良好的结合能,结合能在−40 ~−60 kcal/mol之间。这表明这些化合物可能以高亲和力与LRRK2蛋白结合。作者还分析了Cpd2和Cpd4与参考化合物的结合模式,分别具有较低和较高的结合能。如图2B、C所示,结果表明这些化合物与LRRK2蛋白的关键残基Glu85、Leu86、Ala87等相互作用。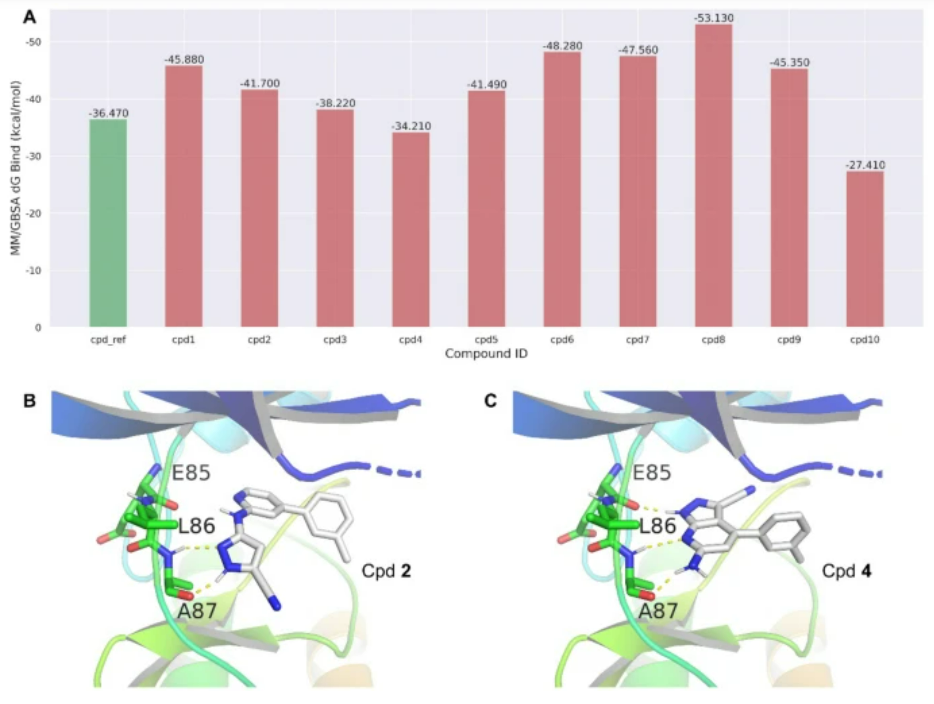 图2 案例分析
图2 案例分析
结果表明,大多数生成的化合物除了具有良好的结合能之外,可能以高亲和力与LRRK2蛋白结合。这些化合物可以进一步优化和合成用于体外测试,发现治疗帕金森病和其他相关疾病的新药。
总结
在这项研究中,作者提出了ScaffoldGVAE模型,这是一个专门为药物分子骨架跳跃设计的深度学习模型。该模型基于变分自编码器的架构,其中编码器组件利用了最先进的多视图图神经网络。该神经网络同时考虑了以边为中心的消息传递和以节点为中心的消息传递,从而增强了编码器的信息传播能力。
解码器采用RNN模型将潜在向量解码为骨架SMILES表示。此外,作者还引入了一种骨架跳跃的算法。结果表明,模型能够探索未知的化学空间,并产生不同于已知化合物的新分子,在骨架跳跃性能方面优于基线模型。这种新方法将有助于未来新药的开发。
参考文献
[1] Hu et al. ScaffoldGVAE: scaffold generation and hopping of drug molecules via a variational autoencoder based on multi-view graph neural networks. J Cheminform. 2023
——— End ———



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


