今天为大家介绍的是来自Nir Yosef团队的一篇论文。作者提出了MultiVI,一种用于分析单细胞的转录组、染色质可访问性和其它分子特性的概率模型,这为研究细胞多样性提供了一个强大的方法。MultiVI能创建一个联合表示,不仅可以分析多组学数据中包含的所有模态,即便是对于那些缺失一种或多种模态的细胞,也能进行分析。这样的方法能够有效地利用多组学数据,提升单一模态数据集的分析能力。

单细胞分辨率下的转录组和染色质景观分析技术的出现,在分类细胞类型和状态方面起着至关重要的作用。然而,大多数使用单细胞RNA测序(scRNA-seq)和单细胞转座酶可访问染色质测序(scATAC-seq)的方法限制在给定细胞只能被一种技术分析。近期,出现了同时分析单个细胞的基因表达、染色质可访问性及表面蛋白丰度的多模态单细胞方法。这种同时测量使得细胞状态的更精细分类成为可能,从而更好地理解其多样性背后的机制。为了提供全面的解决方案,这种整合分析应该在两个层面上进行。首先,它应该生成反映每个细胞状态的低维信息,无论该细胞可用哪种类型的信息。如同在单细胞基因组学的其他应用中常做的那样,这样的表示可以促进亚群的识别,并实现更具信息性的数据可视化。第二层分析应该生成每种高维数据类型的规范化、批量校正视图,无论是观察到的还是推断出的。这样的分析可以使得更广泛地识别表征感兴趣的细胞亚群的分子特征成为可能。在这里,作者介绍了MultiVI,这是一个用于多模态数据集概率分析的深度生成模型,也支持它们与单模态数据集的整合。以基因表达和染色质可访问性为主要案例,作者展示了MultiVI如何为两层分析提供解决方案。
模型部分
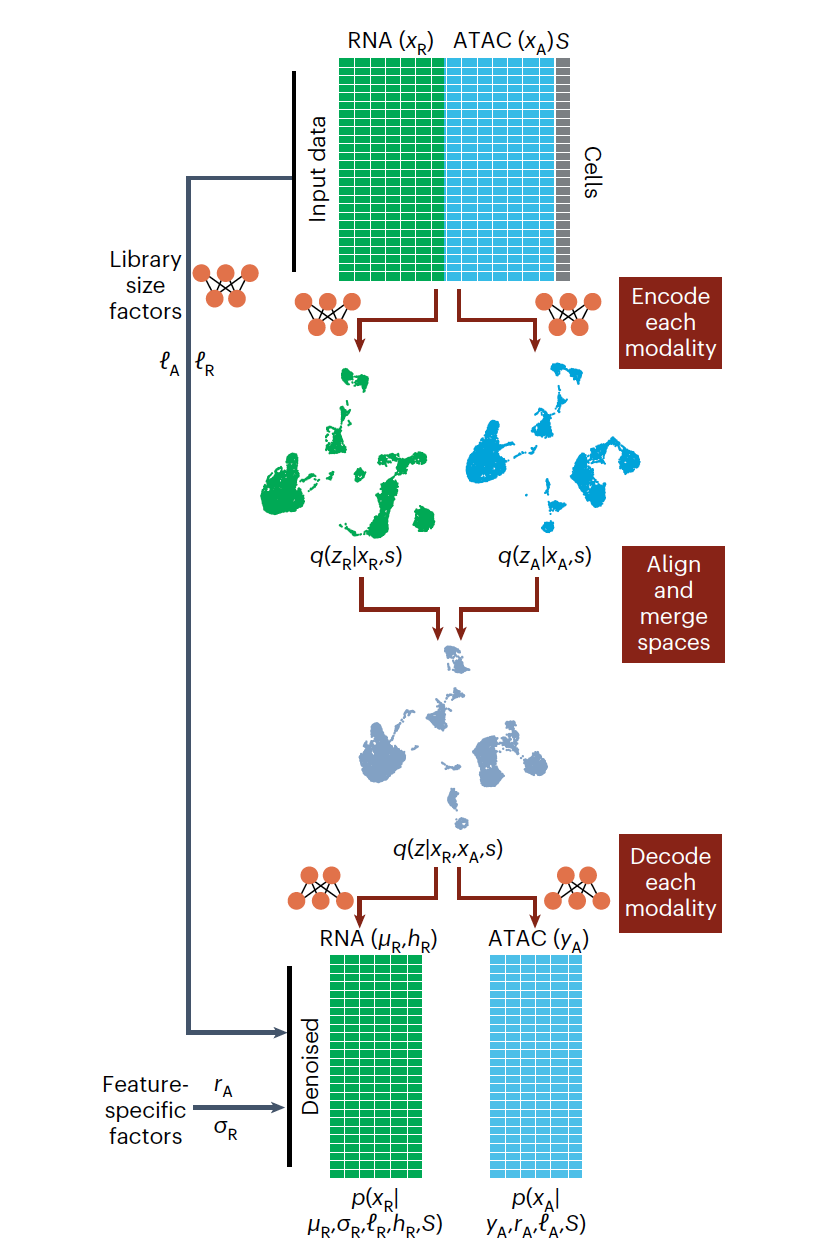
图 1
MultiVI(图1)利用了作者之前提出的针对基因表达(scVI)、染色质可访问性(PeakVI)和蛋白质丰度(totalVI)的变分自编码(VAE)模型。为了清晰起见,这里的讨论重点是联合模拟scRNA-seq和scATAC-seq数据。给定单个细胞的多模态数据X和样本(或批次)S,作者将观测数据分为基因表达(XR)和染色质可访问性(XA)。两个深度神经网络,称为编码器,学习模态特定的、批次校正的多变量正态分布,这些分布代表基于观测数据的细胞潜在状态,即表达观测值的q(zR∣XR, S)和可访问性观测值的q(zA∣XA, S)。为了获得反映两种模态的潜在空间,作者惩罚模型使两个潜在表示之间的距离最小化,然后估计整合细胞状态q(z∣XR, XA, S)作为两个表示的平均值。
对于只有一种模态可用的细胞(即非配对),它们的状态直接从有数据的表示中提取(即zR或zA)。这个编码模型的部分可以自然地扩展以处理其他分子属性(如蛋白质丰度)。在模型的第二部分,观测值从潜在表示中生成,使用模态特定的解码器神经网络。与作者之前针对基因表达(scVI)和可访问性(PeakVI)的模型类似,RNA表达数据来自于负二项分布,可访问性数据来自伯努利分布。对于配对(多模态)细胞,似然从两种模态计算,而对于非配对细胞,则只从相应的模态计算。最后,在训练过程中模型包括了一个对抗组件,如果来自不同模态的信息在潜在空间中过度分离,则对模型进行惩罚。
MultiVI 整合成对和不成对的样本信息
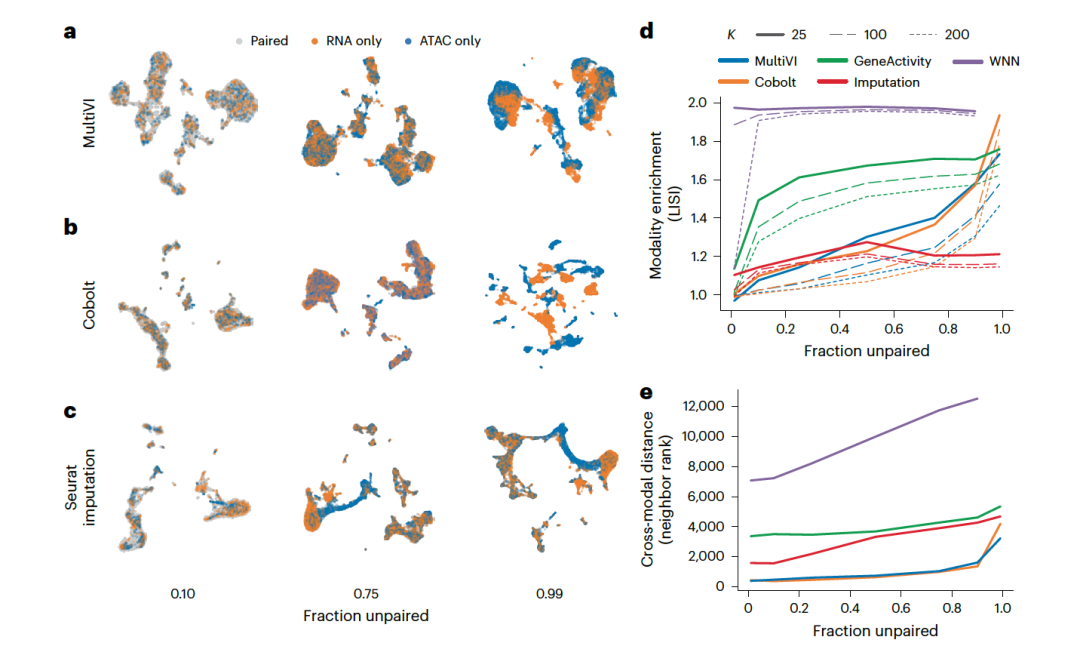
图 2
作者首先在一个完全配对的名为PBMC数据集上进行训练,这个数据集包含了每个细胞的基因表达和染色质可访问性信息。实验发现模型预测与实际观测到的独特分子标识符数量高度相关(皮尔森相关性在两种模态上为 0.97和0.91),这表明模型能够准确地捕捉到数据的重要特征。为了进一步测试模型的性能,作者进行了一个实验,他们人为地将数据集中的一些细胞解除配对,创建了一个异质数据集。这个数据集包含三种类型的细胞:一部分细胞同时具有基因表达和染色质可访问性数据,一部分只有基因表达数据,另一部分只有染色质可访问性数据。这样做的目的是模拟现实世界中常见的情况,即不是所有细胞的所有数据都是完全配对的。通过这种方式,研究者们可以评估MultiVI在整合不完全配对的多模态数据方面的性能。作者使用这些解除配对的数据集将MultiVI与Cobolt模型进行了比较,并同时比较Seurat V4代码库的三种不同方法:(1)基因活性,将仅可访问性细胞的ATAC-seq数据转换为基因活性分数(使用signac程序),然后使用基因级别的数据整合所有细胞(即当RNA-seq不可用时使用基因分数,当RNA-seq可用时使用基因表达);(2)推断,按照(1)中的步骤操作,然后使用Seurat为仅可访问性细胞推断RNA表达值;(3)加权最近邻(WNN),使用WNN图,利用两种模态的信息创建一个联合表示空间,然后将单模态数据投影到这个空间。
实验结果显示基于加权最近邻(WNN)的方法在99%解除配对的数据集上未能产生结果,因为配对细胞数量过少。作者通过计算局部逆辛普森指数(LISI)得分来量化混合性能,发现基于生成模型的算法(如Cobolt和MultiVI)在大多数解除配对细胞的比率上优于基因评分和WNN方法(图2)。然而,基于Seurat的推断方法在所有级别的解除配对细胞上保持较高的混合性能。接下来作者考察了推断潜在空间的准确性,特别关注未配对细胞两种模态特定表示在潜在空间中的距离(图2e),发现MultiVI和Cobolt在保持多模态混合准确性方面表现优于其他方法,但所有方法的性能都随着解除配对细胞的比例增加而下降。
整合不同的数据分析
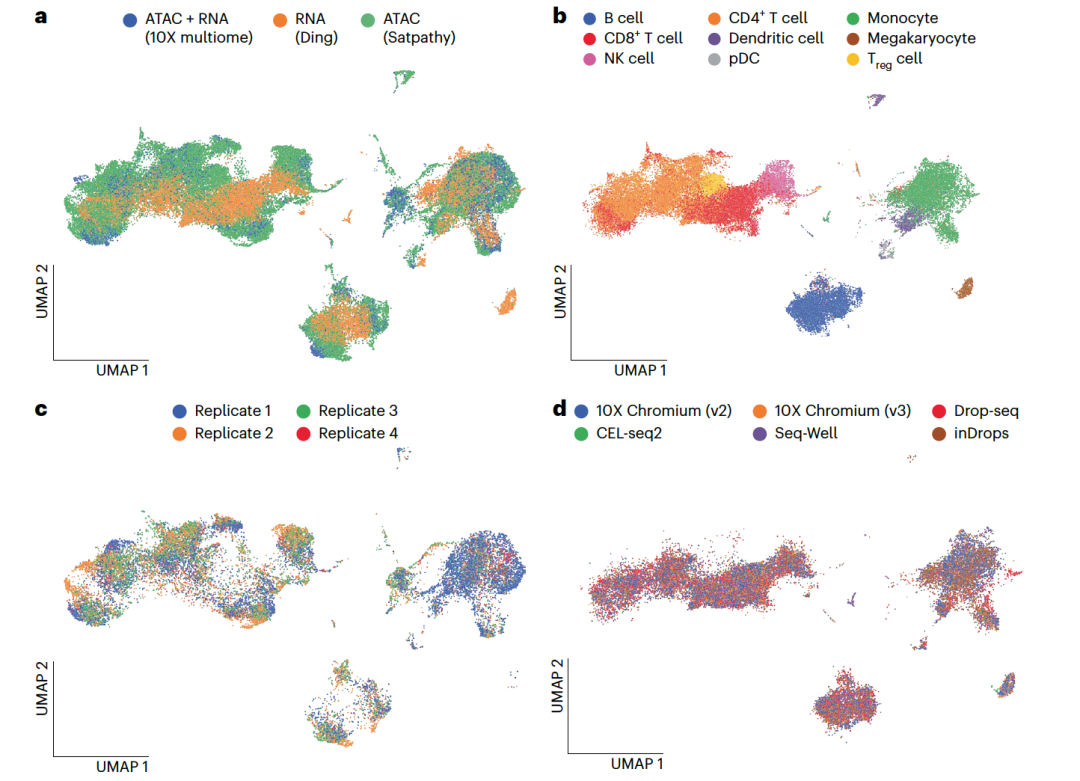
图 3
作者的基准分析(见图2)依赖于人为不配对的数据,这些数据基本上是在单一批次和单一技术中生成的。这并不反映现实世界中的情况,在现实世界中,我们希望整合在不同批次甚至不同研究中生成的数据集。因此,作者试图在一组真实世界数据上演示MultiVI。作者收集了三个不同的外周血单核细胞(PBMCs)数据集。这些数据集被处理以创建一组共享特征并结合成一套共享的细胞类型标签。为了衡量这些数据中批次效应的程度,作者在没有考虑每个样本的来源状况或其特定技术运行了MultiVI。在这种设置下,细胞根据在可访问数据中的样本和在可表达数据中的技术进行分层,表明批次效应影响了潜在的表示。然后作者配置MultiVI来校正每个数据集内的批次效应和技术特异性效应,并重新进行了分析。结果的联合潜在空间很好地混合了这三个数据集(图3a),同时准确匹配了两个数据集中的已标记群体(图3b)。MultiVI在校正数据中的批次效应和数据中的技术特异性效应的同时实现了这一点(图3c,d)。
MultiVI的差补能力
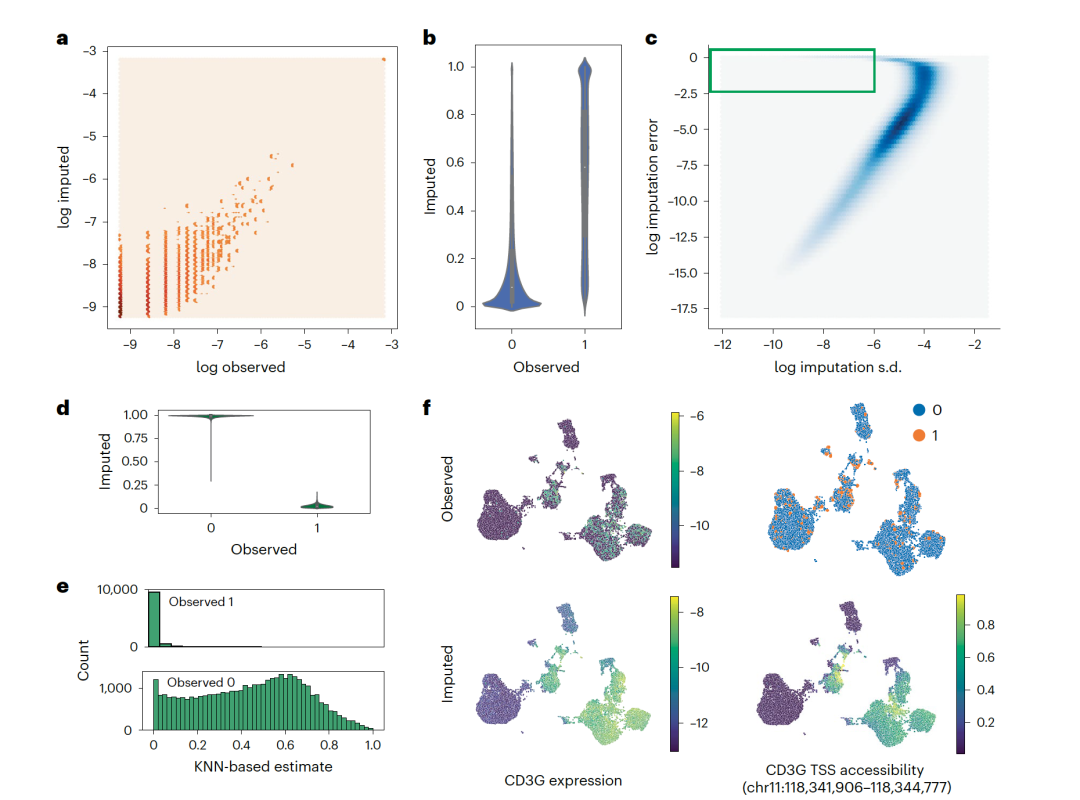
图 4
MultiVI是一种生成模型,它不仅可以分析全高维空间的数据,还能进行缺失观测值和模态的插补、不确定性估计和差异分析。为了评估MultiVI的数据插补能力,作者使用了图2所用的PBMC数据集,其中75%的细胞是人为不配对的。通过利用MultiVI推断缺失模态的细胞值,作者发现对于两种模态,插补的值与观测值有很高的一致性(图4a-c)。考虑所有基因表达条目,MultiVI在插补值和原始观测值(按库大小缩放)之间实现了0.57的斯皮尔曼相关性。由于原始染色质可访问性数据是二进制的,作者计算了精确度-召回曲线下面积来评估插补准确度,MultiVI达到了0.41。作者通过从MultiVI的生成模型中采样,来测量每个插补值的不确定性,并发现估计的不确定性与每个数据点的误差之间有很强的关系。这意味着,当预测值与真实值的差距越大时,模型的预测就越不确定(见图4c)。
编译 | 曾全晨
审稿 | 王建民
参考资料
Ashuach, T., Gabitto, M.I., Koodli, R.V. et al. MultiVI: deep generative model for the integration of multimodal data. Nat Methods 20, 1222–1231 (2023).
https://doi.org/10.1038/s41592-023-01909-9



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


