今天为大家介绍的是来自Ron O. Dror团队的一篇论文。药物设计中一个普遍的挑战是确定如何扩展配体以改善配体的各种属性。添加单个化学基团对于引导优化任务非常重要,而添加多个基团对于基于基团的药物设计至关重要。作者开发了一个综合框架,利用机器学习和三维蛋白质-配体结构来应对这一挑战。

药物发现过程变得越来越漫长和昂贵。通过从可能分子的庞大空间中选择最佳候选者,可以节省测试的时间和金钱,并最终找到更多可行的治疗药物。设计过程的一个关键部分是向起始分子添加化学基团(在下文称为“碎片”),这些起始分子已知能与目标结合,以此调整其属性,如亲和力、选择性和溶解度。例如,引导优化通常涉及一次添加一个或两个碎片到起始分子,然后进行迭代的测试和进一步的修改。在其他情况下,添加多个碎片很有用;在基于碎片的药物发现中,通常会向从结构筛选方法获得的起始分子添加多个碎片。
作者开发了一个综合框架(基于碎片的分子扩展模型(FRAME)),使用机器学习和三维蛋白质-配体结构来应对药物设计中的这一常见挑战。FRAME将扩展过程表示为3D空间中的一系列步骤。给定一个与蛋白质口袋结合的起始分子的输入结构,FRAME确定在哪里附加碎片,选择要添加的碎片,并确定碎片的几何形状。
方法部分
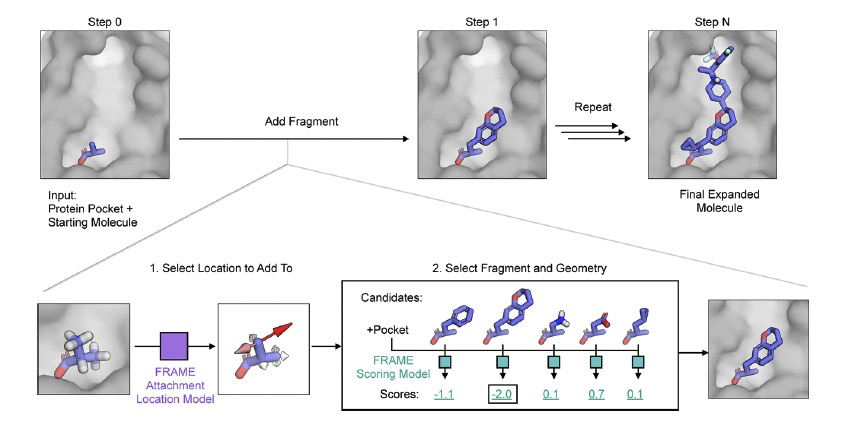
图 1
FRAME利用神经网络,基于当前的分子结构扩展配体分子。最初,结构包括起始分子(任何大小)以及包括每个原子的三维坐标和元素类型的蛋白质口袋(图1)。FRAME依次通过单键添加碎片到配体。每个动作分为两个步骤:首先,选择一个地方附加一个碎片,其次,选择要添加的碎片和附件几何形状。作者训练两个独立的SE(3)-等变神经网络来预测每个步骤(图1)。碎片从用户指定的库中选择;为了基准测试,作者使用了常见碎片的库。可以轻松地将额外的碎片包含在库中,因为FRAME能够评估在训练数据中未见过的碎片。
实验部分
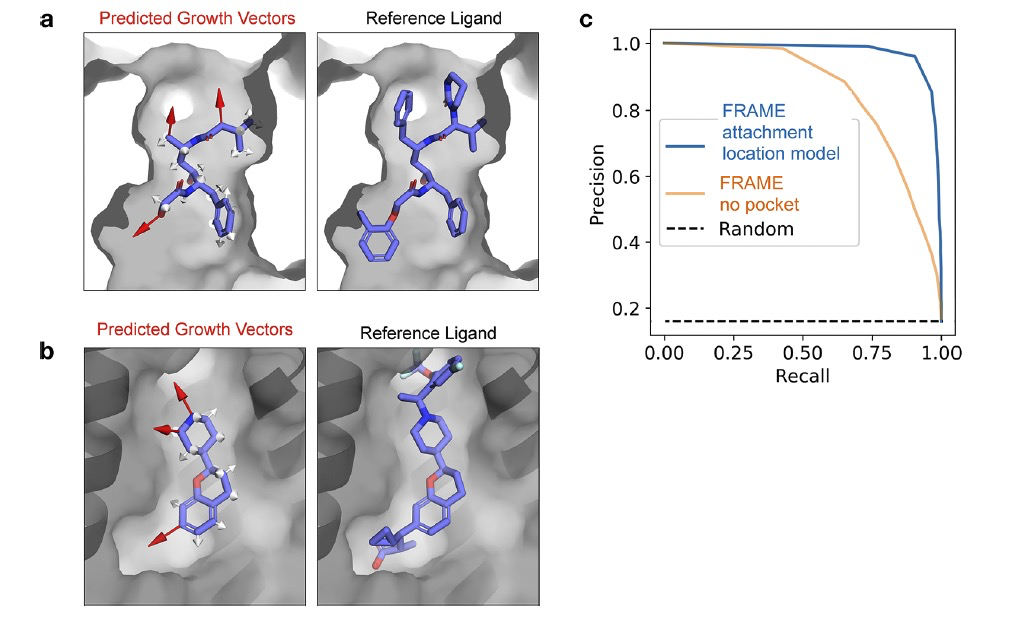
图 2
作者首先评估了模型在选择附着点和排列碎片这些更简单任务上的性能。首先,作者发现FRAME的附着位置模型选择了可行的附着点位置。从视觉检查来看,该模型经常识别出指向蛋白质口袋内未填充区域的无阻碍的附着位置(图2a,b)。给定测试集参考配体轨迹的中间状态,FRAME经常选择参考配体中实际使用的附着点。从定量上看,模型选择的点95%是参考配体中的附着点,92%的参考附着点被模型选中。FRAME模型远远超过了随机基准(图2c),总体而言,该模型在这项任务上泛化得很好。
接下来评估了FRAME在给定附着点选择单个碎片的能力,这适用于用户希望获得候选碎片排名列表的配体优化任务。作者发现模型经常选择形成关键相互作用的碎片,尽管模型对这些相互作用或甚至化学性质如供体和受体没有先验知识(图3a,b)。在一个案例研究中,FRAME选择了形成π−π相互作用和多个氢键的环状碎片——参考配体中发现的重要相互作用(图3a)。值得注意的是,FRAME正确区分了这些杂环与不能形成相同相互作用的其他环。FRAME还能够丰富多样的碎片。在另一个例子中,前三个碎片在大小和化学性质上有所不同,但都与口袋形成了相同的关键氢键(图3b)。
FRAME选择了与蛋白质口袋形成关键相互作用的碎片,这对于配体的亲和力和特异性很重要。配体中的所有碎片并不都同样重要;如盐桥这样的相互作用虽然罕见,但通常对功能效果至关重要。经过微调的FRAME模型选择形成特定相互作用的碎片的频率与参考碎片相似,包括盐桥和π−π相互作用(图3d)。作者还测量了模型选择特定恢复参考碎片相互作用的碎片的能力;经过微调的模型恢复相互作用的概率为78%。相比之下,对接分数选择了比参考碎片更多的相互作用。这些额外的相互作用是否对结合有害尚不清楚。
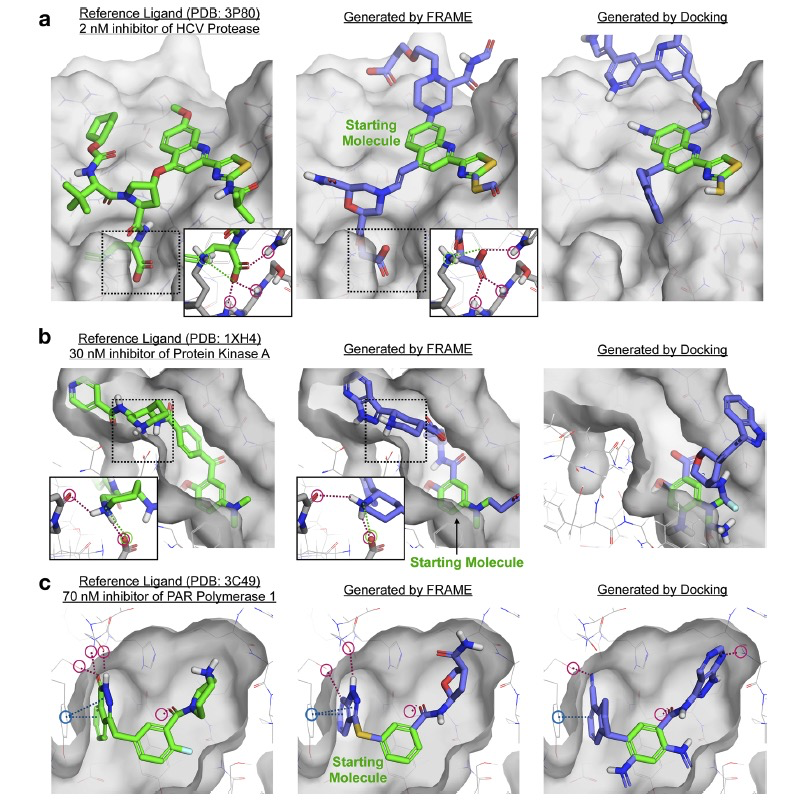
图 4
接下来,作者将FRAME应用于基于碎片的药物发现场景,其中多个碎片被附加到一个小的起始分子上以进行扩展。这个任务需要附着点选择和碎片选择模型的迭代应用,这比评分单个碎片要难得多。为了评估这项任务的性能,作者使用FRAME扩展了100个不同的蛋白质口袋中的100个分子,使用从参考配体中随机选取的小亚结构作为起始分子。模型为每个口袋产生了一个扩展分子。FRAME通常生成的配体形成关键相互作用,并且与参考配体类似地填充口袋,如与测试集蛋白一起的几个案例研究所示(图4)。作为一个具有挑战性的案例研究,作者使用FRAME来设计乙型肝炎病毒蛋白酶的抑制剂,它呈现出一个大部分浅的、暴露于溶剂的结合位点(图4a)。随机选择的起始分子距离需要高亲和力的位点很远,需要几个精确放置的碎片才能达到。有希望的是,FRAME能够向活性位点扩展,并将一个羧酸碎片放置在一个最佳位置以形成与催化位点残基的相互作用。由于FRAME学会模仿配体生长轨迹,它学会了即使在必须添加多个碎片后才能形成能量上有利的相互作用之前,也能以有益的方向扩展分子。相比之下,使用迭代对接生成的配体根本没有进入催化位点。标准对接分数只在每一步评估即时相互作用,因此迭代对接方法没有预见到需要多步添加多个碎片以形成有利相互作用的有利生长方向。
编译 | 曾全晨
审稿 | 王建民
参考资料
Powers A S, Yu H H, Suriana P, et al. Geometric deep learning for structure-based ligand design[J]. ACS Central Science, 2023.




 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง





