
1 介绍
由于深度学习技术能够在没有任何领域知识的情况下从原始输入中自动学习特征表示,因此它们引起了大量关注。到目前为止,已经发展出多种基于深度学习的方法来预测蛋白质–配体结合亲和力,这些方法可以根据所使用的信息分为基于结构的方法和基于序列的方法。
基于结构的方法将蛋白质–配体复合物处理为 3D 网格或分子图,例如 Pafnucy、OnionNet 、FAST和IGN。然而,蛋白质–配体复合物的3D网格或分子图表示在大规模结合亲和力预测任务中会导致巨大的计算成本。此外,这些基于结构的方法的性能在很大程度上依赖于复合物的高质量3D结构,这极大地限制了它们在实际任务中的应用。
基于序列的方法仅将蛋白质和配体的一维序列作为模型输入,例如 DeepDTA和DeepDTAF。众所周知,蛋白质结合口袋和配体之间的结构互补性显着影响蛋白质和配体之间的结合强度,然而现有的基于序列的方法通常只通过两个分离的模块来学习蛋白质结合口袋和配体的特征表示,并简单地将提取的特征向量连接起来,而不考虑它们之间的相互作用。
因此,为了充分考虑口袋和配体之间的相互作用,提出了一种新的基于交叉注意力机制的深度学习方法CAPLA,使用蛋白质和配体的序列信息来改进结合亲和力的预测。CAPLA 包含三个输入,即包含氨基酸类型、二级结构元素SSE和残基理化性质的蛋白质和口袋输入表示,以及配体SMILES串。利用交叉注意力机制捕捉口袋和配体之间的相互作用特征,然后采用扩张卷积分别学习蛋白质、口袋和配体的多尺度远程特征。
2 方法
数据集:选用PDBbind v.2016精炼集和通用集来获得训练集和验证集,采用了与Pafnucy相同的方式进行划分,即从精炼集中随机选择1000个复合物构成验证集,选择通用集中剩余的11906个复合物构成训练集。PDBbind v.2016核心集和CASF-2013数据集被用作两个基准测试集,分别命名为Test2016_290和Test2013_195,对于99%的来自两个测试集的蛋白质序列对相似度不超过60%。
为了评估CAPLA模型的泛化能力,选取CSAR-HIQ数据集作为独立测试集,该数据集包含两个子集,去除重叠的复合物,得到分别由51个和36个复合物组成的测试集,命名为CSAR-HiQ_51和CSAR-HiQ_36。此外,由于不同的样品具有不同的序列长度,将蛋白质、口袋和配体SMILES串的长度分别统一为1000、63、150,这样的长度设置可以覆盖数据库中约90%的数据。
CAPLA输入表示:蛋白质和口袋的输入内容相同,由氨基酸序列、二级结构状态SSE和残基理化性质组成。具体来说,蛋白质序列中通常含有20种不同的氨基酸和一种未知残基,因此这21种不同的残基被编码为21维one-hot向量。SSE的特征由DSSP算法从给定的PDB文件生成,包括八种状态:310-螺旋(G)、α-螺旋(H)、∏-螺旋(I)、β-链(E)、β桥(B)、β-turn(T)、bend(S)、coil(C),由8维one-hot向量表示。残基理化性质根据其侧链分为非极性、极性、酸性、碱性,以及根据其偶极子和侧链体积分为七种不同的聚类,由11维one-hot向量表示。因此,蛋白质每个残基都由40维one-hot向量编码。
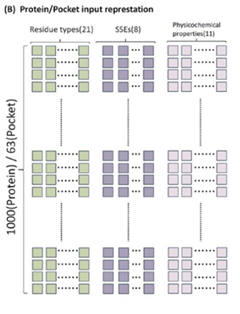
图1 蛋白质、蛋白质口袋输入表示
配体的SMILES字符串由64个字符组成,每个字符由一个特定的整数编码,这64个字符被编码为一个64维one-hot向量。
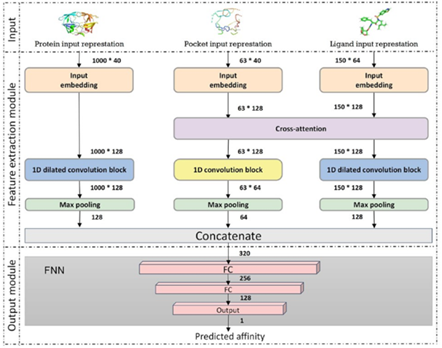
图2 CAPLA模型架构
CAPLA特征提取模块:首先,输入信息被传送到一个线性嵌入层,其作用是将稀疏的one-hot向量转化为密集向量,并将口袋和配体输入表示统一到与交叉注意力机制输入相同的维度,以获得 128维密集向量。
交叉注意力机制能够在两个单独的输入之间构建显式交互,以充分利用其相关性,因此采用交叉注意力机制来交叉提取蛋白质结合口袋和配体的特征。交叉注意力机制的定义如下:
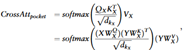
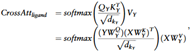
其中X和Y分别表示口袋和配体序列的嵌入矩阵;蛋白质的查询矩阵QX根据X计算,键矩阵KX和价值矩阵VX根据Y计算,配体相反;WKX、WVX、WQX、WKY、WVY、WQY是可学习的参数;dKX、dKY分别为矩阵KX、KY的维度;注意力得分矩阵softmax·V表示口袋序列中的每个氨基酸与配体串中的每个原子之间的相似性。采用了具有两个注意头的多头交叉注意机制,将两个平行交叉注意层的输出连接在一起。
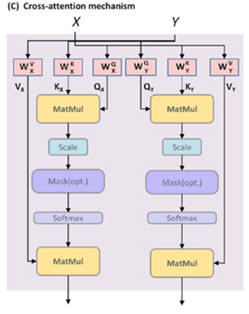
图3 交叉注意力机制实现流程
扩张卷积能够通过增加具有不同扩展率的核的感受野大小来聚合多尺度上下文信息,因此利用扩展卷积来分别捕获蛋白质和配体序列的多尺度远程分子相互作用。在蛋白质特征提取部分,扩展卷积块紧跟着嵌入层,包含4个扩展卷积层,核数分别为32、64、64和128,每个扩展卷积层通过设置1、2、4、8、16五种不同的扩展率来应用内核大小为3的卷积。配体特征提取部分,扩展卷积块遵循交叉注意力层,包含3个扩展卷积层,核数分别为32、64和128,每个扩展卷积层通过设置1、2、4、8四种不同的扩展率来应用核大小为3的卷积。
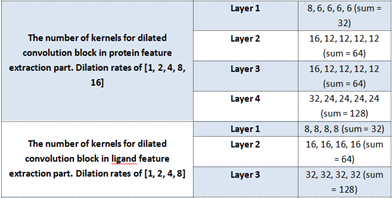
图4 扩张卷积kernel设置
此外,由于口袋是由蛋白质中的不连续序列组成的,在口袋特征提取部分利用1D传统卷积来捕获口袋序列的局部特征。随后,每个一维膨胀卷积块或一维传统卷积块后面都有一个最大池化层。
CAPLA输出模块:蛋白质、口袋、配体的三个特征提取部分的输出被连接起来形成一个长度为320的向量,输入FNN产生最终输出。FNN由两个全连接层(FC)和一个输出层组成。
3 结果
CAPLA的性能及其与先进方法的比较:CAPLA 在R、RMSE、MAE、SD、CI五个指标上都明显优于其他两种基于序列的方法(DeepDTA、DeepDTAF)和六种基于结构的方法(Pafnucy、OnionNet、FAST、IMCP-SF、GLI、IGN)。与基于序列的方法比较结果表明:CAPLA能够从序列水平信息中捕获更具判别性的相互作用特征,以预测结合亲和力。与基于结构的方法比较结果表明:仅利用序列信息仍足以准确预测结合亲和力,且具有更高的计算效率。此外,由于许多蛋白质仍然没有实验鉴定的蛋白质结合口袋,在这种情况下,应用预测的口袋来取代实验口袋作为模型的输入。利用P2Rank工具来预测训练集、验证集和Test2016_290测试集中的蛋白质口袋。首先,P2Rank预测每个蛋白质有多个口袋,选择得分最高的口袋作为结合口袋。然后,在训练集上对CAPLA进行再训练,并在Test2016_290测试集上进行测试。结果表明,当使用预测口袋作为输入时,CAPLA的预测性能会变差,但CAPLA-Pred的结果仍然优于六种基线方法。这也表明了CAPLA模型在没有实验口袋的情况下的普适性。
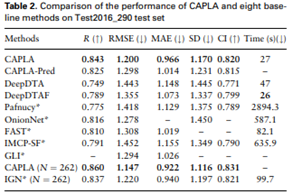
图5 CAPLA以及其他先进方法在Test2016_290上的性能
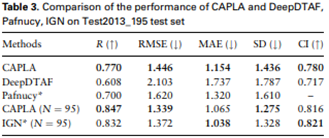
图6 CAPLA以及其他先进方法在Test2013_195上的性能
CAPLA独立来源测试:为了证明CAPLA的泛化能力,对来自独立来源的两个外部数据集进行了两次额外的测试,即CSAR-HIQ_51和CSAR-HIQ_36。由于CSAR数据库不直接提供蛋白质结合口袋,但CAPLA模型又利用口袋作为输入,因此PDB文件中提取配体一定半径内的残基作为口袋。为了确定最佳配体半径,建立了五个不同的半径(分别为9、10、11、12、13 Å),并评估了它们在CSAR-HIQ_51和CSAR-HIQ_36中对预测性能的影响。当配体半径设置为12 Å时,CAPLA在所有评估指标中实现了更好的权衡,而配体半径设置为13 Å时,模型性能开始下降。因此,提取了配体半径12 Å范围内的残基作为口袋。结果显示,CAPLA预测的亲和力与两个独立测试集的实验测量值吻合良好,R值分别为0.686和0.704。这表明,CAPLA能够很好地泛化到不同的独立数据。
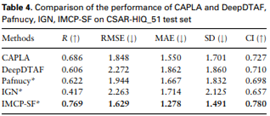
图7 CAPLA以及其他先进方法在CSAR-HIQ_53上的性能
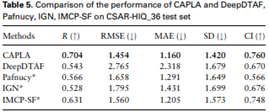
图8 CAPLA以及其他先进方法在CSAR-HIQ_36上的性能
CAPLA可解释性分析:应用t-分布式随机邻居嵌入(t-SNE)工具,将嵌入层、交叉注意力层和卷积层后分别提取的高维特征表示(213D)映射到二维空间上,可以看出,具有不同结合亲和力的样品在嵌入层之后的特征空间中重叠且无法区分,嵌入层只学习到口袋和配体的单独表征;在交叉注意力层之后结合亲和度相近的样本大部分聚集在一起,更重要的是,交叉注意力层之后的表征是口袋和配体的相互作用特征;卷积层后亲和力接近的样本聚类更明显,这表明卷积层也可以进一步学习有效的特征表示。这些结果证实了CAPLA的交叉注意力机制和卷积层学习到的特征表示可以有效提高不同蛋白质–配体结合亲和力的可区分性。
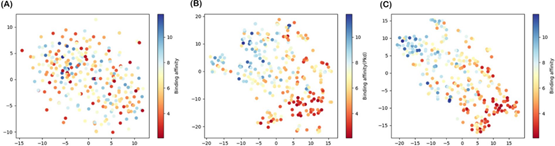
图9 可视化CAPLA嵌入层、交叉注意力层、卷积层之后学习到的特征分布
为了进一步说明CAPLA学到的内容,可视化了蛋白质结合口袋中所有残基的注意力得分,并分析了对预测蛋白质–配体结合亲和力有重大贡献的关键残基。利用交叉注意机制生成了复合物3EHY口袋的注意力分数(①分别提取每个口袋序列的两个注意头生成的两个交叉注意得分矩阵,其中查询矩阵Q是配体SMILES字符串的嵌入表示,键矩阵K是口袋序列的嵌入表示。②通过在查询方向上分别对每个口袋的两个注意力得分矩阵进行平均,为每个口袋生成两个位置得分向量,称为注意力图。)通常认为,在口袋的学习注意力图中,特定位点的某个残基的注意力得分越高,相应残基位点对蛋白质–配体结合的贡献就越大。事实上,配体与蛋白质的结合相互作用主要通过弱的非共价相互作用发生,例如疏水相互作用、氢键、范德华力,前两者对蛋白质–配体结合做出了重大贡献。正如预期的那样,CAPLA模型能够捕获通过氢键和/或疏水相互作用对结合有很大贡献的关键残基,包括头1在交叉注意机制中捕获的Leu181、Tyr240和Tyr242残基,以及头2捕获的His183、His196、Thr210、Thr215、His218和His222残基,其中一些残基与3EHY复合物的实验结构一致。
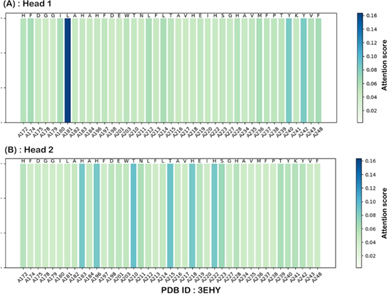
图10 头1和头2在交叉注意机制中学习到的复合物3EHY口袋的可视化注意力图
4 总结
本文开发了一种新的基于交叉注意机制的深度学习方法CAPLA,仅使用蛋白质和配体的序列信息来改进结合亲和力的预测。在各种基准数据集上的综合实验表明,CAPLA的预测能力优于其他最先进的基线方法。此外证明了CAPLA能够揭示口袋序列上的关键残基,这些残基通过交叉注意机制对蛋白质与配体的结合做出更大的贡献。
作者提出模型可以有进一步的提升:①CAPLA仅利用蛋白质和口袋的序列信息作为模型输入,因此可以考虑加入复合物的三维结构信息。②配体的输入表示仅由其SMILES字符串组成,可以考虑将更多的配体特征表示纳入到模型中,例如配体结构和原子的物理化学性质。





 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง





