今天为大家介绍的是来自Connor W. Coley团队的一篇论文。药物发现领域通常会定性或定量地分析结构-属性关系和活性景观,以指导化学空间的探索。这些分子属性景观的粗糙度(或平滑度)是最常研究的几何特性之一,因为它可以表征活性悬崖的存在,一般认为景观越粗糙,优化难度就越大。文章中介绍了一种描述分子属性景观粗糙度的通用量化指标——粗糙度指数(ROGI)。这个指数受到分形维数概念的启发,并且与机器学习模型在众多回归任务中的样本外误差有很强的相关性。

结构-活性关系(SARs)和活性景观常被用于指导分子优化过程(例如,先导化合物优化)中的化学空间探索。定量结构-活性关系(QSAR)模型利用机器学习(ML)模型和化学物质的数值表示来预测生物活性。QSAR概念通过结构-性质关系(SPR)及相关的定量结构-性质关系(QSPR)在化学研究中得到了广泛应用。粗糙度是结构-性质景观中最常讨论的属性之一,这可能是因为在药物设计中鉴定“活性悬崖”引起广泛的关注。活性悬崖指的是由于微小的结构变化而导致的化合物活性的急剧变化,这可能成为准确的QSPR模型开发中的一个主要障碍。因此,许多研究集中于活性悬崖的识别和预测,通常通过分析或预测匹配分子对中的亲和力差异来进行。显然,活性悬崖的存在(或不存在)与性质景观的粗糙度(或平滑度)内在相关。平滑的景观通常更受青睐,因为它们易于由化学家解释和预测,更容易被机器学习算法建模,并且有助于基于相似性的虚拟筛选。这些优点可能会影响发现过程中的战略决策,如优先考虑哪些化合物进行先导优化。
鉴于对量化描述结构-性质景观的兴趣,目前研究人员已经开发了不同的方法来分析它们。为了可视化性质景观,Peltason等人提出使用多维缩放将高维表示投影到二维平面上,并以三维景观的形式展示结构-性质景观。这些三维景观与结构-性质矩阵和分子网格图结合,提供了一个用于组织和分析的工具。图像分析技术也被用于根据粗糙度对三维性质景观进行分类,以及定义它们之间的相似性度量。
尽管目前已经开发了多种景观度量方法(如SALI,SARI,MODI),对分子性质景观的粗糙度的真正通用度量仍然缺失。SALI作为局部度量,不能以单一标量值捕捉分子性质景观的粗糙度。虽然SARI可以做到这一点,但它依赖于需要启发式设置的用户定义超参数,如不连续性评分的相似度阈值和标准化的参考数据集。最后,MODI主要适用于分类任务,扩展到回归任务一直具有挑战性。作者在这项工作中提出的一种新的粗糙度度量方法,适用于度量空间,特别是分子数据集。这个被称为粗糙度指数(ROGI)的新指标,能够捕捉标准化数据集的全局粗糙特征,并以一个介于零到一之间的单一标量值表示,其中零对应于平坦表面,而一对应于所有最近邻都显示极端相反性质值的表面。与上述大多数方法不同,一旦定义了分子表示和度量,ROGI就没有超参数。它自然适用于任何感兴趣属性的回归任务,以及二元分类任务。
方法部分
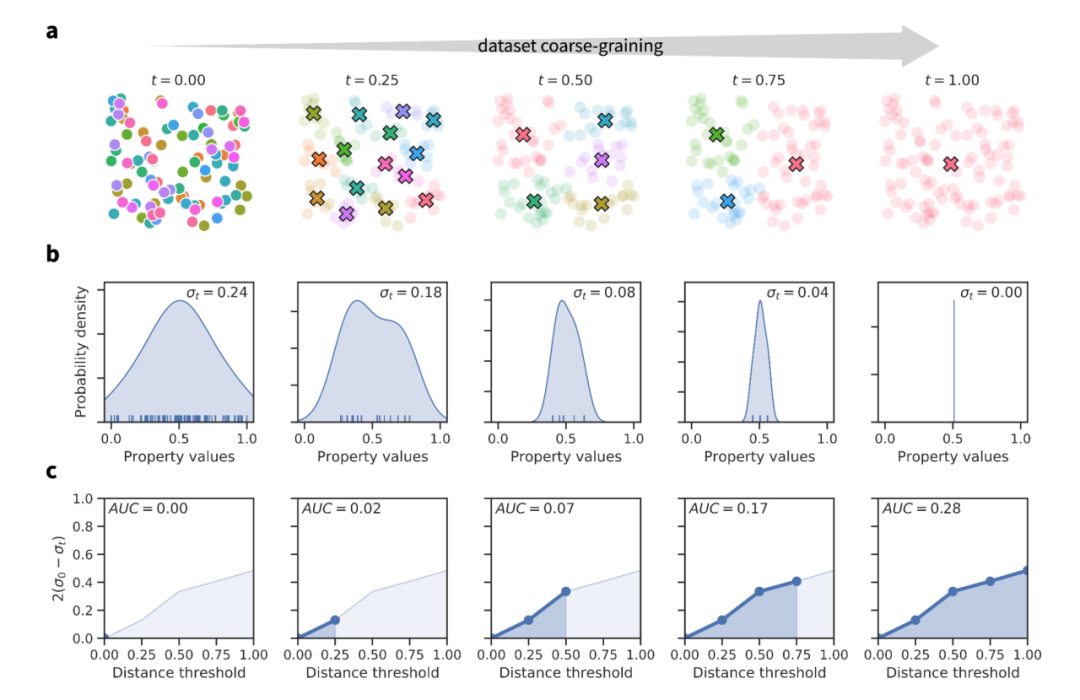
图 1
化学空间可以被定义为度量空间,其中每个分子都与一个表示相关联,而一个距离度量则定义了分子之间的不相似性。这种距离度量是非负的、对称的,并且在实际使用的大多数度量中满足三角不等式。有时,即使分子是不同的(例如,有限半径的二进制指纹或位碰撞),距离度量可能也是零,使得空间更适合被描述为伪度量空间。我们感兴趣的分子和材料属性通常是由连续变量描述的,如回归任务中的情况,机器学习算法试图构建模拟将分子映射到性质的底层函数。作者提出的ROGI的灵感部分来自分形维数的概念,这是一个比较对象某些属性随观测尺度变化的复杂性指标。例如,通过测量观测到的海岸线长度随测量单位减少的增加率(例如,使用越来越短的测量尺),海岸线的粗糙度可以通过其分形维数来量化。本质上,对某个对象采取越来越粗糙的观察方式,并观察其某些属性的变化率与对象的复杂性相关。同样地,为了描述分子属性景观的粗糙度,作者逐渐粗糙化分子数据集,并观察感兴趣的分子属性的分散度受到的影响。
粗糙度指数(ROGI)背后的直觉思路如图1所示。以一个分子数据集{xi}和相关的性质值{yi}为例,其中xi属于X空间,yi属于Y空间且为实数。假设数据集中所有分子之间的成对距离已被规范化,d(xi, xj)属于[0,1]对所有的xi, xj属于X空间。然后使用完全链接聚类方法对数据集进行聚类,根据不同的距离阈值t属于[0,1],使得一个聚类中任意两个元素之间的距离最多为t(见图1a)。给定Y是一个连续性质,作者使用分布的标准差σ来衡量离散度。对于每一个距离阈值t,考虑数据集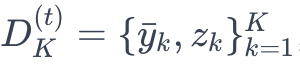 ,其中K是聚类的数量,
,其中K是聚类的数量, 是聚类k中的平均分子性质,z是聚类大小。加权标准差
是聚类k中的平均分子性质,z是聚类大小。加权标准差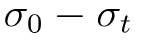 是根据权重
是根据权重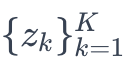 计算的(图1B)。这等同于给每个聚类的所有成员分配平均性质值然后计算整个数据集的标准差。在t = 0时,每个分子属于其独立的聚类,是原始数据集中值的标准差。当t = 1时,数据集被描述为一个具有零标准差的单一聚类。在t的中间值,我们有效地得到了数据集的粗粒化版本,其中每个聚类k由一个具有平均性质值的虚构平均分子表示(见图1a)。随着粗粒化分子数据集不断加深,可以监测离散度的损失
计算的(图1B)。这等同于给每个聚类的所有成员分配平均性质值然后计算整个数据集的标准差。在t = 0时,每个分子属于其独立的聚类,是原始数据集中值的标准差。当t = 1时,数据集被描述为一个具有零标准差的单一聚类。在t的中间值,我们有效地得到了数据集的粗粒化版本,其中每个聚类k由一个具有平均性质值的虚构平均分子表示(见图1a)。随着粗粒化分子数据集不断加深,可以监测离散度的损失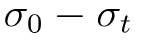 (见图1)。直觉上,如果相似的分子具有极其不同的性质值,它们会在低t值时被聚类,而跨聚类的离散度会迅速降低。相反,如果相似的分子具有相似的性质值,用它们的平均值替换会对整个性质跨聚类的离散度影响较小,因此
(见图1)。直觉上,如果相似的分子具有极其不同的性质值,它们会在低t值时被聚类,而跨聚类的离散度会迅速降低。相反,如果相似的分子具有相似的性质值,用它们的平均值替换会对整个性质跨聚类的离散度影响较小,因此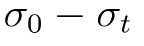 缓慢增加。为了衡量随着t增加离散度损失的速度,在0到1之间积分
缓慢增加。为了衡量随着t增加离散度损失的速度,在0到1之间积分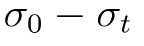 得到最后的ROGI表示:
得到最后的ROGI表示: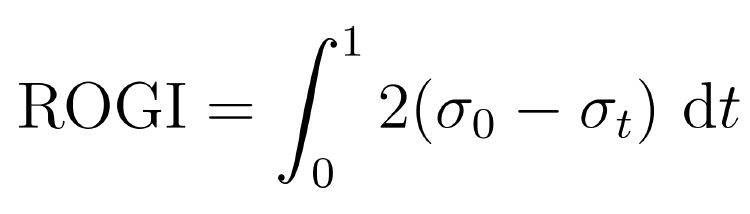 标准差是从归一化的性质值中获得的(见图1c)。需要注意的是,虽然ROGI最初是为回归问题设计的,但它也可以直接应用于二元分类。
标准差是从归一化的性质值中获得的(见图1c)。需要注意的是,虽然ROGI最初是为回归问题设计的,但它也可以直接应用于二元分类。
模型表现
作者通过测试ROGI与交叉验证模型误差的相关性来评估ROGI捕捉性质景观粗糙度的能力。他们在55个回归任务上进行了测试,并发现ROGI与所有测试的回归机器学习模型的预测误差有积极且通常很强的相关性。这种相关性在基于ZINC+GuacaMol随机样本子集的数据集和TDC中的药效学和毒理学数据集上尤其明显。ROGI值和模型误差之间的相关性通常超过0.8,有时甚至接近或超过0.9(图2)。尽管预期不同大小的数据集会因机器学习模型的性能而影响相关性,但TDC数据集的相关性通常很强。
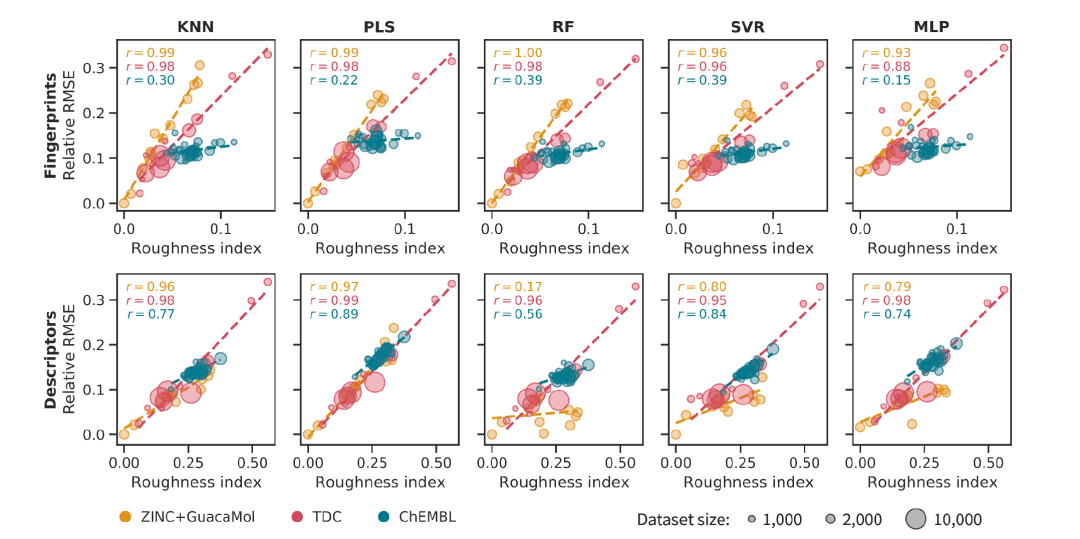
图 2
ZINC+GuacaMol这个数据集包含2000种分子,这些分子有不同的性质。作者发现,在大多数情况下,ROGI这个指数能够很好地预测机器学习模型在这些分子上的错误率。但是有一个例外,那就是当分子通过物理化学描述符来表示,并且用随机森林(RF)模型进行预测时,ROGI就不太能准确预测模型的错误率了。这可能是因为在这种情况下,RF模型能够有效地区分哪些描述符是有用的,哪些是没什么信息的。在另一个数据集ChEMBL中,当分子用物理化学描述符表示时,ROGI和模型错误率之间有中等到很强的相关性;但是当用指纹来表示分子时,这个相关性就弱多了。一个可能的原因是在ChEMBL数据集中,ROGI的数值变化范围和模型的错误率都比较小。这意味着要准确地预测模型错误,ROGI需要比现在更精准。使用指纹表示分子时,KNN模型在不同数据集上的预测误差(RMSE)范围有所不同。相比之下,使用描述符时,虽然RMSE的范围类似,但指纹表示的ROGI值分布更加集中,可能导致了相关性较低的问题。此外,使用指纹表示分子时得到的ROGI值通常比使用描述符得到的值要小,这可能是因为分子之间距离的分布方式不同。用指纹表示的分子之间的Tanimoto距离一般比用描述符计算出来的欧氏距离要大。如果分子间的距离小,ROGI就会表明表面比较粗糙;而距离大的话,ROGI就会显示表面较平滑。如果所有分子之间的距离都很大(即最大距离),ROGI会是零,表示我们没有足够的信息来判断分子性质表面的粗糙度。在实际应用ROGI时需要考虑到分子间距离的分布情况。
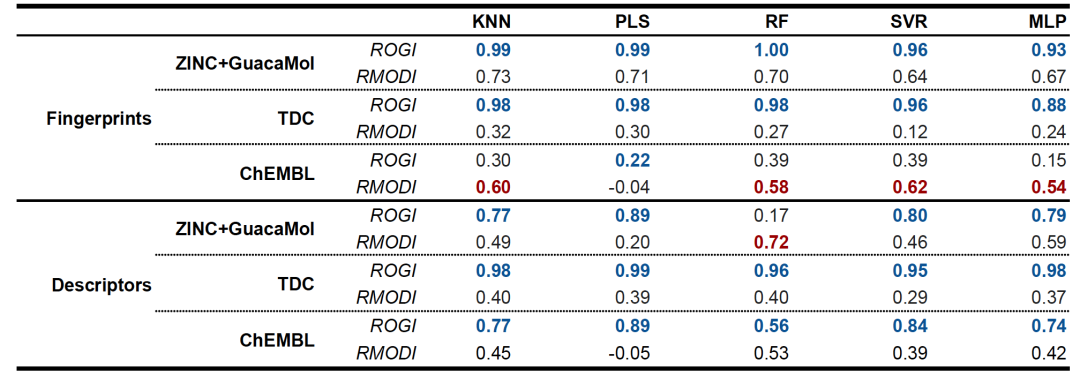
表 1
作者将ROGI与现有的其他方法进行了比较。他们使用ChEMBL数据集评估了SARI(一种用于蛋白质-配体结合亲和力的指标)与模型误差之间的相关性。发现SARI与模型误差之间的相关性普遍低于ROGI。他们还使用回归模型可塑性指数(RMODI)对所有数据集进行了相同的分析。RMODI得到的与模型误差的相关性高于SARI。然而,除了5个特定情况外(在ChEMBL数据集的指纹表示中有4个,ZINC+GuacaMol数据集的描述符和RF表示中有1个),ROGI一致地显示出更强的相关性(表1)。
编译 | 曾全晨
审稿 | 王建民
参考资料
Aldeghi, M., Graff, D. E., Frey, N., Morrone, J. A., Pyzer-Knapp, E. O., Jordan, K. E., & Coley, C. W. (2022). Roughness of molecular property landscapes and its impact on modellability. Journal of Chemical Information and Modeling, 62(19), 4660-4671.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


