今天为大家介绍的是来自Xutong Li, Xiaomin Luo和Mingyue Zheng团队的一篇论文。先导化合物优化是药物发现领域的一个关键挑战。目前,这一领域大部分还是依赖于假设和药物化学家的先前经验。作者为了应对这一挑战,提出了一种基于物理信息图注意力机制的配对结合比较网络(PBCNet),专门用于对同类配体的相对结合亲和力进行排名。通过在两个独立数据集上的基准测试, PBCNet在预测准确性和计算效率方面都显示出了显著的优势。

最近the Critical Assessment of Computational Hit-finding Experiments(CACHE),一个公共基准测试项目,受到了计算化学界和制药行业的关注。CACHE的目的是提升寻找小分子药物的效率。然而,从找到潜在的药物分子到优化它们成为实际的药物过程,目前仍然很依赖于药物化学家的经验和假设。先导优化的目标是设计出更高亲和力的配体,同时保持其他重要的化学性质。在这个优化过程中,会产生一系列类似的配体,它们通常有相同的核心结构,只在一些小的部分(取代基团)上有所不同。这个优化过程需要涵盖成百上千种化合物,因此需要大量的实验评估资源。因此,开发计算上的预测工具变得非常重要,这些工具可以帮助加速药物的发现过程。通过减少设计、制造、测试和分析的循环次数,这些工具有助于快速获得具有理想亲和力和性质特征的化合物。
在过去几十年里,为了优化先导化合物,研究人员提出了许多计算相对结合自由能的模拟方法。这些方法得益于更好的力场和采样算法的发展。例如, FEP是一种被广泛使用的计算方法,它在特定系统上达到了接近1千卡/摩尔的精确度。但是,FEP方法也有它的局限性,比如它的准确性很依赖于系统准备的过程,计算成本高等。另一种RBFE模拟方法是终点采样方法,比如分子力学通用Born表面积(MMGB/SA)。终点采样方法降低了计算需求,但同时也牺牲了一些性能。近年来在药物先导优化领域出现了一些基于人工智能(AI)模型。首先,受到相对结合自由能(RBFE)模拟方法的启发,Jiménez-Luna等人提出了一种卷积孪生神经网络DeltaDelta。这个模型的目的是直接确定两个结合配体之间的RBFE。McNutt等人最近提出了一种多任务卷积孪生神经网络模型。他们的方法是使用两个输入配体表示之间的明显差异作为分子对表示,与DeltaDelta相比,他们的模型并没有显示出显著的性能提升。尽管新型AI模型提供了一些创新思路,但它们在实际应用中仍面临着一定的挑战和局限性。因此开发一种高效准确的方法来指导药物先导优化是一个迫切需求。为此,作者们提出了一种名为配对结合比较网络(Pairwise Binding Comparison Network,简称PBCNet)的方法。这个方法基于物理信息图注意力机制,特别设计用于比较同类一系列配体之间的相对结合亲和力。
PBCNet引入了几种以物理为导向的建模策略。这是基于一个理念:分子间相互作用总是遵循严格的几何规则。通过解读研究,作者们发现赋予蛋白质-配体原子对较高的注意力得分可能表明更显著的相互作用。换句话说,如果PBCNet在分析蛋白质和药物分子结合时,给某些原子对分配了较高的分数,这可能意味着这些原子对之间的相互作用特别重要。此外,PBCNet还专注于那些能形成分子间相互作用的分子亚结构。这意味着它不仅分析整个分子,还关注分子中那些对结合特别重要的部分。通过这种方式,PBCNet能够更准确地预测不同药物分子与目标蛋白之间的结合亲和力。
模型
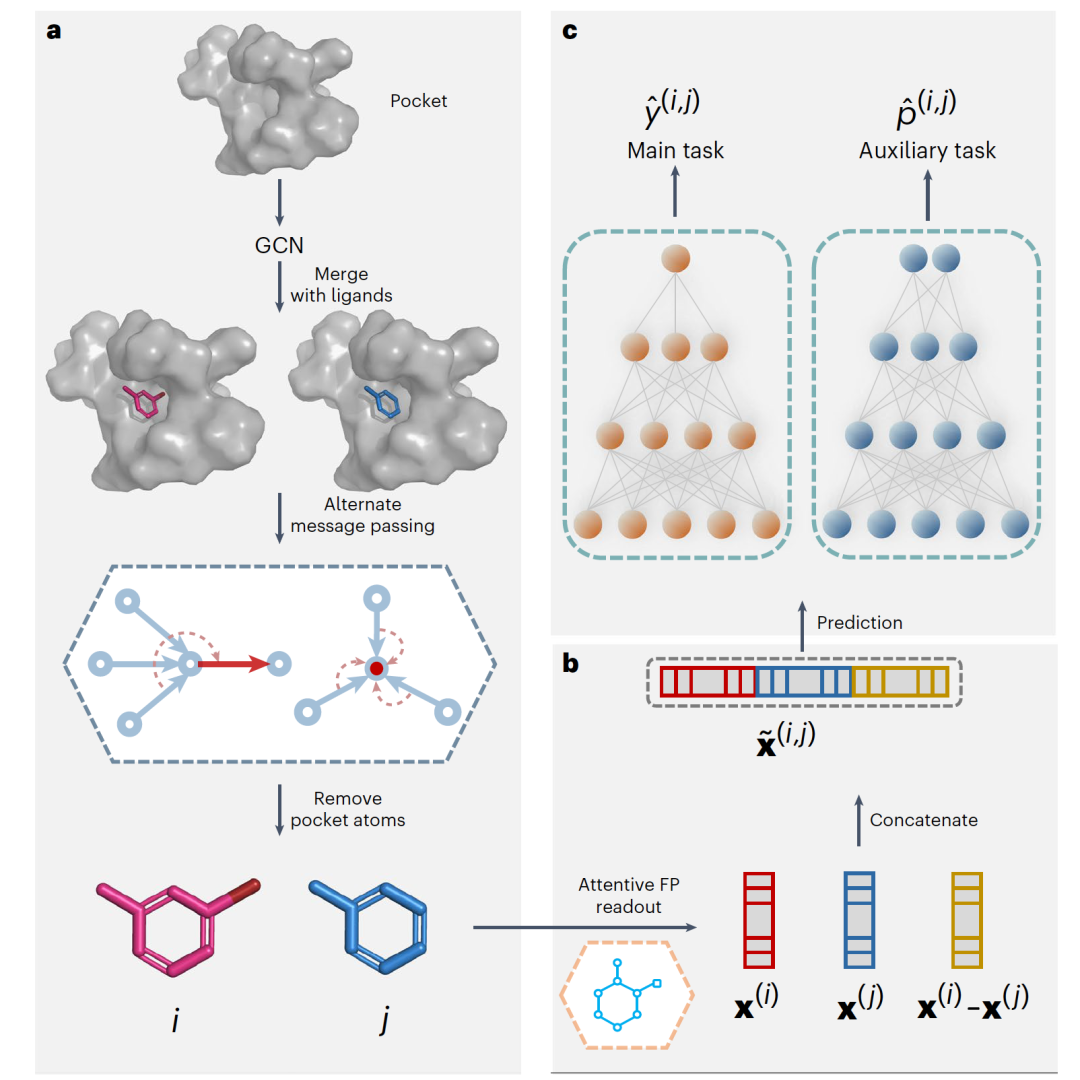
图 1
模型整体结构如图1所示,PBCnet分为三个子阶段。1. 信息传递阶段:PBCNet的输入是成对口袋-配体复合物,这里的配体是一组结构类似物,而口袋部分完全相同。选取与配体最近距离小于或等于8.0埃的氨基酸残基作为蛋白质口袋。首先,使用图卷积网络(GCN)更新蛋白质口袋的原子表示。然后,将更新后的蛋白质口袋与两个配体结合起来,构建原子对之间距离小于5.0埃的边。接下来,使用信息传递网络在分子图中传递信息。最后,从分子图中移除口袋,只保留配体。2. 读出阶段:这一阶段的目标是获取分子的表示(图级别)。在这一阶段,通过Attentive FP读出操作计算配体的分子表示(图1中的x(i)和x(j))。然后得到成对信息表示。3. 预测阶段:在这一阶段,通过优化两个任务的损失来学习分子对表示:(1)亲和力差异的预测和(2)配体i的亲和力大于配体j的概率。这是通过三层前馈神经网络的两个独立分支完成的(参见模型训练和微调过程部分)。在推理过程中,研究人员只需要提供一对结构相似的小分子到同一蛋白质的对接位向,就可以获得预测的相对结合亲和力。
Zero-shot学习实验结果
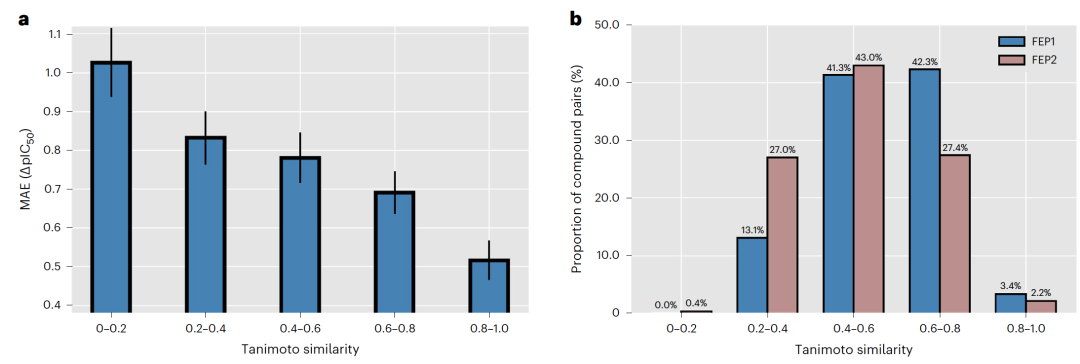
图 2
零样本学习是指将训练实例中包含的知识转移到测试实例预测的任务上。这种评估旨在模拟先导优化活动的早期阶段,那时通常缺乏已知活性的化合物。在这项分析中,作者使用了两个保留测试集(FEP1和FEP2集)来评估PBCNet的零样本性能,并选取了Schrödinger的FEP+、Schrödinger的Glide SP、MM-GB/SA,以及四个基于AI的模型(DeltaDelta、Default2018、Dense和PIGNet)作为基准。对于每个测试系列,作者随机选取一个配体作为参考配体,用于推断其余配体的绝对结合亲和力。这个过程重复了十次,以避免随机性。为了评估这些方法的性能,使用了皮尔逊相关系数(R)、斯皮尔曼等级相关系数(ρ)和成对均方根误差(r.m.s.e.pw)。
结果显示,PBCNet的性能明显优于除FEP+之外的所有基线方法(具体结果参考论文附录)。在FEP1测试集上,PBCNet的准确度达到了1.11 kcal/mol,接近于1 kcal/mol的理想标准,并且在FEP2测试集上实现了最低的平均成对均方根误差(r.m.s.e.pw)1.49 kcal/mol。此外,PBCNet的性能表现出稳健性,与其他高通量基线方法相比,在所有测试系列中表现更为稳定。这从斯皮尔曼等级相关系数可以看出;PBCNet在所有测试系列中的相关性都超过0.30,而其他高通量基线方法的相关性波动较大。PBCNet在FEP1测试集上的性能优于FEP2集,可能是由于FEP2集中有一些超出领域的样本。作为先导优化的模型,PBCNet旨在推断结构类似物的活性差异。为了与应用场景保持一致,训练集由Tanimoto相似度得分高于0.6的分子对组成。图2a显示了模型准确度与分子相似度之间的关系,可以观察到明显的负相关性。因此,PBCNet的相似度依赖性能是可以预料的,因为识别不同结构的分子更多地与虚拟筛选相关,而不是先导优化。相应地,为虚拟筛选设计的方法和模型在先导优化方面表现通常较差,例如在这里评估的Glide和PIGNet。作者还统计了FEP1和FEP2测试集中不同相似度得分的配体对的比例。图2b显示,在FEP2集中,Tanimoto相似度得分低于0.6的分子对比例显著高于FEP1集(70.4%对54.4%),这可能导致了模型在两个测试集上的性能差异。然而,PBCNet在FEP2测试集上的排名性能仍然超过了所有基线方法,除了FEP+。
Few-shot学习实验结果
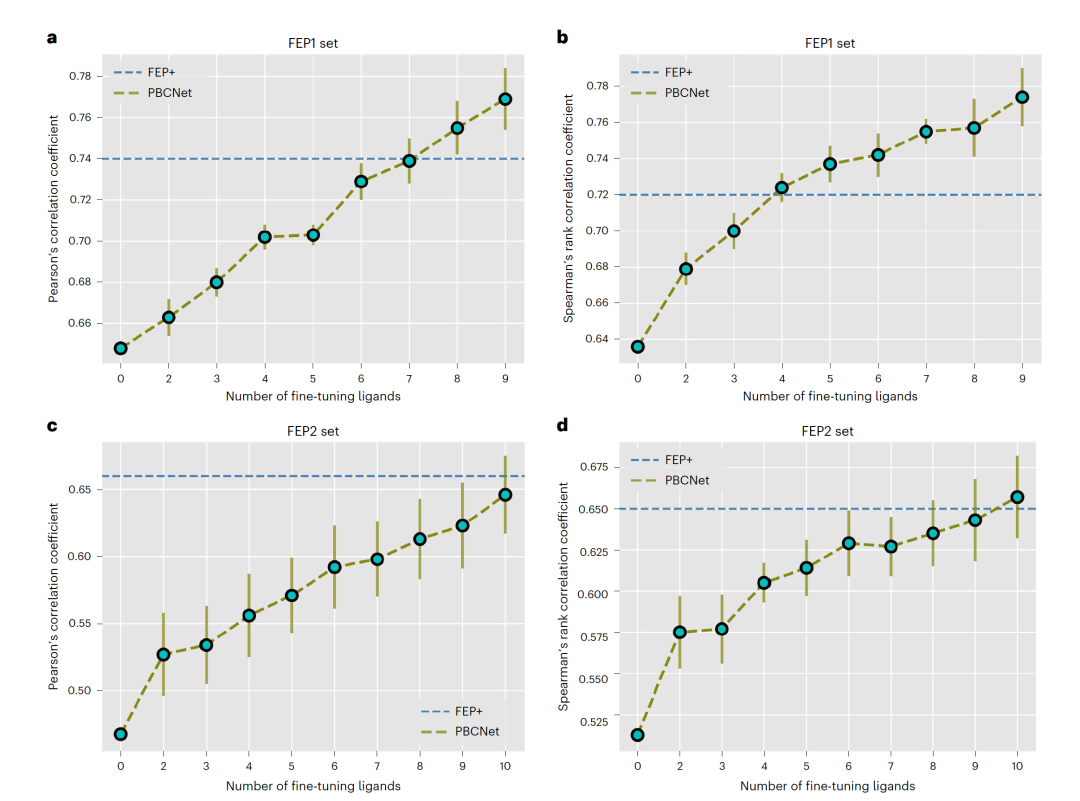
图 3
随后作者讨论了PBCNet在Few-shot learning情境下的表现及其与FEP+的比较。首先,作者指出PBCNet相对于FEP+在排名能力上的不足之处在于FEP+能够采样多种结合构象,而其他方法只使用单一快照,导致对分子结合过程的信息不够全面。然而,PBCNet在实际应用中对FEP+有两个优势:1. 高通量处理能力:PBCNet不受分子处理量的限制,允许全面探索先导优化。根据公开信息,运行FEP+每天处理四次变化需要八个Nvidia GTX-780 GPU,而PBCNet只需要使用一个Nvidia V100 GPU就能在0.9秒内计算一次变化。粗略估计,PBCNet的速度比FEP+快约10万倍。2. 灵活性:在先导优化过程中,新生成的结合亲和力数据可以用于微调PBCNet。为此使用了少样本学习。在每个测试的同类配体系列中,随机选择几个(大约2-10个)已知结合亲和力的配体作为微调配体,这些也在推断阶段作为参考配体。剩余的配体是待测试的配体。为避免随机性,重复上述过程十次。在新测试系列上,经过微调的模型表现在图3中进行了总结。图3显示少样本学习策略显著提升了PBCNet的性能,且随着微调配体数量的增加,性能提升。作者发现,微调后,PBCNet的排名能力与FEP+相当。例如,使用四个配体微调的PBCNet在FEP1集上的斯皮尔曼等级相关系数甚至超过了FEP+(0.724对0.720)。
模型的可解释能力
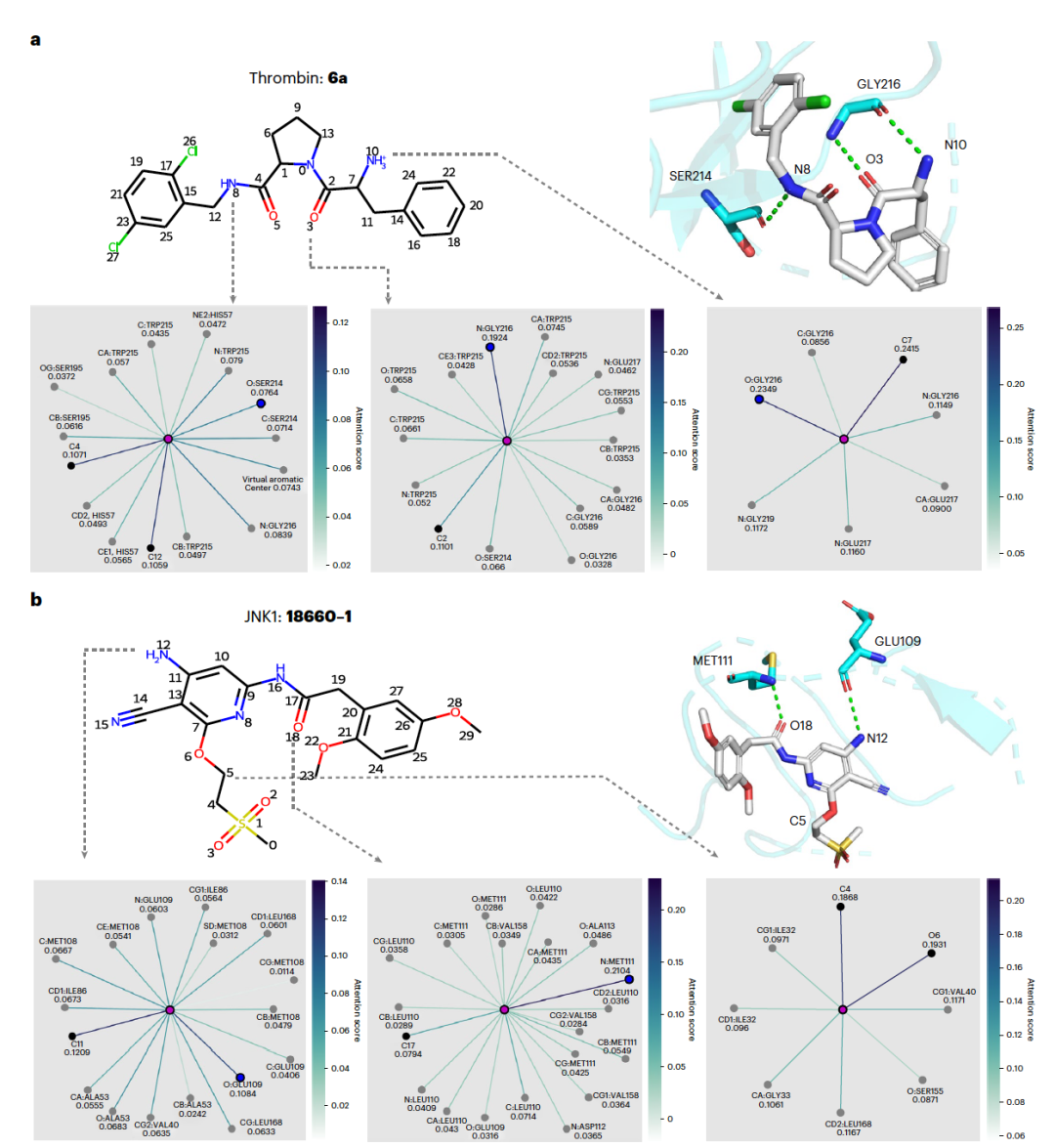
图 4
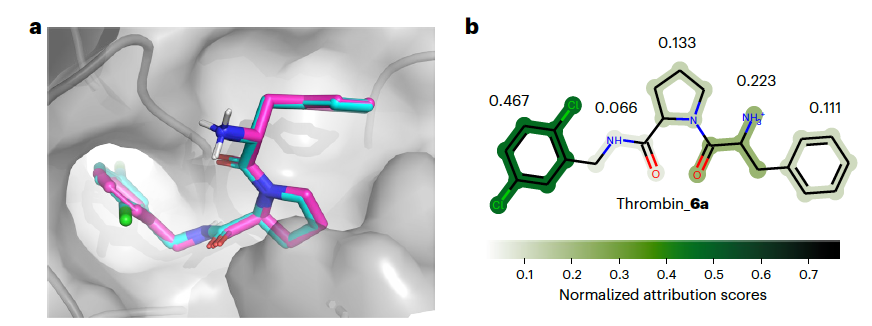
图 5
随后作者讨论了PBCNet在预测过程中如何工作,特别是它如何通过关注分子间的关键相互作用来做出预测。由于PBCNet是基于注意力机制的,因此原子对之间的注意力分数可以视为其重要性的衡量。一个强大的模型应该能够给形成关键分子间相互作用的原子对分配高分。为了阐明这一点,作者进行了一个案例研究,他们选取了FEP1测试集中的两种不同配体,专注于识别氢键,氢键是一种至关重要且常见的分子间相互作用。首先,他们使用Schrödinger2020计算了配体和蛋白质之间的分子间相互作用。因为氢原子的位置在很大程度上取决于用于添加氢原子的程序,所以他们没有考虑氢原子。对于氢键供体,他们选择了与氢原子共价连接的重原子进行进一步分析。这些操作的结果在图4中进行了展示。对于凝血酶系列中的化合物6a:该化合物与目标蛋白形成了三个氢键,位置分别在3、8和10(如图4a所示)。PBCNet强调了位于3和10位置的氢键,而共价键也被强调。这与化学先验知识一致,即配体原子的化学环境主要由与其共价连接的原子和参与关键分子间相互作用的蛋白质原子决定。然而,位于8位置的计算出的氢键没有被强调,可能是因为那里的酰胺供体氢原子具有相对较弱的氢键供体特性。对于JNK1系列中的化合物18660-1:这个化合物在12和18位置与目标形成了两个氢键(如图4b所示),这些位置都被PBCNet强调。此外,位于5位置的碳原子(根据Schrödinger的计算,这个位置没有形成任何关键的分子间相互作用)被选为负样本。可以清楚地看到,只有共价键被分配了相对较高的注意力分数,而虚拟距离键的注意力分数小且值一致。
药物化学家更倾向于从化学意义上的片段(亚结构)而非单个原子来探究分子的属性。因此,研究者扩展了他们的分析,以包括亚结构级别的可解释性。在这项分析中,作者采用了亚结构掩码解释(Substructure Mask Explanation, SME)方法(类似Shapley Value)。假设模型对一个化合物的预测值为y。然后,使用BRICS方法将化合物分割成亚结构。在读出阶段,依次掩盖每个亚结构的原子的表示,从而得到相应的预测值。当预测值代表化合物的活性时,如果某个亚结构掩盖后相比降低得更多,就认为相应的亚结构在模型预测中扮演着更关键的角色。作为案例研究,他们选取了凝血酶系统中的化合物6a,并使用化合物1a作为参考配体来说明PBCNet对化合物6a活性的预测(见图5a)。使用BRICS方法,将化合物6a分割成七个亚结构,酰胺组被分为两个不同的亚结构。为了给药物化学家提供更直观的表示,他们手动将酰胺组合并为一个整体。这一过程可视化展示在图5b中。结果显示,Sub4和Sub1对预测结果影响最大。PBCNet旨在预测配体对的相对结合亲和力,这主要来自一对配体的不同亚结构。Sub4作为化合物6a与化合物1a结构上的偏差部分被强调,这表明PBCNet确实捕捉到了输入配体之间的结构差异。此外,如图4a所示,Sub1与蛋白形成了两个氢键,因此Sub1的强调也意味着PBCNet专注于形成分子间相互作用的关键分子部分。
编译 | 曾全晨
审稿 | 王建民
参考资料
Yu, J., Li, Z., Chen, G. et al. Computing the relative binding affinity of ligands based on a pairwise binding comparison network. Nat Comput Sci 3, 860–872 (2023).
https://doi.org/10.1038/s43588-023-00529-9



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


