今天为大家介绍的是来自Elena Papaleo团队的一篇论文。论文讨论了一种名为PDBminer的开源软件包,它旨在简化和加速蛋白质结构的识别和选择过程,减少错误。

结构生物学对药物发现、基础生物学研究以及生物技术和生物工程有重要影响。结构生物学技术被用于研究蛋白质的各种功能,为生物数据提供机制解释。例如,科学家们利用这些方法研究突变、蛋白质之间的相互作用、配体结合、功能性基序和变化等。在这一背景下,蛋白质结构数据库,如PDB,对结构生物学家和生物信息学家来说至关重要。尽管传统上依赖于实验解决的结构,但蛋白质结构的解析既耗时又昂贵。目前的技术并不能解析所有蛋白质,这意味着与已发现的蛋白质序列相比,可用的蛋白质结构较少。计算结构预测工具试图弥补这一差距,依赖于PDB中的现有结构进行训练和验证。自2021年以来,随着AlphaFold2和RoseTTAFold等工具的引入,结构预测的质量大幅提高,预测结果变得更加可靠。AlphaFold蛋白质结构数据库的发布降低了入门门槛,让许多非计算科学家也能利用这些预测结构。这些结构信息在3D-Beacons上方便地提供,后者是一个提供结构模型程序化访问的PDBe知识库网络服务器。然而,选择用于后续分析的蛋白质结构并非易事。为此作者介绍了PDBminer,这是一个帮助选择用于分析或比较的蛋白质结构的工具,它挖掘PDB和AlphaFoldDB。PDBminer为用户提供信息,如目标蛋白质结构所覆盖的氨基酸范围(不论PDB文件中的编号如何)、蛋白质结构本身的质量信息、与其他蛋白、核酸链和配体的复合物细节等信息。
数据集特点
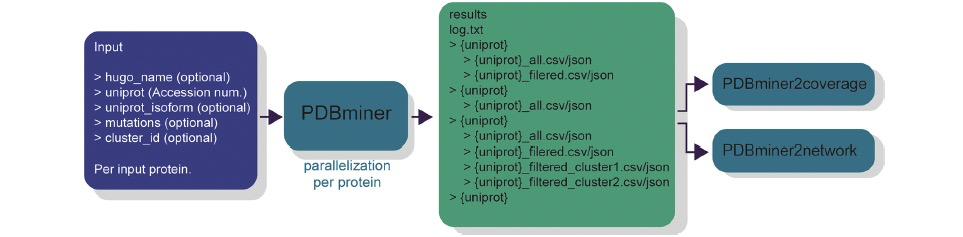
图 1
PDBminer的主要作用是自动化并简化搜索可用的结构数据库的任务。它接受UniProt访问号作为输入,并生成一个输出文件,列出了该蛋白质的所有可用结构及其相应的详细信息。它利用3D-Beacons、UniProt、PDBe和AlphafoldDB的APIs来识别合适的PDB结构及相关的元数据,如存放日期、实验方法和分辨率。找到的实验结构基于其元数据进行排名。每个实验结构序列使用成对比对与UniProt的参考序列对齐,以注释缺失残基、与UniProt序列的偏差和突变。如果有特定的感兴趣突变,可以在输入中包括它们,并在这种情况下,输出被过滤以仅包括覆盖定义的突变位点的结构。
PDBminer可以通过命令行选项直接运行,也可以使用配置文件来详细设置运行条件(图1)。使用命令行适合简单搜索,而使用配置文件则更适合于查找多个蛋白质的结构或覆盖突变簇的结构。对于配置文件或命令行中的每个UniProt访问号,PDBminer使用3D-Beacons数据库或PDBe来识别与特定蛋白质相关的所有PDB结构,并访问其元数据。如果3D-Beacons数据库中没有该蛋白质的可用条目,PDBminer将查询UniProt知识库和PDBe以获取可用结构列表和元数据。PDBminer将相关的元数据分配给每个结构,包括存放日期、解析结构所用的实验方法,以及该方法是X射线晶体学或Cryo-EM的结构分辨率。如果PDBminer需要利用UniProt来识别可用结构,最新发布的结构可能不会出现在输出中,因为它们在PDB上的发布时间和在UniProt上的可用性之间存在延迟。在这种情况下,会向用户发出警告。此外,PDBminer还识别AlphaFoldDB中可用的最新AlphaFold模型,并为AlphaFold模型提供相应的元数据,注明实验方法为预测。
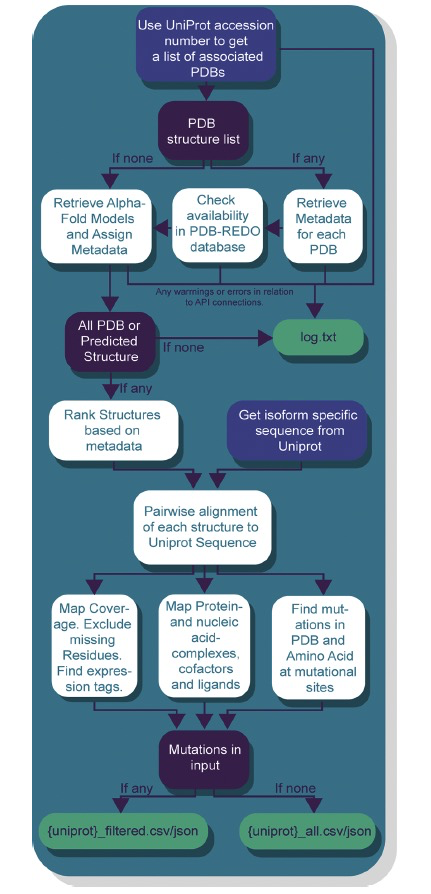
图 2
PDBminer根据元数据对可用的PDB或AlphaFold结构进行排名。AlphaFold模型始终排在第一位,以便与实验结构进行容易的比较。其余结构随后根据以前发布的最佳结果进行排名。排名使用的实验方法按以下顺序:X射线晶体学、Cryo-EM、NMR,然后是其他较少使用的方法,如中子衍射和纤维衍射。这些信息都可在输出文件中找到,允许用户根据需要进行筛选。使用相同方法的所有结构则根据X射线晶体学和Cryo-EM的分辨率进行内部排名。如果结构分辨率相同,则按各自的存放日期进行排名,优先考虑较新的结构。NMR和其他方法的条目排名由存放日期确定。如果一个蛋白质既没有与之相关的实验结构,也没有AlphaFold数据库中的AlphaFold预测,那么唯一的输出将是一个指明这一点的文件(图2)。
可视化部分
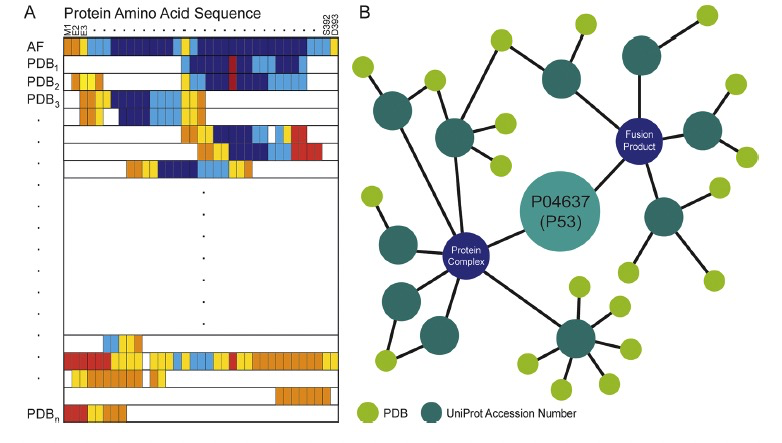
图 3
PDBminer提供了两个可视化工具:PDBminer2coverage和PDBminer2network。
PDBminer2coverage 是一个命令行工具,用于生成一个图表(图3A),提供PDBminer生成的蛋白质结构的结构覆盖范围的视觉概览。图表的x轴显示蛋白质的规范序列,而y轴显示覆盖这一序列的结构模型。工具会用颜色标记UniProt序列中每个结构所覆盖的部分,未覆盖的区域则为白色,包括缺失的残基。当一个结构中有多个链对应于感兴趣的蛋白时,该工具会分别注释每个链。图表的着色对应于AlphaFold模型的pLDDT分数或实验结构的b因子(如果可用)。对于pLDDT分数,使用了AlphaFoldDB的常规颜色方案,对于PDB结构,残基根据b因子着色,同样的颜色方案允许将最低值的b因子标记为最高质量。用户可以设置颜色的默认值和阈值。此外,PDB文件中编码的蛋白质序列与UniProt序列的任何差异都以红色突出显示,便于检查突变的存在。PDBminer2network 则可视化PDBminer找到的蛋白质复合物,通过创建网络图表来实现(图3B)。网络图将感兴趣的蛋白质通过其UniProt访问号放在中心,并分支出其他节点。每个节点进一步分支到通过其UniProt访问号识别的结合蛋白质。
PDBminer地址
http://github.com/ELELAB/PDBminer
编译 | 曾全晨
审稿 | 王建民
参考资料
Degn, K., Beltrame, L., Tiberti, M., & Papaleo, E. (2023). PDBminer to Find and Annotate Protein Structures for Computational Analysis. bioRxiv, 2023-05.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


