点击蓝字 关注我们
AItellU
编者 | 刹那芳华
制版 | 戈戈

第一作者:Ana Sanchez-Fernandez
通讯作者:Günter Klambauer
第一单位:约翰·开普勒林茨大学
DOI:10.1038/s41467-023-42328-w
1
全文一句话速览
本文利用多模态对比学习范式,通过生物图像和分子结构编码器将生物图像和化学结构嵌入到一个统一的空间,实现查询化学分子对应功能的正确生物图像。
2
研究背景
生物成像数据库目前的查询机制局限于根据文本注释查询,而无法通过化学结构进行关联查询。然而,将生物成像和化学结构嵌入统一的空间,并建立共同的嵌入表示,可以实现通过化学结构查询生物成像数据库。为了实现这一目标,作者借鉴了多模态对比学习的思想,使用生物成像和分子结构编码器将生物成像和化学结构嵌入到同一空间中。根据实验结果,基于多模态对比学习的检索系统的准确率比随机基线高出70倍以上,能够准确识别给定化学结构对应的生物图像。此外,CLOOME中的生物图像编码器在药物发现的活性预测、分子分类和作用机制识别等各种预测任务中也展现出良好的迁移性。
3
全文亮点
1.开发CLOOME新方法,可以用化学结构查询大型生物影像数据库
4
图文解析
成像和化学数据库检索系统
CLOOME能够通过化学结构查询大型化学数据库,并通过显微镜图像查询生物成像数据库。此外,作者还展示了CLOOME在活性预测、分子分类和作用机制识别等药物发现领域的预测任务中的转移能力。
具体来说,在这个检索系统中,作者使用了多模态对比学习的方法,将生物成像和化学结构嵌入到一个统一的空间中,通过生物成像编码器和分子结构编码器实现(图1)。这个共享的嵌入空间允许使用化学结构查询生物成像数据库,这些化学结构能够引起不同表型效应。实验结果表明,基于多模态对比学习的检索系统的准确率比随机基线高出70倍以上,能够准确识别与给定化学结构对应的生物成像。此外,CLOOME中的生物成像编码器在药物发现的活性预测、分子分类和作用机制识别等不同预测任务中表现出很好的迁移能力。
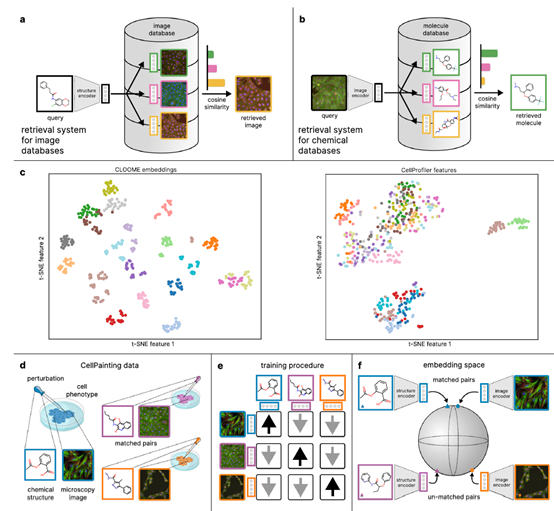
图1 CLOOME框架概述
生物活性预测作为下游任务
实验中使用了由CLOOME生成的图像嵌入向量来预测化合物在特定测定中的活性,并仅使用逻辑回归模型进行拟合。在预测这些活性时,CLOOME的性能超过了完全监督的方法,例如M-CNN和SC-CNN,评价指标为AUC。此外,CLOOME的图像编码器在药物发现中的其他预测任务,如图像分类和作用机制识别中也表现出了良好的迁移能力。
在具体的实验中,使用了Cell Painting数据集的子集,包括284,035张图像和10,574种化合物,涉及209个生物活性预测任务。在实验中,使用CLOOME通过对嵌入图像的预测值进行逻辑回归拟合,使用AUC作为评估指标。实验结果显示,CLOOME的预测性能非常好,并且超过了其他基准方法,这表明CLOOME生成的图像嵌入向量对于下游的生物活性预测任务具有较好的迁移能力(表1)。
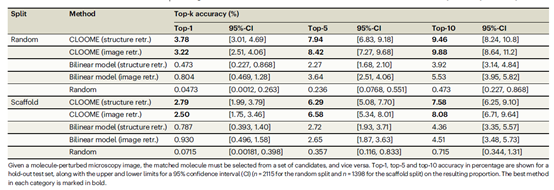
表1 候选药物的检索任务
零样本图像到图像分子分类
CLOOME在零样本图像转图像的分子分类和作用机制分类任务中的应用。通过将待查询图像与具有已知分子或作用机制的代表性图像进行比较,CLOOME能够推断待查询图像中应用的化合物或作用机制,并实现准确的分类。
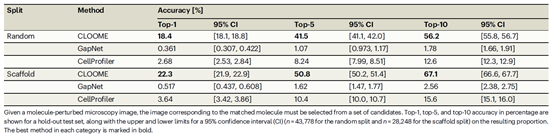
表2 零样本图像到图像分子分类任务结果
5
通讯作者介绍
Günter Klambauer,约翰·开普勒林茨大学机器学习研究所副教授。研究方向:1.药物发现的机器学习方法;2.生命科学数据分析和生物信息学。
6
编者有话说
对于带有化学结构的查询,对比学习可以解锁生物成像数据库。使用多模态对比学习方法,可以将生物成像和化学结构嵌入到一个统一的空间中,从而实现生物成像数据库与引发不同表型效应的化学结构之间的查询。通过多模态对比学习构建的检索系统可以比随机基线提高超过70倍的准确率,可以识别与给定化学结构对应的正确生物成像。此外,CLOOME中的生物成像编码器在药物发现的各种预测任务中都显示出迁移性,例如活性预测、分子分类和作用机制识别。
转载须知
【原创文章】AItellU原创文章,欢迎个人转发分享,未经允许禁止转载,所刊登的所有作品的著作权均为AItellU所有。AItellU保留所有法定权利,违者必究。

投稿、转载及合作邮箱:
zjhuangjunjie@163.com
原文链接:
https://www.nature.com/articles/s41467-023-42328-w
点击下方蓝字阅读原文



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


