
编译 | 刘洛涛
审核 | 黄 锋
今天给大家分享的是山东大学团队在今年发表在nature
communications上的一篇论文:“MarsGT: Multi-omics analysis for rare population inference using single-cell graph transformer”,利用单细胞图Transformer进行罕见细胞群推断的多组学分析。罕见细胞群是肿瘤进展和治疗反应的关键,提供了潜在的干预靶点。然而,它们的计算识别和分析往往落后于主要的细胞类型。为了填补这一空白,作者引入了MarsGT,它使用基于概率的异构图transformer在单细胞多组学数据上识别稀有细胞群体。在550个模拟数据集和4个真实人类数据集中,MarsGT在识别稀有细胞方面优于现有工具。在人类淋巴结数据中,MarsGT检测到一种可能作为淋巴瘤前体的中间体B细胞群。MarsGT为疾病的早期发现和治疗干预提供了潜在的策略。
Part1背景
多细胞生物包括各种各样的特化细胞。识别这些细胞类型在免疫治疗和临床场景中至关重要,因为它阐明了免疫机制,有助于设计靶向治疗,并通过揭示每个患者独特的细胞组成来支持个性化医疗。然而,当遇到罕见细胞时,困难就出现了。尽管它们很稀少,但稀有细胞群在各种生物过程中发挥着至关重要的作用。例如,抗原特异性记忆T细胞对于持续的免疫监视和长期免疫是不可或缺的,即使在无感染时期也是如此。相反,由于其强大的免疫调节功能,不变的自然杀伤T细胞影响多种病理,包括微生物感染和自身免疫性疾病。此外,微小残留疾病,表示治疗后微小的癌细胞群,作为潜在肿瘤复发的重要早期指标,突出了在疾病动力学和治疗干预中识别和理解这些罕见细胞群的必要性。对这些罕见细胞群的精细把握,最终得到更详细的描述,将阐明我们对肿瘤微环境和引导免疫治疗反应的复杂机制的理解。
Part2研究方法
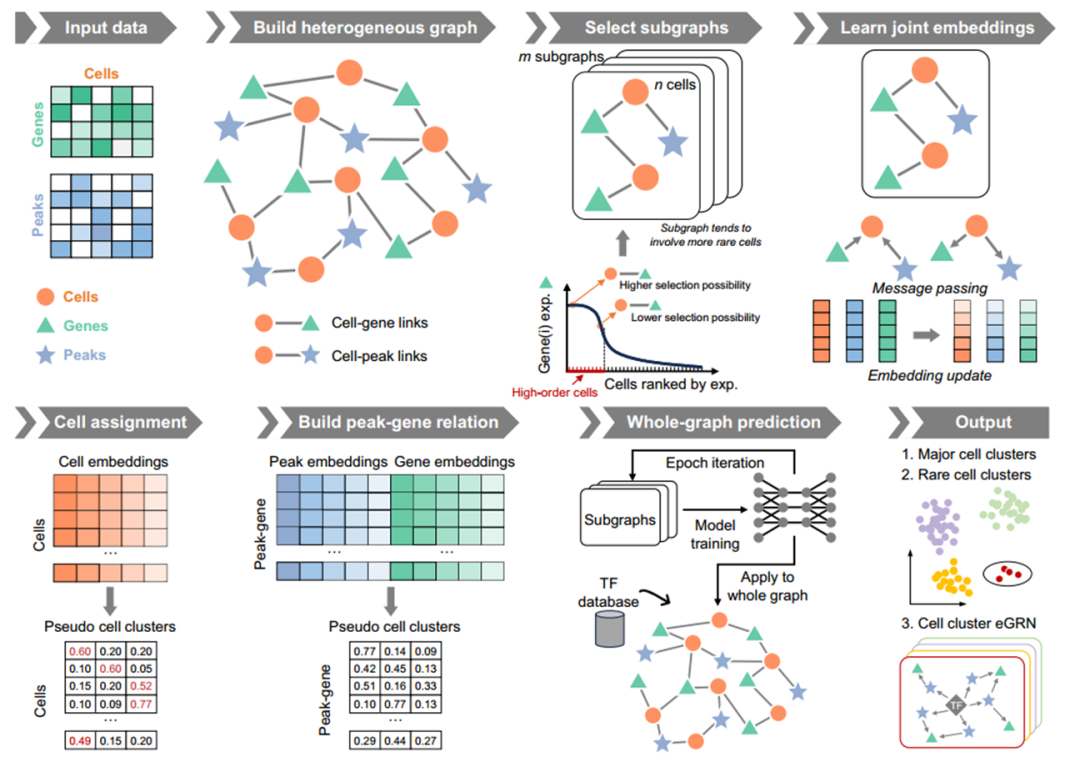
MarsGT模型图
MarsGT结合scRNA-seq和scATAC-seq数据,得到主要的和稀有的细胞群(细胞聚类)及其各自的基因调控关系。模型图主要有六步:首先构建由细胞、基因和峰组成的异构图。第二步是学习嵌入,它通过异构图transformer传递消息。第三步涉及细胞簇预测。从概率矩阵中推断细胞簇。第四步是通过由基因和峰嵌入计算的矩阵来构建峰–基因关系。第五步将训练好的模型应用到整个图中。最后整合转录因子数据库信息来推断基因调控网络。
数据处理
本文输入的多组学数据主要包括scRNA-seq和scATAC-seq,其矩阵形式如下所示: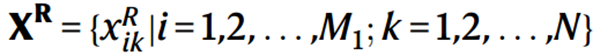 ,
,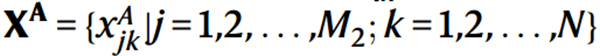 。然后构造评分矩阵:
。然后构造评分矩阵: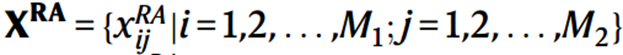 ,在这个矩阵中,
,在这个矩阵中,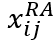 表示峰j相对于基因i的调控潜力。这个潜力是根据峰j和基因i之间的基因组距离确定的。
表示峰j相对于基因i的调控潜力。这个潜力是根据峰j和基因i之间的基因组距离确定的。

异质图构建
定义异构图为G
= (V,E,F),节点集V=
VC∪VE∪VG,表示细胞、峰、基因。边集E构成为:
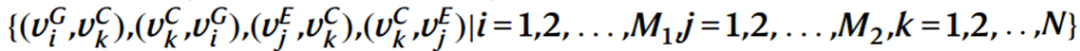
G中节点的初始特征向量F如下:
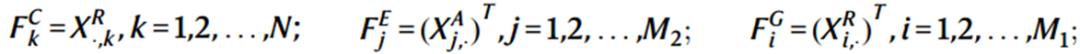
子图采样
为了提高效率,需选择子图。为了识别稀有细胞,作者设计了一种基于概率的子抽样方法【鉴定在靶细胞中高表达但在其他细胞中低表达或不表达的基因或峰是必要的】。第一步过滤掉低表达的基因,这些基因不应该被视为罕见的相关特征。第二步,根据以下公式计算的概率选择基因和峰值:

基因或者峰对应概率越大,更可能显示细胞的罕见信号,越易被选择到对应细胞的子图中。保留基因数用Ng表示,min(Ng,20)。每个子图随机包含30个细胞及其选择的邻居节点。MarsGT使用多个小批进行训练,每个小批由一个子图表示。
MarsGT 嵌入更新
初始嵌入维度统一:
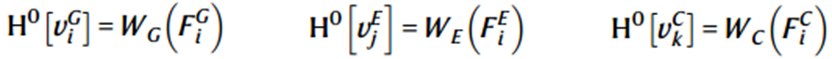
随后使用多头注意力机制:
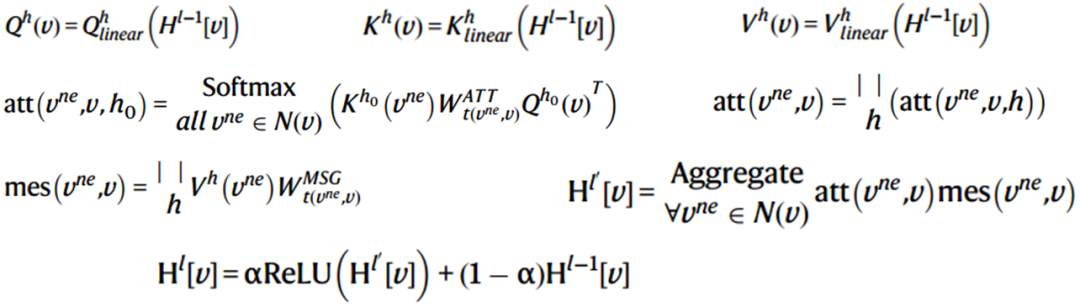
子图训练
更新后的嵌入表示:
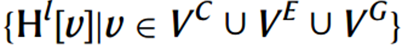
再进行归一化及维度映射,得到细胞矩阵P,行表示细胞,列表示细胞簇(类型);峰-基因矩阵Q,由这两者嵌入拼接而成,映射后,列也表示细胞族(类型),输出是峰-基因属于每个细胞簇的概率,记为O ̂。基峰基因关系根据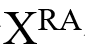 确定,并根据细胞簇中所有细胞对应的基因表达和染色质可及性进行调整,记为O。作者设计了一个多任务损失函数,主要包括四部分。损失(1)和(2)被设计用于获得高质量的节点嵌入。损失(3)和(4)是同时识别稀有和主要细胞群体的关键。基于(3),模型训练过程中引入了峰-基因关系,可以提供更准确的稀有信号。多任务损失函数定义如下:
确定,并根据细胞簇中所有细胞对应的基因表达和染色质可及性进行调整,记为O。作者设计了一个多任务损失函数,主要包括四部分。损失(1)和(2)被设计用于获得高质量的节点嵌入。损失(3)和(4)是同时识别稀有和主要细胞群体的关键。基于(3),模型训练过程中引入了峰-基因关系,可以提供更准确的稀有信号。多任务损失函数定义如下:

MarsGT预测整个图中的细胞簇和eGRN(基因调控网络)
为了确保每个细胞都被映射到相应的预测簇中,并且每个基因和峰都与细胞簇特定的峰-基因调控信息相关联,将训练好的模型应用于整个图。对于细胞簇特异性峰-基因链接,使用最终预测的细胞簇结果来计算所有基因和峰,根据基因表达、峰可及性和峰对基因的调控潜力来确定基因-峰评分。基因-峰评分(PGS)定义为:
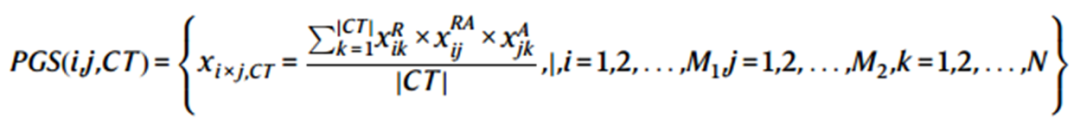
其中|CT|为细胞簇CT中的细胞数。然后推断出细胞簇对应的基因-峰。为了推断eGRN,需要引入转录因子(transcription
Factor,TF)信息。作者从JASPAR检索了基因组浏览器跟踪文件,该文件存储了每个TF的所有已知的TF结合位点。然后,如果一个TF结合位点与预测的峰-基因中的任何峰区域重叠,它将被保留,否则将被删除。最后得到TF峰关系,并推断出每个细胞簇中的基因调控网络(enhancer
gene regulatory networks,eGRN)。
Part3研究结果
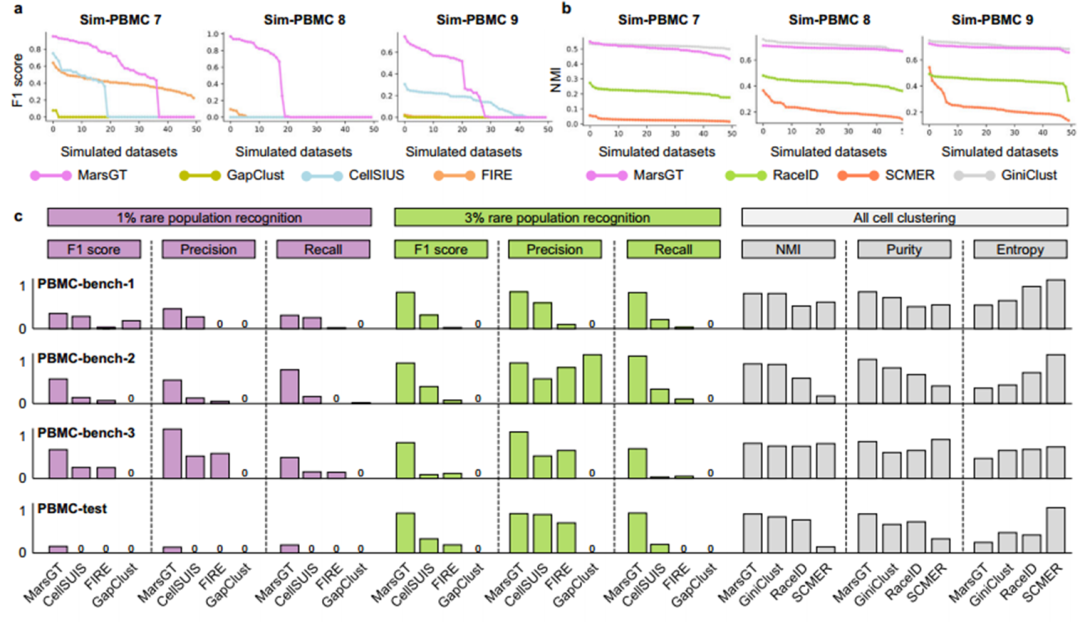
MarsGT在模拟和真实数据集上实现了对稀有的和主要的细胞群的识别

MarsGT在B淋巴瘤数据中识别出处于中间过渡状态的罕见细胞,B淋巴瘤状态-1(BLS1),它有预防淋巴瘤发展的潜力
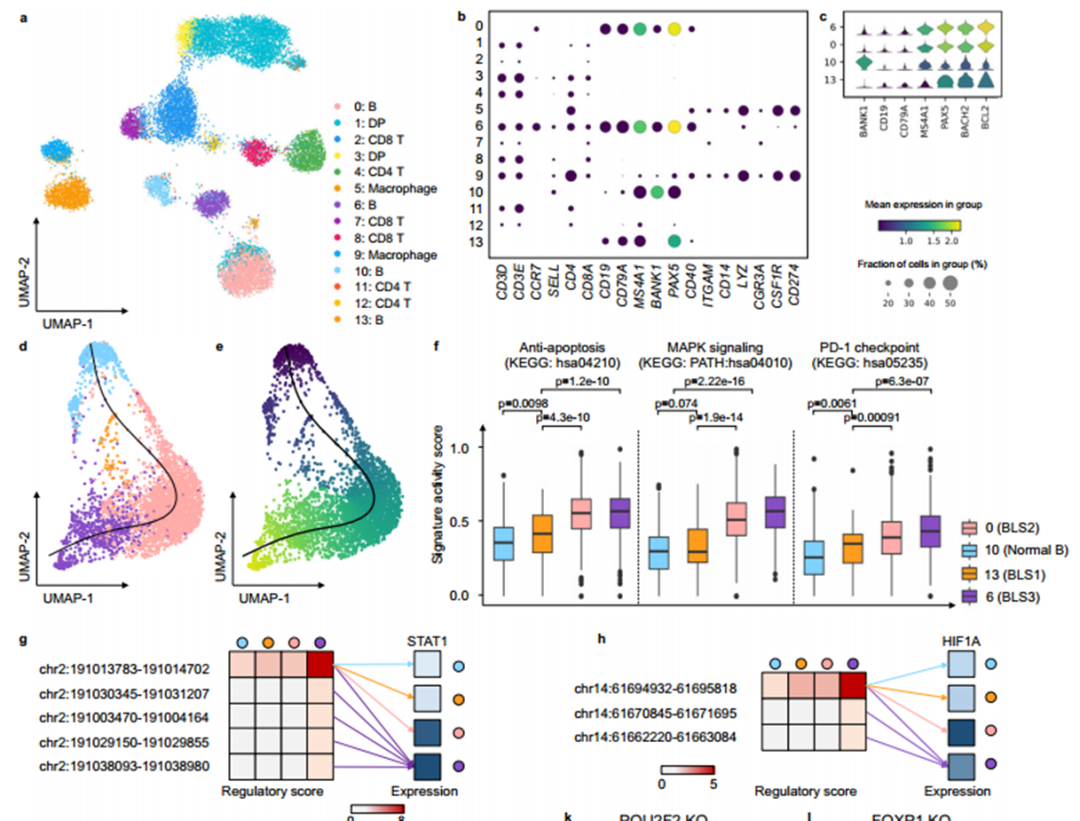
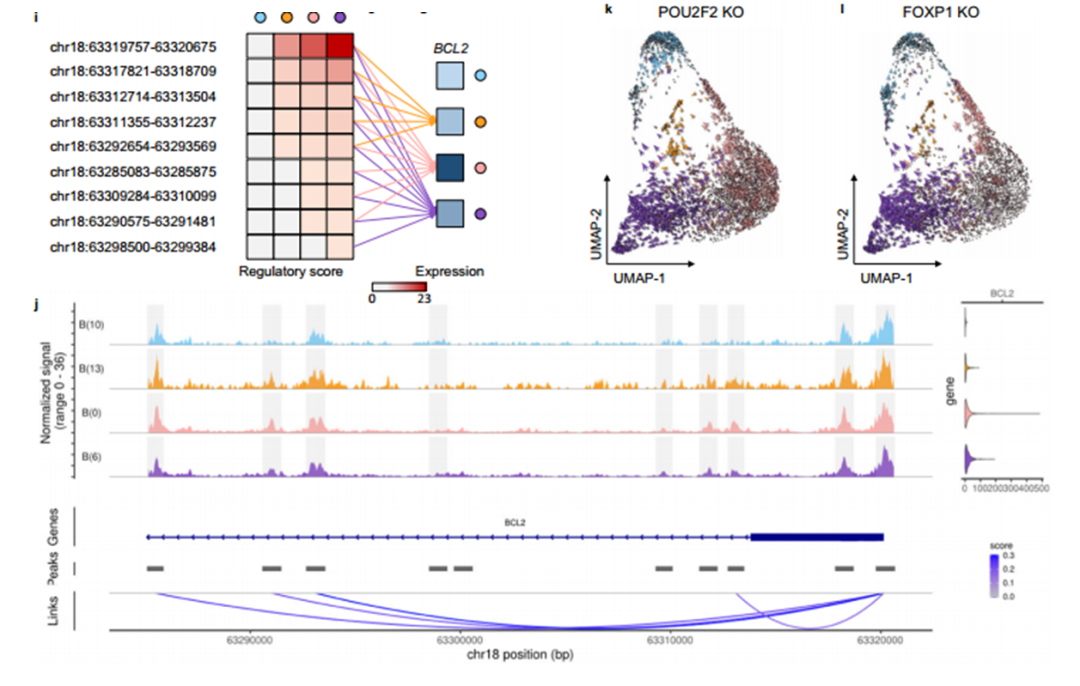
注:更多实验结果及分析请查看原文。
参考资料
原文:https://www.nature.com/articles/s41467-023-44570-8



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


