基于AlphaFold2的静态蛋白结构预测方法已经接近实验精度,为结构生物领域带来了巨大的影响。然而,在药物开发中,靶蛋白的功能性运动非常关键,它会对药物的活性产生影响,而目前的算法模型无法捕捉到大分子的功能构象动态变化过程。近日,上海交通大学郑双佳课题组与星药科技研究团队,美国莱斯大学Peter G. Wolynes联合团队在《Nature Communications》发表题为“DynamicBind: predicting ligand-specific protein-ligand complex structure with a deep equivariant generative model”的研究论文。该论文提出的新方法DynamicBind可基于AF2预测的初始非结合构象,生成与对应输入药物分子相匹配的蛋白结合构象及转化过程,并在计算过程中自动寻找口袋位置,让对接中发现隐藏口袋成为可能。该研究对于蛋白质功能机制的理解以及难成药靶点的药物筛选具有重要意义,为后AlphaFold时代的药物研发提供了一种基于深度学习的,考虑蛋白动态变化的新范式。

背景介绍
继2018年、2020年的两代AlphaFold的横空出世后,蛋白质结构预测已经取得了突破性的进展。尽管蛋白质本质上是动态的,能够采取多种构象以发挥其生物功能,但AlphaFold2等方法通常只能为每种蛋白质序列预测少数几种构象,无法预测配体特异性的功能构象。
目前,蛋白质与配体之间的相互作用主要通过实验或计算方法进行研究,这是基于结构的药物发现的关键步骤。尽管蛋白质动态性的重要性得到了广泛认可,传统的计算方法往往将蛋白质视为刚性体,或者在某些情况下认为其部分灵活,只允许特定的侧链移动,以降低计算成本。因此,当使用AlphaFold预测的无配体蛋白质结构作为对接输入时,所得到的配体位置预测往往与配体结合的共晶结构不吻合。AlphaFold预测的结构常常未能展现出与配体结合最有利的侧链和主链构型,导致相关的活性位点不在正确的位置上,这使得目前很难利用AlphaFold的结构来进行药物筛选和设计。
为了解决这一问题,上海交通大学郑双佳课题组联合星药科技研究团队及美国莱斯大学,结合团队在复合物结构建模和药物筛选领域的前期工作TANKBind(NeurIPS, 2022)和DrugVQA(Nat. Mach. Intel, 2020),提出一种为“动态结合”设计的几何深度生成模型DynamicBind。与传统的将蛋白质视为大多数刚性实体的对接方法不同,DynamicBind使用了先进的深度扩散模型和等变几何神经网络技术,创新性地将定向扩散(morph)和多体玻尔兹曼等变转换器相结合,在对接中同时对分子姿态和蛋白构象的运动进行预测,实现配体特异性的蛋白低能构象的动态跃迁,从而可以找到更适合的蛋白构象和潜在的结合口袋,提高了分子对接功能准确度的上限,同时,该方法可以部分解决传统对接软件无法给出对接结果的困难场景(如别构口袋,隐蔽口袋等问题),更善于找到针对难成药靶点的活性分子。该方法的另一个重要优势在于其采样效率,与传统基于全原子力场的分子动力学方法不同,DynamicBind可以在更短的时间内实现更长级别的蛋白运动预测和轨迹模拟,对毫秒级的构象运动的模拟仅需要一张GPU 2-5分钟的时间完成。
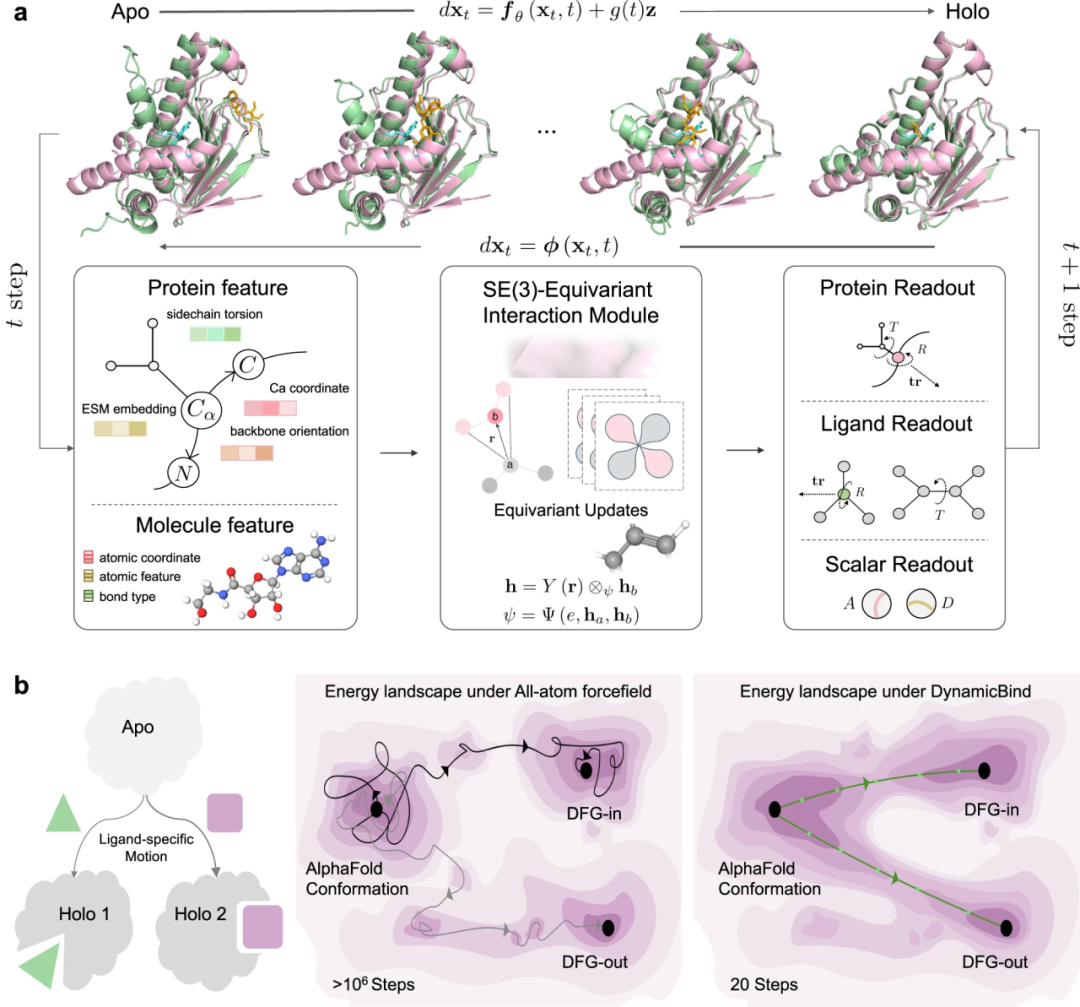
图1. (a)DynamicBind模型示意图,(b) 当蛋白质与两种不同配体结合时,DynamicBind可以预测出两种不同的整体构象,并能在 20 步内预测出蛋白质的结合构象,而要找到相同的结合态则需要数百万步的全原子 MD 模拟。
复合物结构预测性能比较
模型在复合物结构预测性能测试中采取了一种具有挑战性且现实的情境,即假设结合状态的晶体蛋白(holo)的构象不可用且口袋位置不可知,仅使用由AlphaFold预测的非结合蛋白质构象(apo)作为模型的输入,试图预测最终的配体-蛋白复合物结构。实验证明DynamicBind在公开的基准测试集上的预测精度和多种指标下的对接成功率均领先传统基于力场的局部分子对接软件方法(给定口袋)和基于深度学习的对接方法(图2)
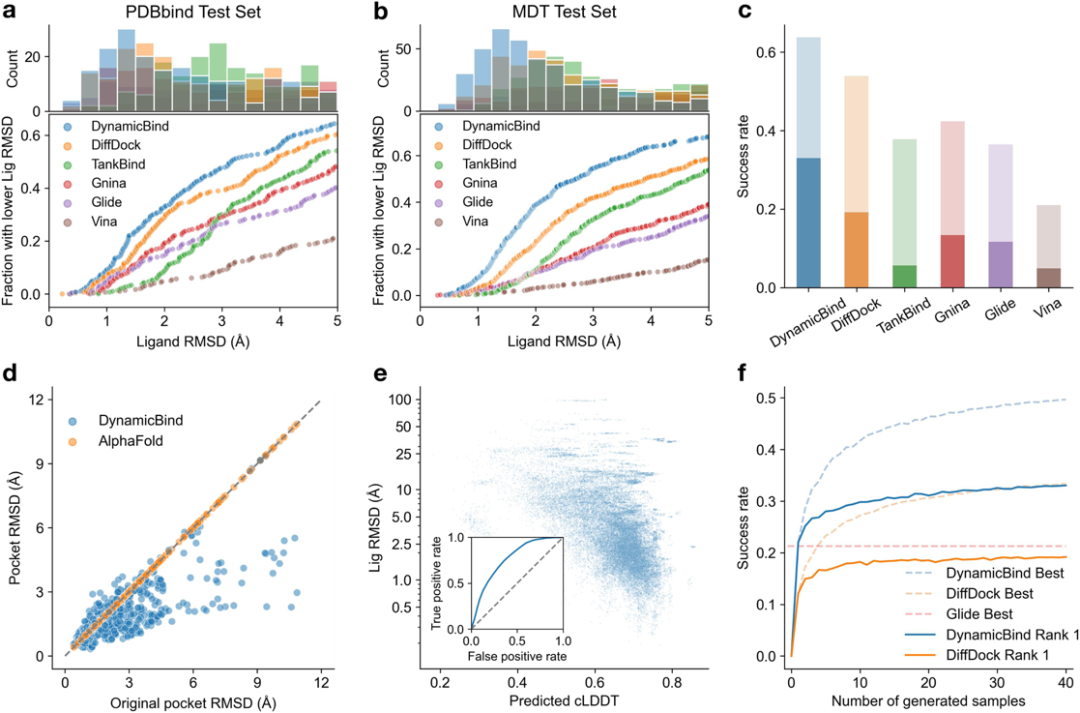
图2. DynamicBind与多种对接方法和深度学习方法的全局双盲对接实验比较结果。
DynamicBind可捕捉配体特异性的构象变化
传统的对接方法通常将蛋白质构象采样作为与对接过程分开的步骤进行。然而,在许多情况下,两种不同的配体可能适配于一个蛋白的两个完全不同的功能构象。在以往的对接模型中,蛋白质必须预设为正确的构象,才有可能识别出配体的适当结合姿态。相比之下,DynamicBind利用由AlphaFold预测的蛋白质构象,能够动态调整蛋白质构象,以找到最适合感兴趣配体的最优构象(图3)。
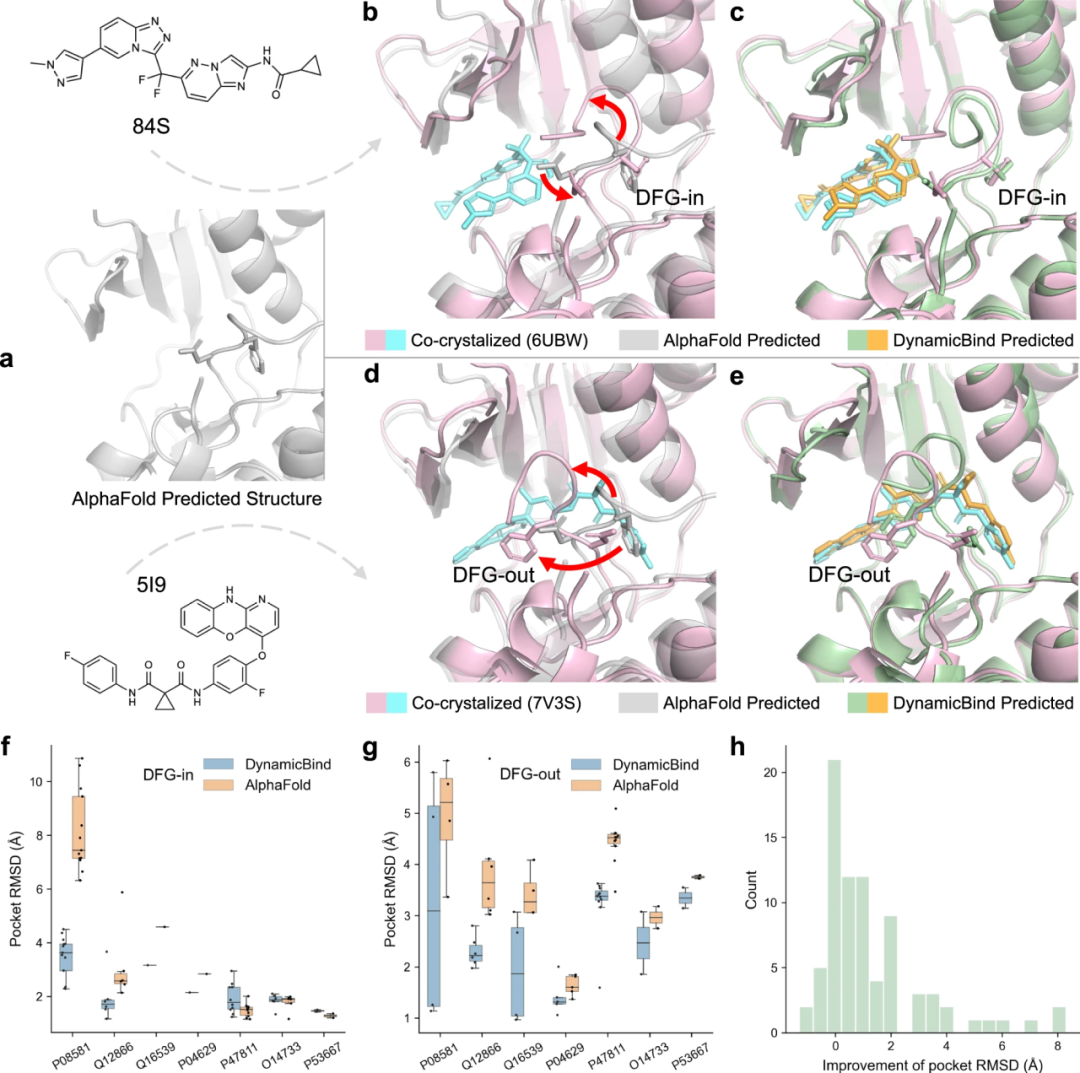
图3: DynamicBind利用由AlphaFold预测的蛋白质构象,能够动态调整蛋白质构象。c-Met激酶可以采取两种不同的构象,分别对应于活性和非活性状态,通常被DFG-in和DFG-out构象。DynamicBind可以根据不同的配体生成匹配的复合物构象。
DynamicBind可捕捉跨尺度的构象变化
研究者发现该模型可以捕捉皮秒级到毫秒级等六种不同类型的跨尺度的构象变化,这使得该方法在大的构象变化捕捉上相比起基于力场的分子动力学方法具有明显优势。图4中展示了DynamicBind预测测试集中的6种不同构象变化的结果。
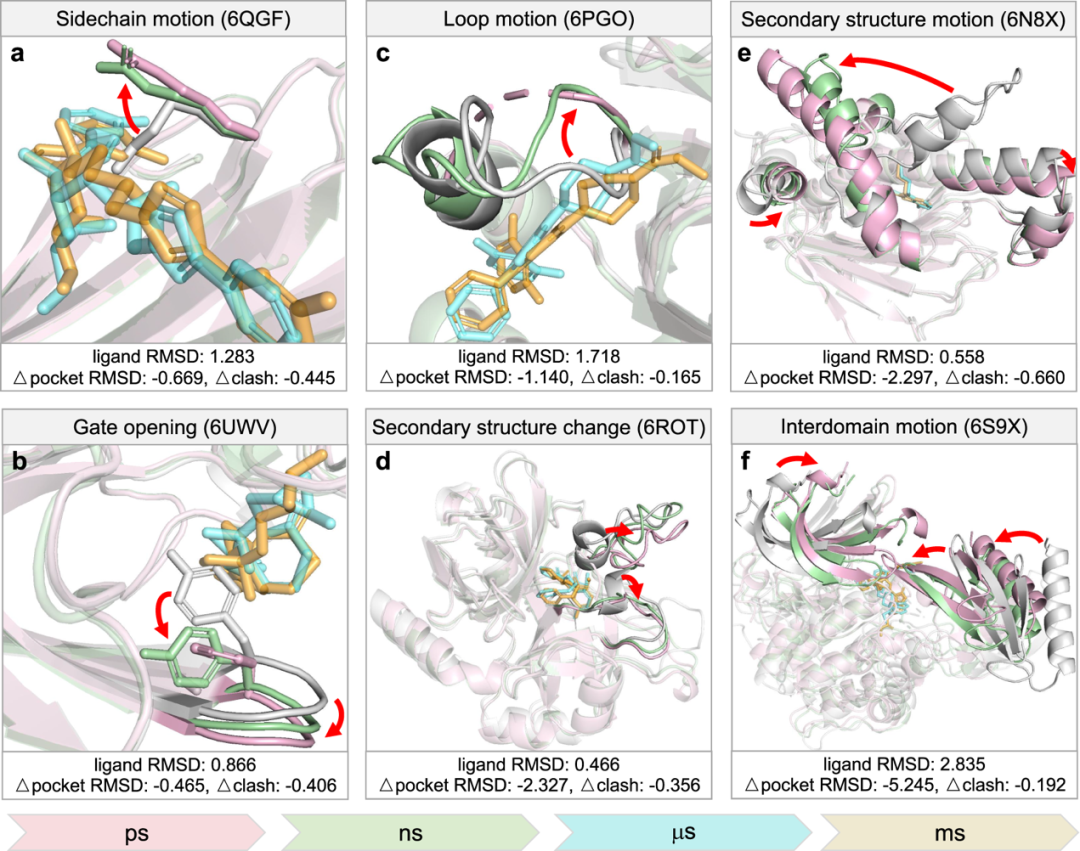
图4:DynamicBind可以捕捉蛋白质在与小分子配体结合后的一系列跨尺度变化。
DynamicBind可识别隐蔽结合位点
传统对接方法通常采用刚性对接模式。对于没有复合物晶体结构的靶蛋白,用于对接的AlphaFold预测的蛋白构象通常和复合物中的真实蛋白构象不同,可能存在未展开的隐藏口袋,从而导致传统对接软件无法顺利地放置目标配体,准确度较低。而Dynamicbind具有识别并模拟“隐蔽位点”(Cryptic Pockets)的能力,可以将配体正确地放入本不存在的全新隐藏口袋中。图5中研究者发现DynamicBind针对SETD2靶点在没有任何提示的情况下可以正确的将配体分子放入本不存在的隐蔽口袋。这是多数传统算法忽视的关键因素,可以有助于药物分子作用机理的研究,对于开发新型药物靶点具有重大意义。
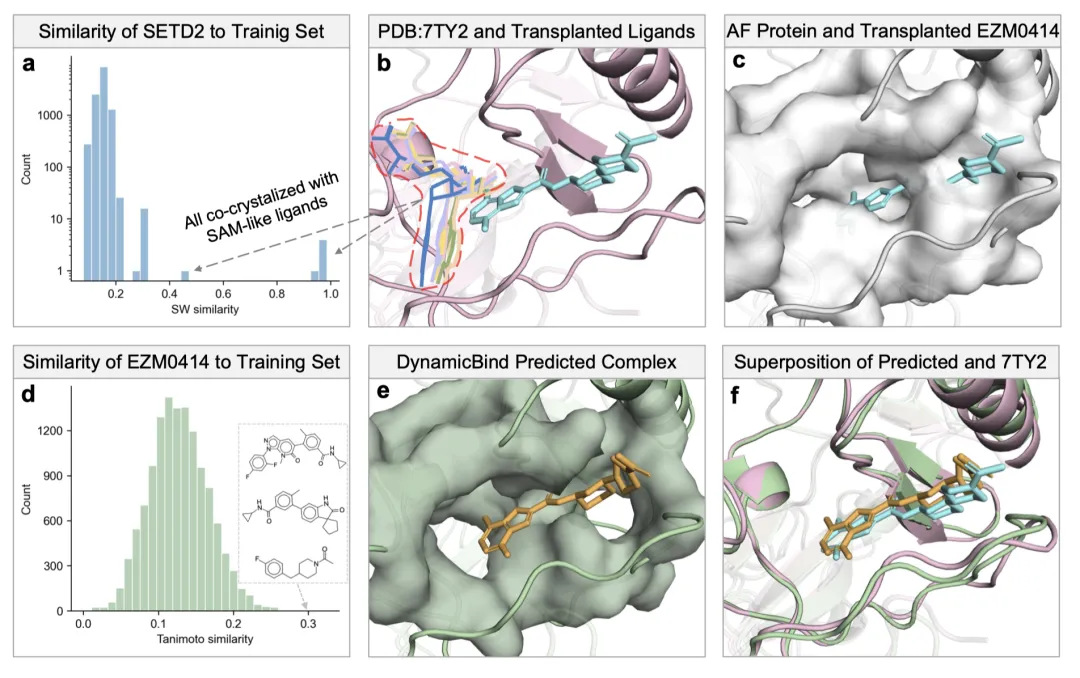
图5:Dynamicbind能够找到AF2的构象中无法发现的隐蔽位点
总结
DynamicBind将传统上分开的两个步骤——蛋白质构象生成和配体姿态对接——统一到一个框架中。作为一个端到端的深度学习方法,它在蛋白质构象采样方面比传统的分子动力学模拟快数个数量级。与需要预定义结合口袋的传统对接方法不同,DynamicBind能够进行全局对接,这对于尚未确认结合口袋的靶蛋白药物发现是一个关键特性。这些优势赋予了DynamicBind在虚拟筛选中发现能够和隐蔽口袋特异性结合分子的能力。DynamicBind不仅为药物虚拟筛选提供一种高效的新方法,而且为后AlphaFold时代的药物研发提供了一种基于深度学习的,考虑蛋白动态变化的新型应用范式。
代码
https://github.com/luwei0917/DynamicBind
撰稿人:纳贤说药
参考资料
-
K., Bates, R., _Zı´dek, A., Potapenko, A., et al. (2021). Highly accurate protein structure prediction with AlphaFold[J]. Nature, 596, 583–589.
-
Fraser J S, Murcko M A. Structure is beauty, but not always truth[J]. Cell, 2024, 187(3): 517-520.
-
Lu W, Wu Q, Zhang J, et al. Tankbind: Trigonometry-aware neural networks for drug-protein binding structure prediction[C]. NeurIPS, 2022, 35: 7236-7249.
-
Zheng S, Li Y, Chen S, et al. Predicting drug–protein interaction using quasi-visual question answering system[J]. Nature Machine Intelligence, 2020, 2(2): 134-140.
-
Lu, W., Zhang, J., Huang, W. et al. DynamicBind: predicting ligand-specific protein-ligand complex structure with a deep equivariant generative model. Nat Commun 15, 1071 (2024). https://doi.org/10.1038/s41467-024-45461-2




 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง





