今天为大家介绍的是来由 Gianni De Fabritiis团队的开发的一个基于网络的数据可视化工具包PlayMolecule Viewer,该工具旨在简化结构生物信息学或计算机辅助药物设计中的数据探索过程。通过利用最新的如 WebAssembly等网络技术,PlayMolecule Viewer 在浏览器环境中直接整合了强大的 Python 库,增强了其处理多种分子数据的能力。PlayMolecule Viewer拥有直观的界面,使用户能够轻松地上传、可视化、选择和操作分子结构及相关数据。该工具包支持多种常见的结构文件格式,并提供多种分子表示方法,以满足不同的可视化需求。
分子可视化对于计算化学家和生物学家至关重要,它使他们能够探索和理解复杂的蛋白质结构,并为药物发现揭示重要见解。通过三维模型的视觉检查,可以更好地理解蛋白质结构与功能之间的关系。此外,它还允许探索蛋白质的活性位点和功能热点,这些都是药物设计中的潜在靶点。传统上的分子可视化工具主要是桌面应用程序,但这些工具的可访问性和适用性常常受到限制。相比之下,基于网络的工具作为分析大分子结构的有价值替代品出现。网络版分子可视化工具具有诸多优势,例如平台独立性和无需安装软件等。这些工具为科学家和非科学社群提供了一个用户友好且易于访问的平台,使他们能够与分子可视化交互,并拓宽蛋白质研究的范围。WebAssembl是一种允许在任何现代浏览器中运行多种语言编写的代码的网络技术,包括在浏览器内直接运行诸如Python这样的解释型语言编写的脚本的能力。WebAssembly为分子科学领域的网络应用发展提供了巨大潜力,已有多个工具在开发中使用了这项技术。
在这种背景下,作者开发了PlayMolecule Viewer,一个旨在促进结构生物信息学或计算机辅助药物设计成果的可视化和理解的网络数据可视化工具包。该工具包使用户能夠轻松地上传、可视化、选择和操作分子结构及其相关数据。通过结合WebAssembly在浏览器环境中直接利用Python库的优势,增强了其处理从静态和动态结构到数据表和图表等多种数据类型的能力。这种多功能性使用户能够将其数据整合到 PlayMolecule Viewer 中,从其分子结构中获得有价值的见解。
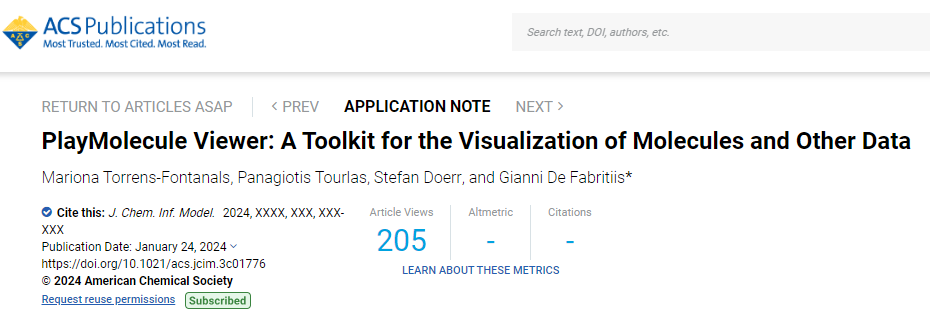
PlayMolecule Viewer对分子结构的可视化提供全面的支持,用于加载包括蛋白质结构、小分子、分子动力学轨迹等数据。这种灵活性使研究人员能够在应用程序内分析广泛的结构数据。如图1,加载的结构显示在“3D 视图控制”面板中,该面板允许用户高效地将上传的结构组织成组,专注于特定的感兴趣结构,并控制其表示方式。PlayMolecule Viewer 提供了多种分子结构表示方式,如球棒模型、空间填充模型或卡通模型,这些可以组合以创建复杂且信息丰富的分子视图。此外,还提供了多种配色方案,为结构的可视化提供了进一步的选择。用户还可以选择并控制结构子集的表示,以增强了他们专注和分析特定感兴趣区域的能力(图2)。

图 1
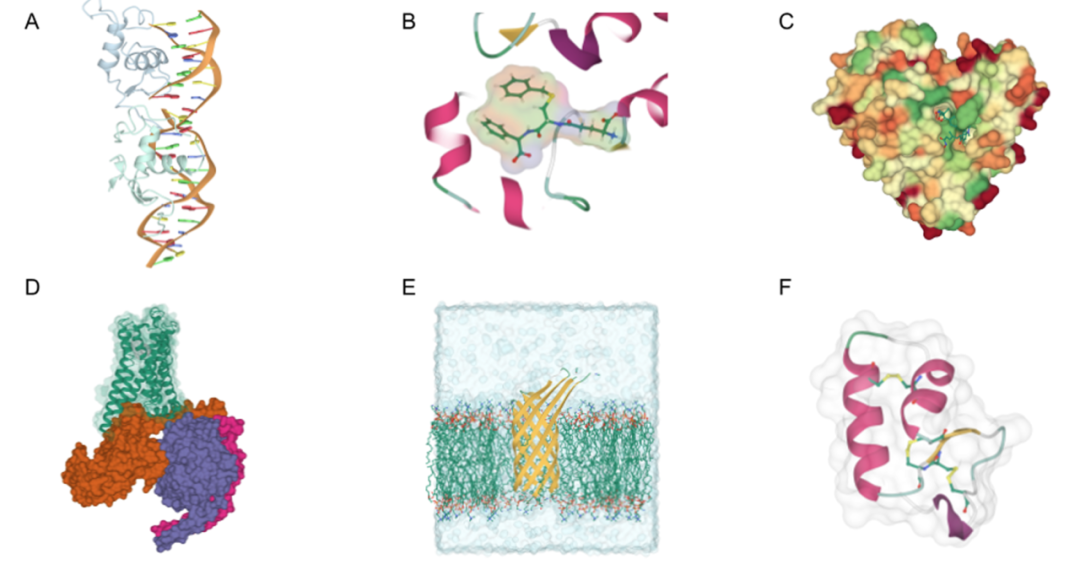
图 2
提供不同的选择是数据可视化的一个基本方面,它使用户能够创建引人入胜、信息丰富且复杂的可视化效果。为此,PlayMolecule Viewer 使用了基于 VMD 选择语言,该语言基于关键词及其关联值(见表1)。用户可以基于布尔关键词(如 protein、water 和 backbone)选择原子。例如,选择“protein”将显示所有蛋白质原子。选择也可以表述为关键词后跟一个列表或值范围。一些例子包括“name CA”(所有名为 CA 的原子)、“resname ALA PHE ASP”(丙氨酸、苯丙氨酸或天冬氨酸中的所有原子)或“resid 50 to 100”(残基索引在50到100之间的原子)。选择还可以与布尔运算符 and 和 or 结合使用,并通过 not 进行修改。例如,“protein and chain A and not name H”对应于链 A 中非氢的所有蛋白质原子。最后,高级标准,如正则表达式(例如,“name ‘C.*’”选择所有以 C 开头的原子名称)和空间选择(例如,“same residue as within 5 of resname BEN”选择与名为 BEN 的残基相距 5 Å 内的残基)也被接受,无疑为选择过程增加了额外的灵活性。
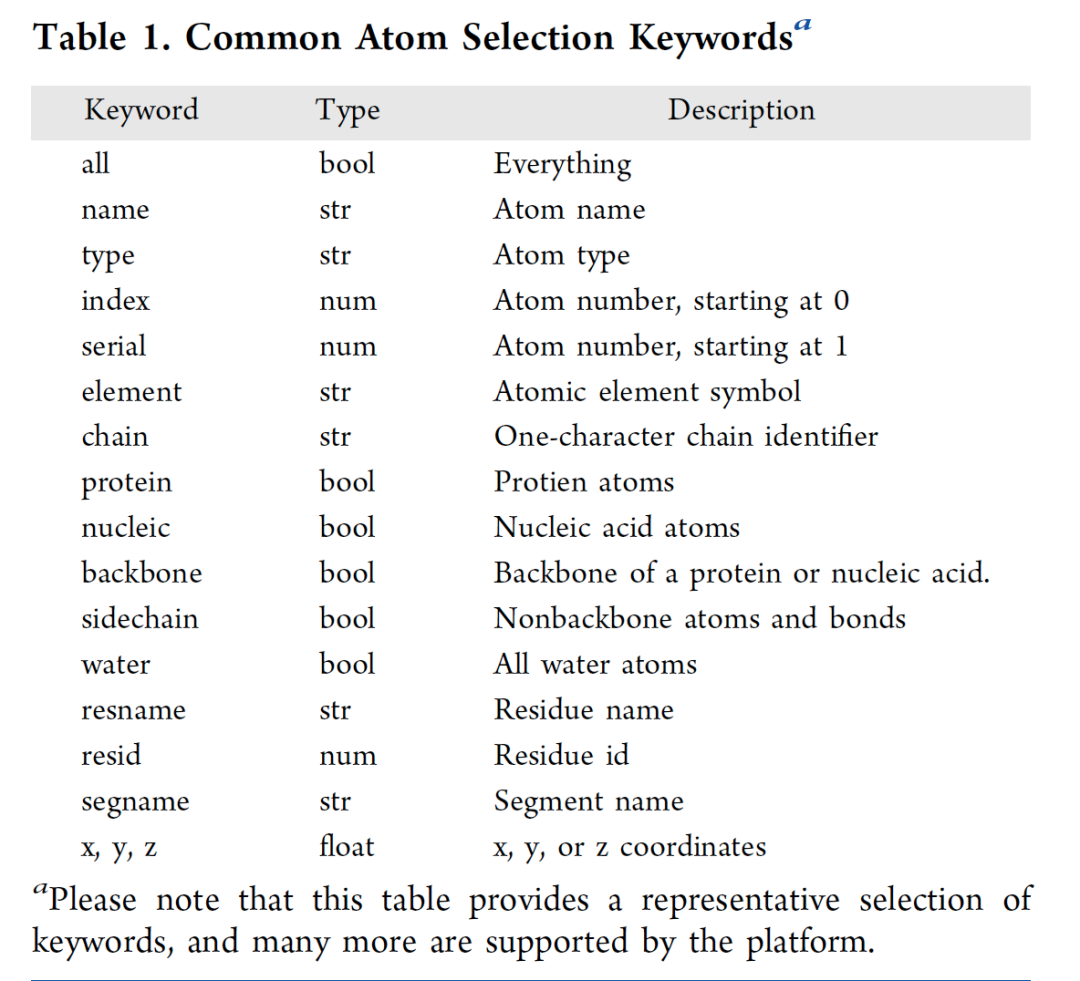
除了分子结构之外,PlayMolecule Viewer 还允许用户以 CSV 格式上传表格(见图3)。这一功能在处理小分子库等方面特别有用。值得注意的是,如果表格包含一个标有“SMILES”的列,应用程序会自动将 SMILES 符号转换为小分子的视觉描述。此外,生成的表格提供了交互式功能。例如,用户可以通过添加一个名为“Path”的列来丰富表格,其中包含已加载分子结构的文件名。当点击表格的一行时,查看器会自动聚焦到相应的结构上,方便用户在表格数据及其相关分子表示之间导航。这种交互性增强了用户体验,并简化了对复杂分子数据集的理解和探索。PlayMolecule Viewer 还提供了静态和交互式图表的可视化,允许用户将复杂的数据表示与他们的分子结构整合(图4)。这些数据可视化功能有助于显示分子系统的时间依赖性属性以及分子动力学轨迹,提供动态行为的全面视图。此外,将能量景观或结合亲和力剖面与蛋白质结构一起显示,有助于理解蛋白质构象。PlayMolecule Viewer还提供了交互式分子分析的 Python 终端,用户可以在终端上执行 Python 代码,获取分子信息、应用过滤器、执行计算等。为了便于工作的连续性,PlayMolecule Viewer 允许用户通过点击 “Save Viewer State”来保存会话状态。该功能可将当前配置保存在会话文件中,包括所选择的表征、选择和对结构所做的任何修改。用户稍后可以重新加载会话文件,恢复之前的可视化设置,确保分析工作顺利进行。此外,目前PlayMolecule Viewer能够加载和可视化多种文件格式。
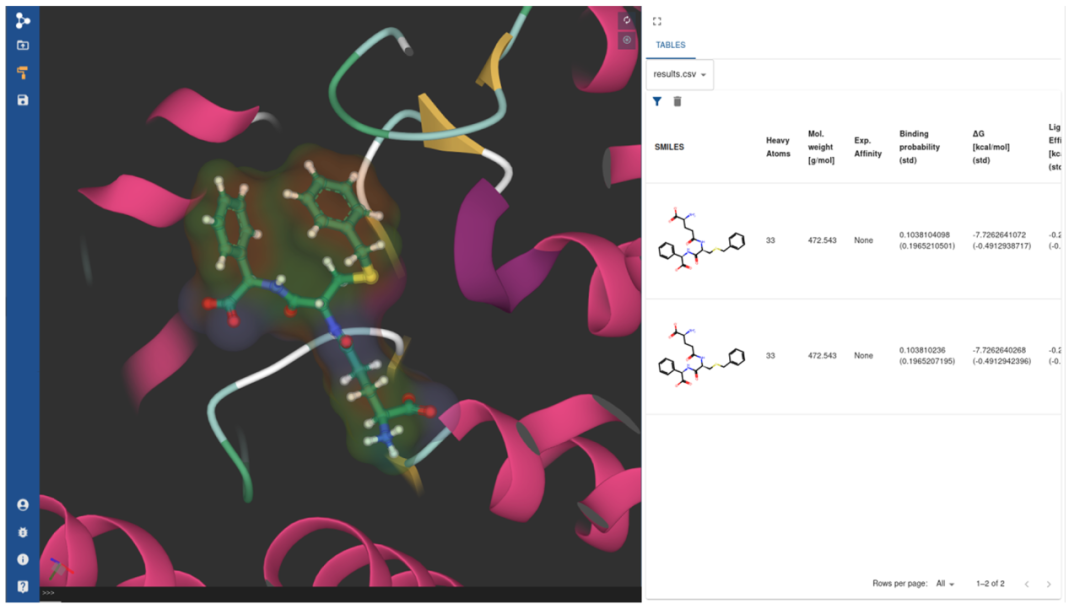
图3
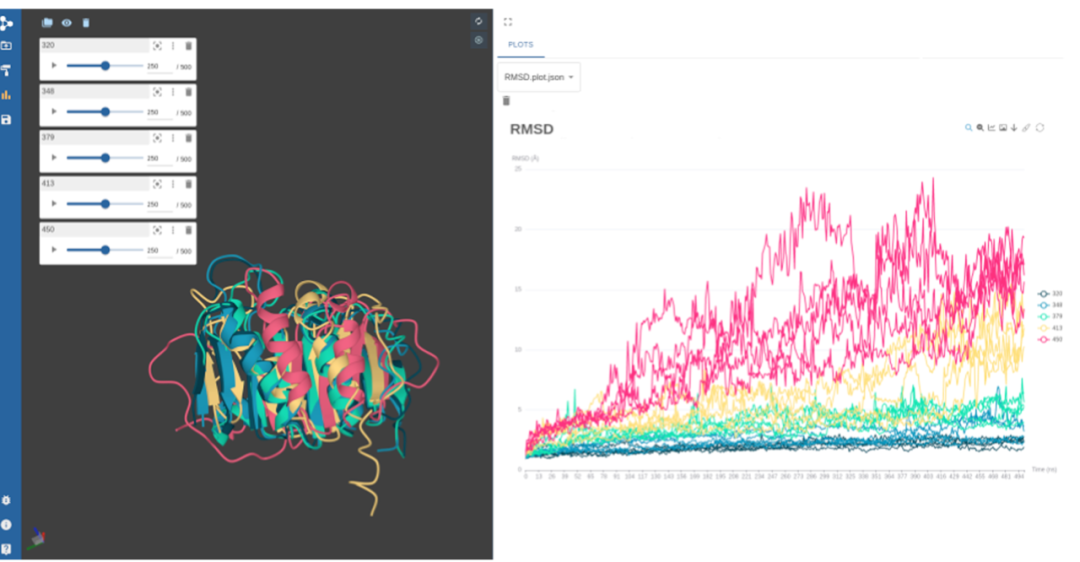
图 4
结论
PlayMolecule Viewer作为一个功能强大且多功能的网络应用程序,用于交互式分子数据可视化。它拥有用户友好的界面,允许用户探索广泛的结构文件格式,包括分子动力学模拟以及数据表和图表。凭借丰富的分子表示类型和色彩方案,以及选择和叠加功能,用户可以创建复杂的分子视图。保存可视化状态的能力确保了之前工作的容易恢复。值得注意的是,该工具包整合了Python终端,使用户能够使用Moleculekit Python库与加载的分子进行交互。PlayMolecule Viewer可在open.playmolecule.org免费使用。
编译 | 曾全晨
审稿 | 王建民
参考资料
Torrens-Fontanals, M., Tourlas, P., Doerr, S., & De Fabritiis, G. (2023). PlayMolecule Viewer: a toolkit for the visualization of molecules and other data. Journal of Chemical Information and Modeling.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


