今天为大家介绍的是来自Yutaka Akiyama团队的一篇论文。环状肽具有许多优异的特性,如高结合亲和力、毒性小以及应对复杂蛋白靶点的潜力。然而,它们在药物领域的应用受到了其低膜透过性的限制——这是口服生物利用度和细胞内靶向的一个重要指标。

环状肽在药物研究中的价值受到重视。它们独特的结构特性源于其受限的构象灵活性和局部的二级结构模式,这使得环状肽能够呈现出具有显著效力和选择性的生物活性构象。在过去的一个世纪里,环状肽药物主要来源于天然产品,包括抗菌剂和人体肽激素。近年来,合成和筛选系统的进步带来了环状肽药物发现的突破。新的筛选和环化策略在过去二十年大大推动了环状肽药物的发展。尽管环状肽具有巨大的药理潜力,它们通常表现出较差的膜透过性,这严重限制了它们的生物学应用和口服药物的开发。尽管环状肽穿越细胞膜的机制尚不清楚,但在疏水环境中呈现“闭合”构象的环状肽展示出了增强的透过性。这种“闭合”构象通过分子内氢键隐藏极性基团和亲脂侧链,有助于提高它们的渗透效率。为了改善膜透过性,研究人员出了各种策略,如构象控制、酰胺到酯的替代、酰胺到硫酰胺的替代以及侧链修饰。然而,这些策略并不适用于所有的环状肽。环状肽的特点是复杂的构象动态,即使是单个残基的微小改变也可能导致它们膜透过性的显著变化。在小分子性质的预测以及肽-蛋白质结合的预测中,多尺度分子特征的结合使用可以提高预测准确性。因此作者提出了CycPeptMP:一种环状肽膜透过性预测模型,该模型有效地整合了多层次特征和最先进的深度学习技术。作者在多个尺度级别设计了特征,以同时捕获环状肽的局部序列变化和全局构象变化。另外,为了提高针对复杂环状肽的训练效率,作者在这三个尺度上采用了数据增强方法。
数据
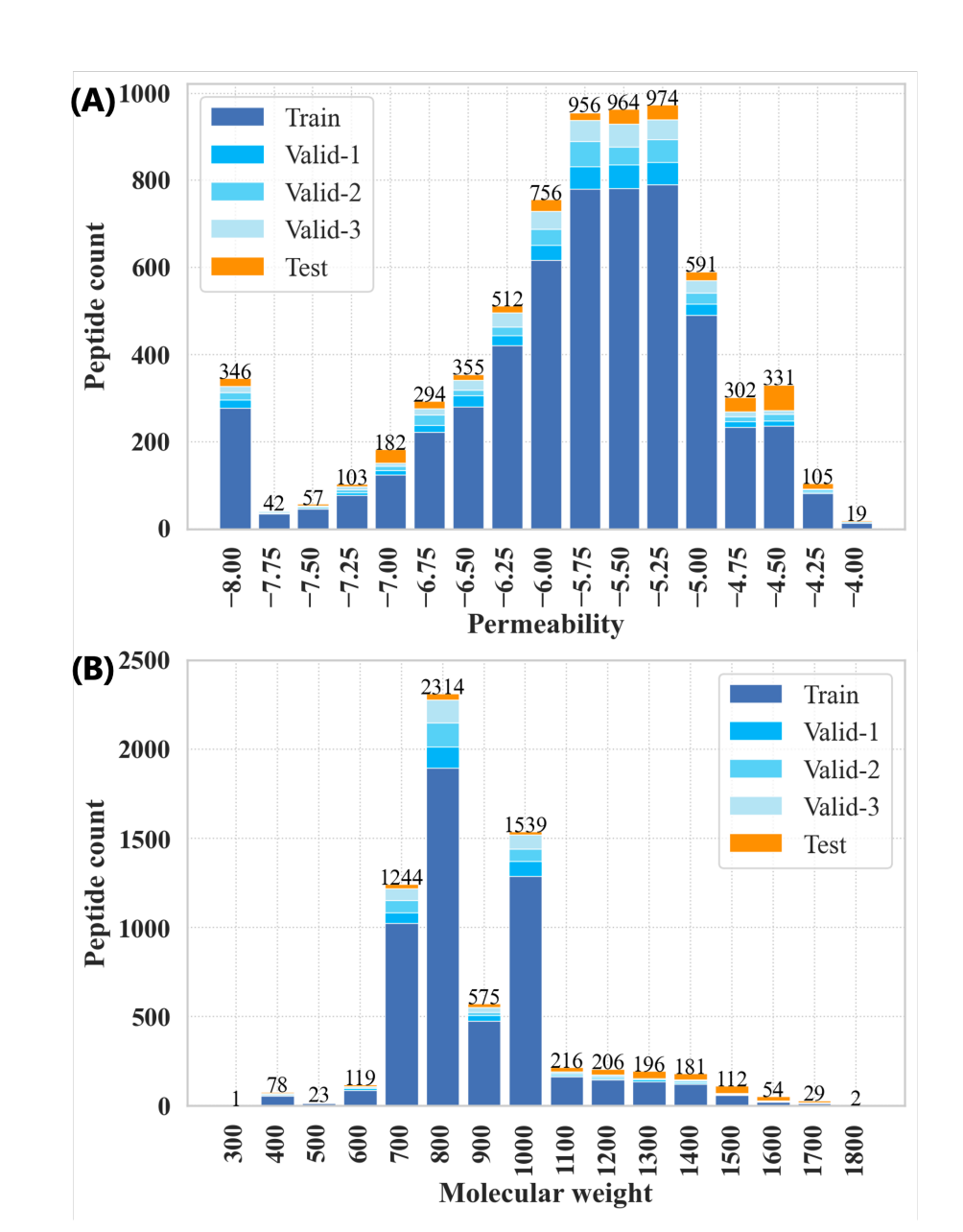
图 1
作者使用了CycPeptMPDB数据集中的肽结构和膜透过性数据,选择了数据点最多的PAMPA测试得到的穿膜结果。因此,选定了6889个肽,覆盖了极其广泛的分子量范围,从342.44到1777.74。验证集和测试集是从总体数据中提取出来用于模型评估。首先,作者使用Kennard-Stone (KS)算法提取了5%的所有数据(344个肽)作为测试集,这些测试集应该均匀覆盖多维空间。作者利用2048位的Morgan指纹(Morgan FP,半径:2)来选择了测试集,使得每个数据点之间的欧几里得距离通过KS算法最大化。从剩余数据中,作者随机提取了5%三次作为验证集(344个肽),数据集之间没有重叠。每组的膜透过性和分子量分布显示在图1中。
模型
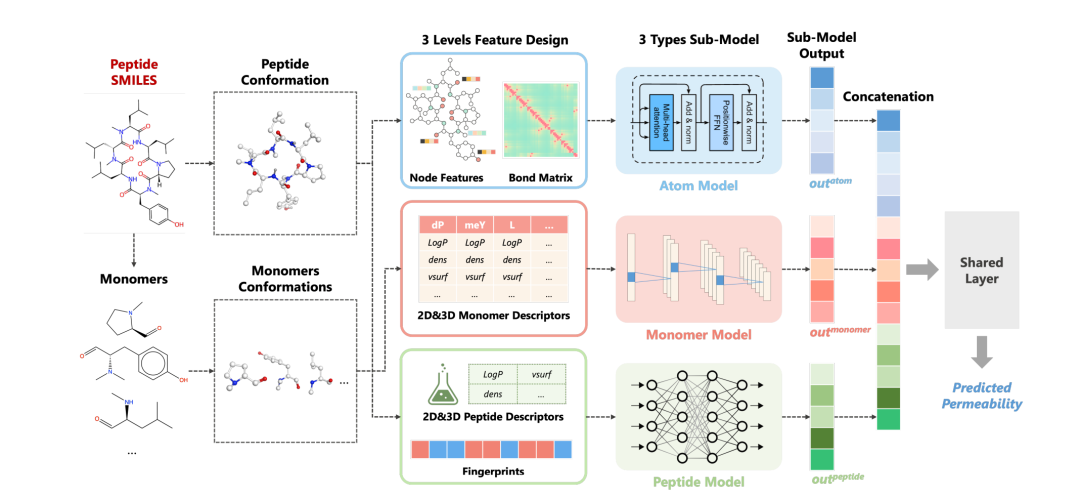
图 2
图2展示了CycPeptMP模型的总体架构。作者设计了三级肽表示法,并分别用于三个不同的子模型,以提取原子级、单体级和肽级的分子表示。首先,输入的肽被分解成单体,并分别为肽和单体生成相应的三维构象。接着,从肽构象中提取原子级和肽级特征,并分别作为原子模型和肽模型的输入。从单体构象中提取的单体级特征被用作单体模型的输入。最后,使用这三个子模型提取的三级潜在特征向量被组合起来,以推导出膜透过性预测值。这里利用单体级特征来准确捕捉环状肽的微妙结构变化,单体的定义与CycPeptMPDB数据集提供的定义相对应。

表 1
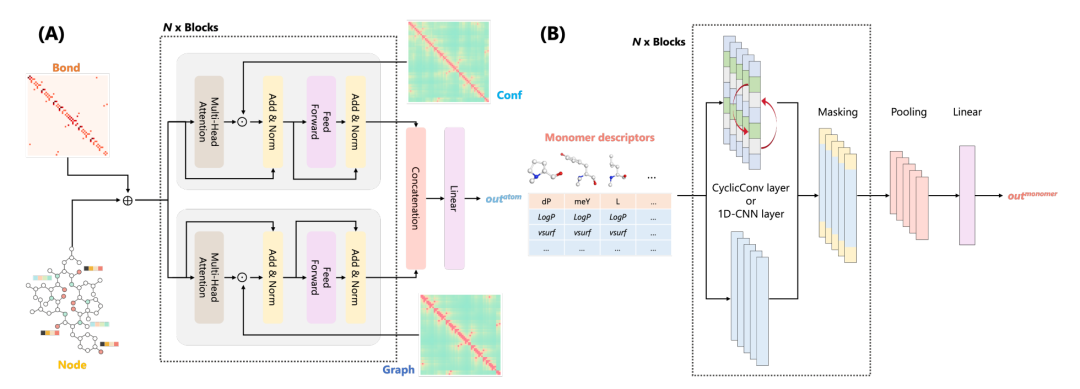
图 3
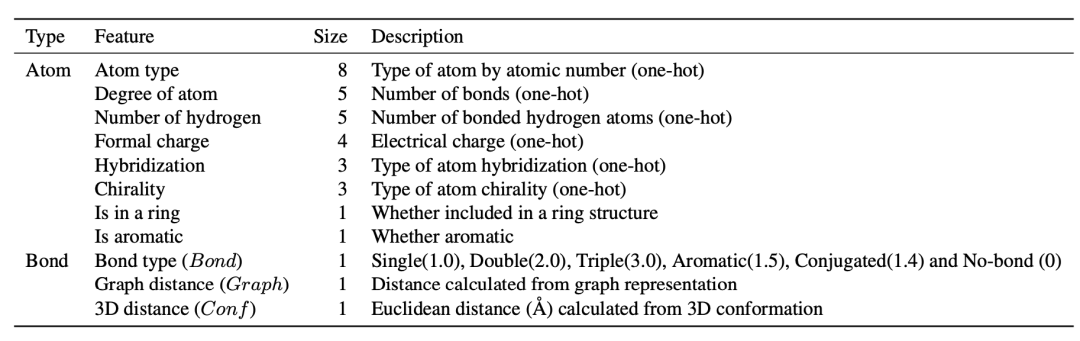
表 2
为了设计肽和单体级特征,肽和单体被表示为16种描述符(表1)。作者依靠随机森林从茫茫多的上千种描述符中提取出这些重要的描述符,具体过程请参考原文附录图S2。此外,作者构建了一个基于transformer的原子模型来捕捉肽的整体图结构和3D构象(图3 A)。作者使用了节点特征(Node)和三种类型的键权重矩阵作为原子模型的输入(Bond, Graph, Conf)。如表2所示,重原子被视为节点,节点特征被表示为Node,键相互作用权重被表示为Bond,图形的成对距离被表示为Graph,而3D的成对距离被表示为Conf。在此设置中,原子模型旨在通过捕捉肽的详细化学结构和空间关系来理解其分子特性,这对于精确预测膜透过性至关重要。通过集成多种类型的数据(如节点特征和不同类型的距离度量),该模型可以更全面地捕捉肽的复杂性质,并有助于提高预测的准确性和可靠性。
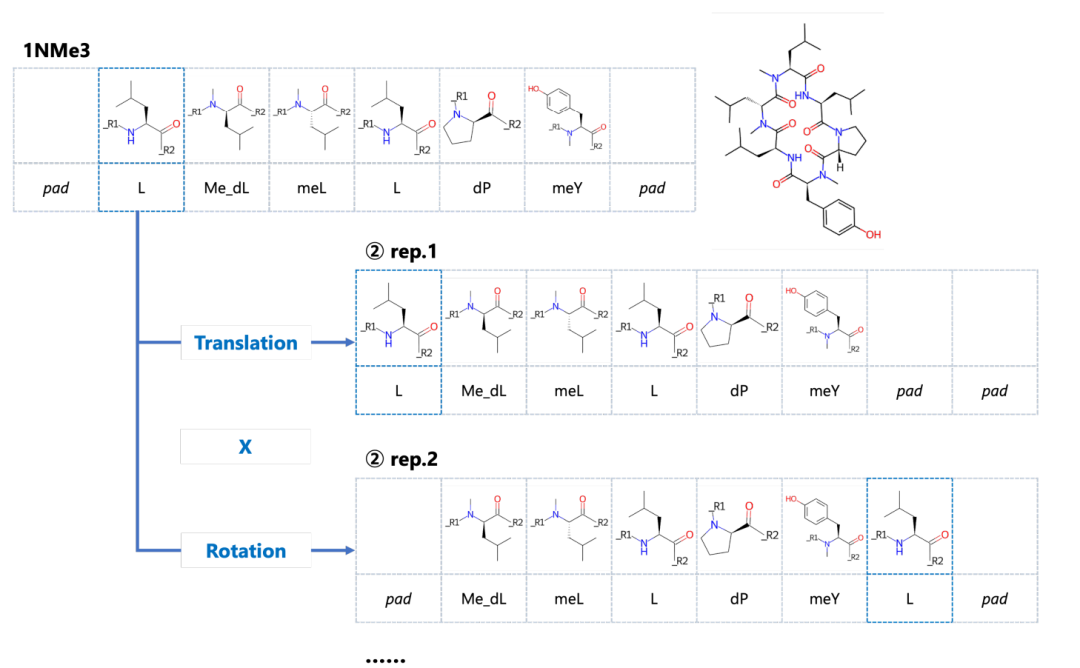
图 4
尽管近年来可用的生物数据量有所增加,但与自然语言处理和计算机视觉领域的数据相比,实验数据仍然有限。例如,涉及小分子毒性分类的Tox21数据集只有大约8000个数据点。这种生物数据的局限性促使人们越来越多地使用自监督学习方法,利用对比学习和预训练来增强模型的学习效率。然而,由于环状肽相较于小分子更有限的可用性,这些技术对环状肽而言仍然具有挑战性。为了提高模型的学习效率,作者使用了三种增强方法,基于SMILES的属性、环状肽序列的性质以及环状肽结构变化的复杂性,生成了60种扩增的肽表示。首先,使用SMILES枚举技术来置换原子顺序,并为原子模型生成不同顺序的输入。接着,考虑到环状肽的环状性,使用序列排列法重新排列了单体模型的输入——如图4所示,对齐的单体描述符被平移和旋转。最后,使用RDKit为每个肽/单体生成了60种构象,以将更多样化的3D信息纳入模型。在训练过程中,每个副本被赋予相同的标签并作为独立数据处理。在推断过程中,60种表示的平均值被用作最终预测值。通过这种方法,模型能够有效地利用有限的数据,通过增强技术生成更多样化和丰富的训练样本,进而提高学习效率和预测性能。
实验部分
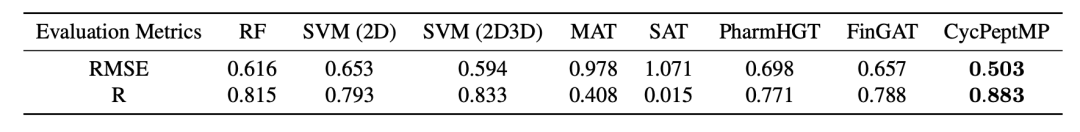
表 3
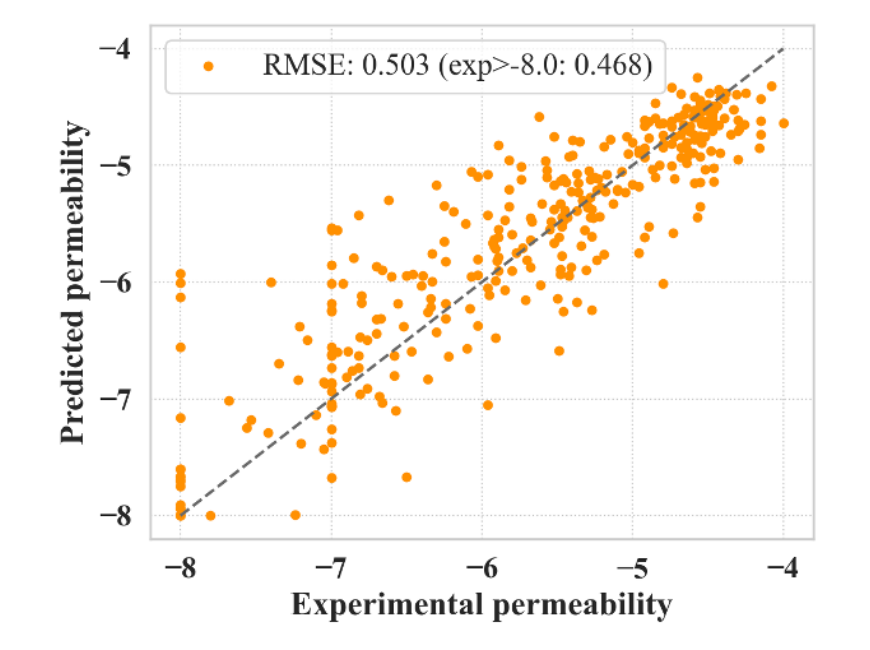
图 5
针对测试集的预测准确性结果显示在表3中。CycPeptMP在所有评估指标中排名第一(RMSE = 0.503,R = 0.883),反映出与所有现有方法相比,预测性能有显著提升。考虑到测试集的结构多样性,CycPeptMP展现出良好的泛化性能,能够学习环状肽的复杂结构。基于Morgan 指纹构建的随机森林模型显示出良好的预测性能,在所有方法中排名第三(RMSE = 0.616,R = 0.815)。与随机森林模型相比,使用2D肽描述符的支持向量机(RMSE = 0.653,R = 0.793)预测精度较低,而使用3D描述符的支持向量机提高了预测准确性,在测试集中表现优于随机森林模型(RMSE = 0.594,R = 0.833)。被动扩散的环状肽膜渗透与分子大小呈负相关。支持向量机可以通过使用诸如LogP之类的亲脂性描述符在某种程度上预测透过性,这些描述符主要依赖于分子量。CycPeptMP有效地结合了Morgan 指纹和16个2D和3D肽描述符作为肽级信息,从拓扑和物理化学的角度全面描述了肽结构,从而提高了预测能力。基于图表示transformer的MAT(RMSE = 0.978,R = 0.408)和SAT(RMSE = 1.071,R = 0.015)无法预测膜透过性。尽管MAT和SAT是预测小分子性质的最先进方法,但它们无法有效学习更复杂的环状肽结构。除了原子级信息外,带有片段MACCS键的PharmHGT(RMSE = 0.698,R = 0.771)和带有分子Morgan 指纹的FinGAT(RMSE = 0.657,R = 0.788)与MAT和SAT相比有显著提高的预测准确性。FinGAT的准确性与随机森林模型和2D 支持向量机相当。这些结果揭示了从不同角度设计特征可能是成功预测环状肽膜透过性的关键。
这些发现表明CycPeptMP有效地利用三级特征来捕捉从最小的原子细节到更广泛的肽级构象的广泛结构信息。此外,实验值为-8的肽无法被CycPeptMP预测(图5)。与所有数据的RMSE(0.503)相比,排除实验值为-8的肽的数据的RMSE显著降低(0.468)。由于不同检测环境的检测限制,作者表示在数据处理将从-8到-10的数据四舍五入到-8,这可能不是唯一合适的方法。
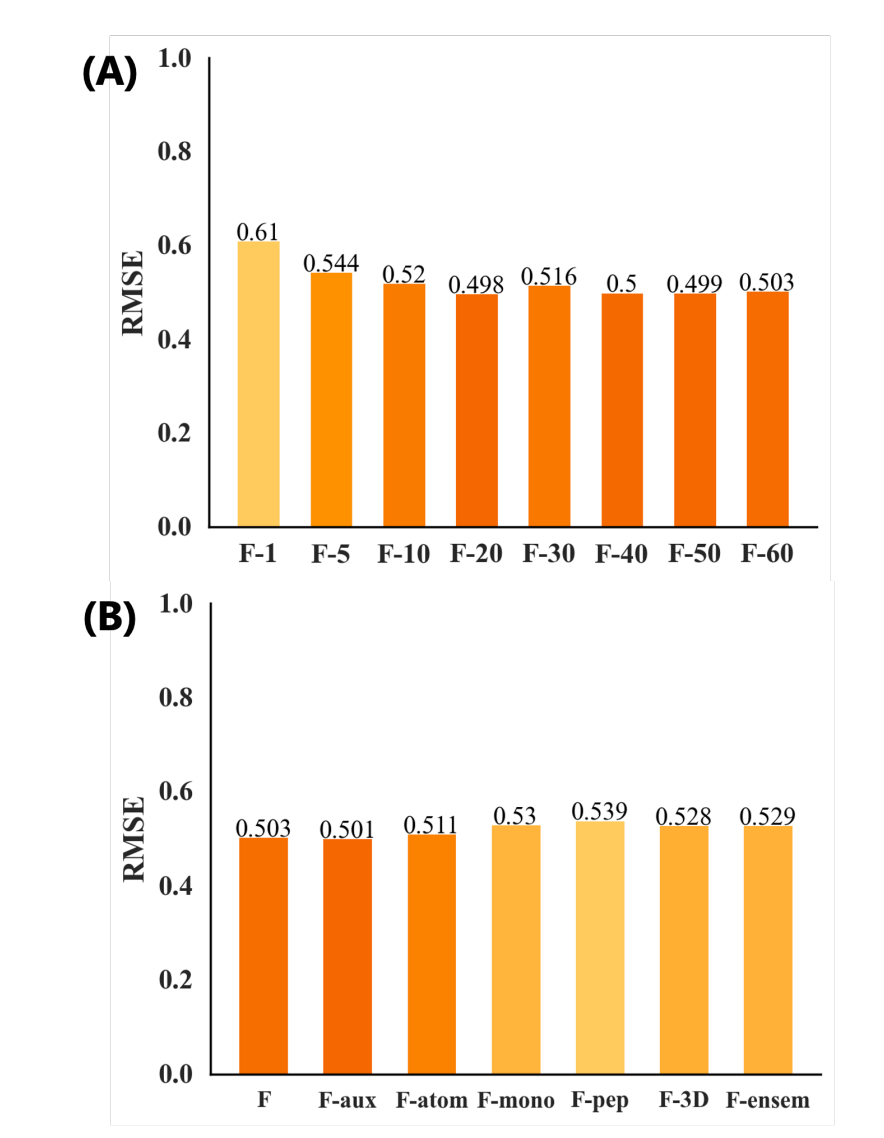
图 6
作者还做了与基于分子动力学方法的比较。Sugita等人使用了一种增强的分子动力学技术,来加速对环状肽膜渗透过程的模拟。尽管如此,他们的方法在预测23个肽的膜透过性时表现不佳(RMSE = 1.899)。CycPeptMP能够对这23个肽透过性进行准确预测(RMSE = 0.127)(图6)。总的来说,与基于MD的方法相比,CycPeptMP能够准确且迅速地预测肽的透过性,具有远远优于分子动力学方法的性能。因此CycPeptMP代表了环状肽药物发现的一个相当有希望的深度学习工具。
编译 | 曾全晨
审稿 | 王建民
参考资料
Li, J., Yanagisawa, K., & Akiyama, Y. (2023). CycPeptMP: Enhancing Membrane Permeability Prediction of Cyclic Peptides with Multi-Level Molecular Features and Data Augmentation. bioRxiv, 2023-12.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


