今天为大家介绍的是来自Pietro Sormanni团队的一篇论文。单克隆抗体已成为关键的治疗药物。特别是纳米抗体,这种小型的、单域的抗体自然表达于骆驼科动物中。自2019年首个纳米抗体药物获批后迅速受到关注。然而,将这些生物制品作为治疗药物开发仍然具有挑战性。尽管目前研究人员已经开发体外相对快速低廉的定向进化技术,但从动物免疫或患者身上发现治疗性抗体仍是黄金标准。源自免疫系统的抗体在体内通常具有诸如长半衰期、低自身抗原反应性和低毒性等有利特性。

抗体是一类具有显著能力的生物分子,它们能选择性地紧密结合到分子靶点。因此,抗体在生物研究和医学领域中有着重要应用,广泛用作诊断和治疗剂。纳米抗体(Nb),即单域抗体(VHH),自然表达于骆驼科动物中的。它们因其独特的结构特性而日益受到欢迎。自2019年首个纳米抗体药物Caplacizumab获批以来,它们作为治疗剂的潜力得到了更广泛的认可。
发现新的针对特定靶点的抗体或纳米抗体的现有方法大致可以分为第一代体内方法(如动物免疫)和第二代体外技术(如实验室库构建和筛选)。近年来,基于计算设计的第三代方法开始出现。自20世纪90年代中期以来,体外方法如噬菌体展示法显示出替代动物免疫或其他体内技术以分离新抗体的潜力。体外选择比体内方法更快速、成本更低,伦理问题更少,并且能更好地控制。然而,尽管成本和复杂性增加,许多制药和生物技术公司更倾向于通过免疫具有人类化免疫系统的转基因动物或直接从患者中分离抗体来获得新抗体。这种选择的原因是,与体外定向进化相比,免疫系统进行的抗体选择通常产生具有更高开发潜力和尤其是更好体内特性的抗体,好处包括长半衰期、低免疫原性、无毒性和低自身抗原交叉反应性。到目前为止,大多数治疗性抗体仍来自动物免疫。因此,这引发了一个问题,即计算设计策略是否能够满足生成具有这些属性的抗体的挑战。在这项工作中,作者引入了一种新的深度学习方法来解决这些挑战,使计算工程能够处理与免疫系统获得的抗体和纳米抗体序列几乎无区别的的抗体和纳米抗体序列。作者将这种方法称为AbNatiV,因为它能够准确地量化给定序列属于源自人类或骆驼科免疫系统的天然可变域(Fv)序列分布的可能性。
模型
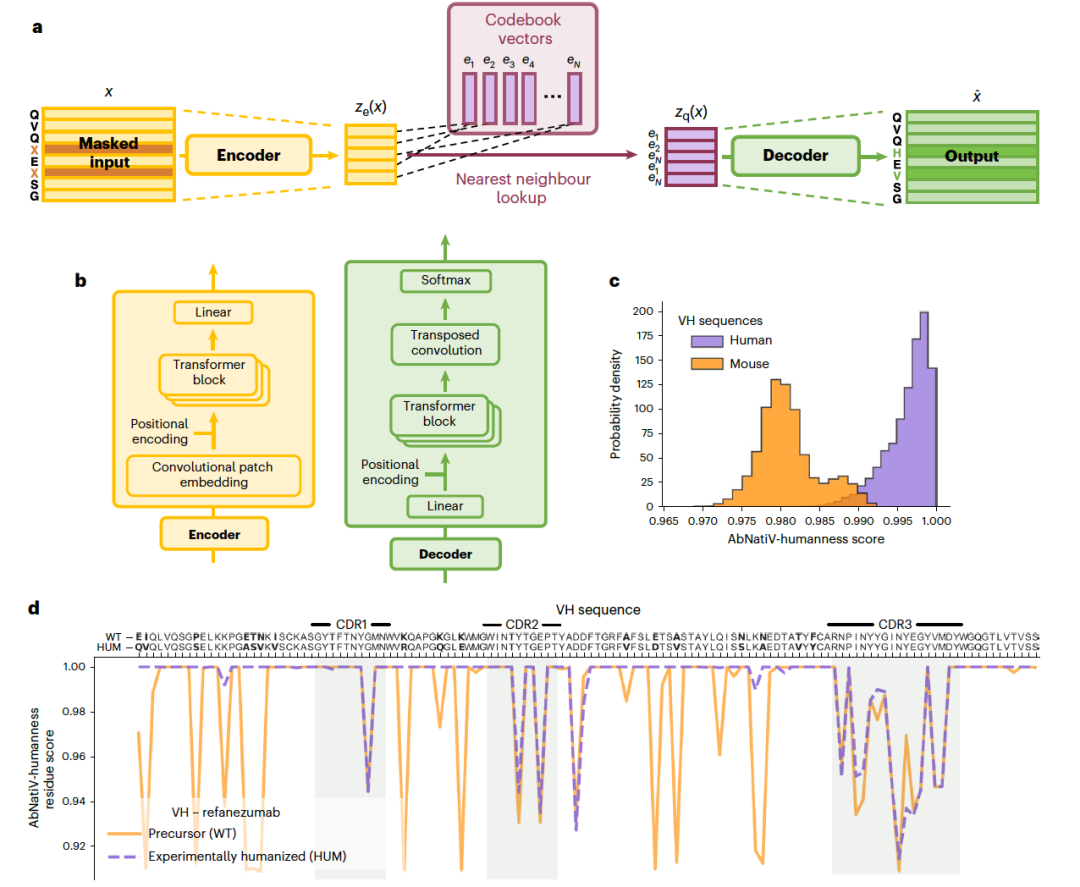
图 1
AbNatiV是一个基于免疫系统来源的抗体序列训练的深度学习模型。它使用的架构灵感来自于VQ-VAE。AbNatiV架构将氨基酸序列压缩进嵌入层,其中每个潜在变量都映射到可学习码本中最近的代码向量,然后通过解码器重构(图1a)。来自码本的向量量化导致了与标准VAE中的连续潜在表示不同的离散潜在表示。选择VQ架构是因为蛋白质序列是离散的对象,可能更适合离散表示,而且它被证明能够规避有时影响标准VAE的后验崩溃问题。模型通过掩码非监督学习进行训练。非监督学习的工作假设是,每个抗体都遵循一些生物物理和进化规则,使其能够被生物体产生并执行其生物功能,而不引起毒性。如果输入中存在某种结构,正如天然抗体序列的情况一样,这种结构应该被学习,并在输入通过网络嵌入层被利用。因此,AbNatiV架构能够学习天然抗体的表示,捕捉残基位置之间的高阶关系,以提供对抗体自然性的高度敏感度量。重构精度用均方误差(m.s.e.)量化,该误差是在one-hot编码的输入序列和重构的输出序列之间计算的。
AbNatiV的架构和掩码非监督学习策略共同促使该模型捕捉到了一系列天然抗体序列数据库中共有的基本特征。AbNatiV在从Observed Antibody Space (OAS)数据库和其他来源策展的免疫库中获取的天然抗体对齐序列上进行训练。该模型分别对人类的VH、Vκ、Vλ和骆驼科VHH序列(每个训练集大约有200万个独特序列)进行了十个训练周期。κ和λ轻链由于它们的显著差异被单独处理。AbNatiV在单个GPU(NVIDIA RTX 8000)上训练每个周期大约需要1小时。每个模型都有一个由5万个独特序列组成的验证数据集,这些序列与训练集中的序列不同,用于监测是否存在过拟合并用于超参数优化。另外,1万个与训练和验证集不同的独特序列被留作测试。对于每个输入的Fv序列,训练有素的AbNatiV模型返回一个抗体自然性分数和序列概况。自然性分数量化了输入序列与学习分布的接近程度,即接近该模型所训练的免疫系统衍生的天然抗体序列。为了方便解释这个分数并比较不同训练模型的分数,AbNatiV分数被定义为接近1的高自然性序列,0.8则是最好地区分天然和非天然序列的阈值。在训练VH、Vλ和Vκ人类链的AbNatiV情况下,这个分数被称为AbNatiV人性化分数(图1cd)。类似地,对于在骆驼科VHH序列上训练的AbNatiV,这个分数被称为AbNatiV-VHH自然性分数。序列概况由输入序列中每个残基位置决定,总共包括149个条目。在这里,接近1的条目表示高自然性,小于1表示逐渐降低的自然性。这个概况有助于理解哪些序列区域或残基对整体自然性贡献最大,哪些可能是潜在问题。例如,图1d显示了一种小鼠抗体(WT前体)的VH序列的人性化概况,并与其人源化对应物治疗性抗体refanezumab相比。refanezumab包含的低分区域要少得多。这个例子表明,序列概况可以作为指导抗体工程的强大工具,通过促进设计突变来提高抗体的自然性。
抗体分类能力
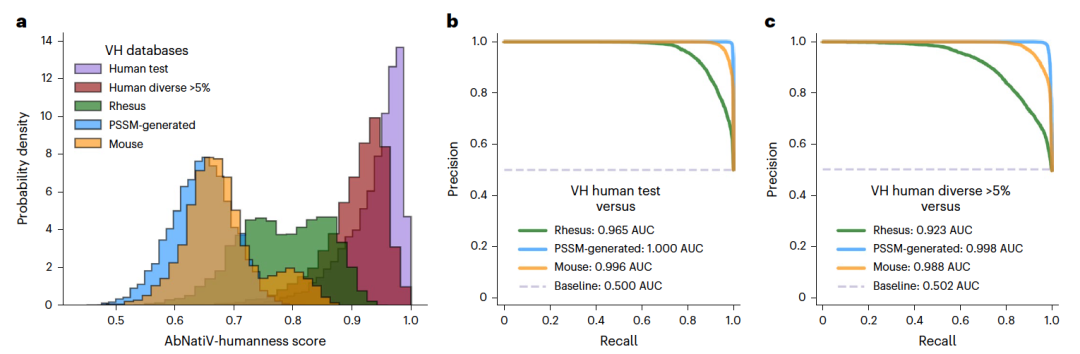
图 2
为了量化AbNatiV的性能,作者首先评估了它区分人类抗体Fv序列和其他物种抗体Fv序列的能力(图2)。使用ROC-AUC和PR-AUC来量化模型正确分类序列的能力。AbNatiV可以准确地区分其测试集中的人类VH序列和小鼠VH序列,PR-AUC为0.996,ROC-AUC为0.995。同样,AbNatiV也可以成功地区分人类和恒河猴(Macaca mulatta)的序列。尽管这两个物种在遗传上非常相似,模型仍能非常好地分离VH序列,PR-AUC为0.965,ROC-AUC为0.958。此外,作者使用了两个验证数据集来进行基准测试:一个用于学习高阶关系,另一个用于确认没有过拟合并验证模型对未见序列空间的泛化能力。对于后者,作者编译了一个由高度多样化的人类Fv序列组成的数据集,称为多样化大于5%的数据集(与训练集中任何序列的差异至少为5%)。分类性能在这个多样化数据集上略有下降,但总体仍然非常高。在VH模型中,与恒河猴序列相比,PR-AUC从0.965下降到0.923。然而,VH模型仍然能够将大多数多样化大于5%的序列分类为人类。只有5.5%的序列的分数低于0.8的自然性阈值。对于轻链模型,性能更为相似。
在抗体治疗的应用
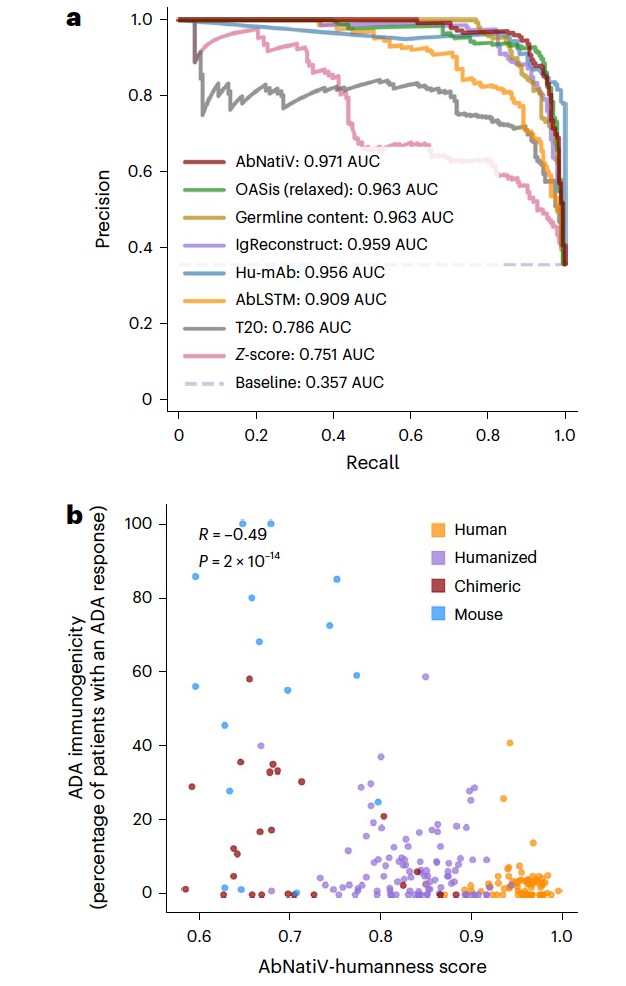
图 3
在抗体药物开发中,评估抗体人类可用性是一个关键步骤,目的是确保药物候选物对患者的最小风险。因此,作者使用AbNatiV对治疗性抗体序列进行了运算,并从相关的AbNatiV模型(即针对VH、Vκ或Vλ训练的模型)计算了重链和轻链的平均人性化分数。更具体地,作者评估了该方法区分196个人类治疗性抗体和353个非人类来源的抗体治疗剂的性能(图3)。AbNatiV在考虑两种AUCs时表现优于所有其他方法,PR-AUC为0.971,ROC-AUC为0.979。在AbNatiV之后的第二好方法是OASis,PR-AUC为0.963,ROC-AUC为0.975,以及Hu-mAb,ROC-AUC为0.979,PR-AUC为0.956。在抗体评估方面的一个核心兴趣是减少它们在人类免疫系统中的免疫原性。评估早期临床试验中的免疫原性的一种方法是评估接受治疗性抗体治疗的患者中发展抗药物抗体(ADAs)的比例。作者发现AbNatiV人性化分数与216种不同治疗性抗体治疗中发展ADAs的患者比例之间存在皮尔逊相关系数为-0.49。作者注意到这些ADA数据是高度异质的,因此很难更强的相关性。
天然骆驼科纳米抗体
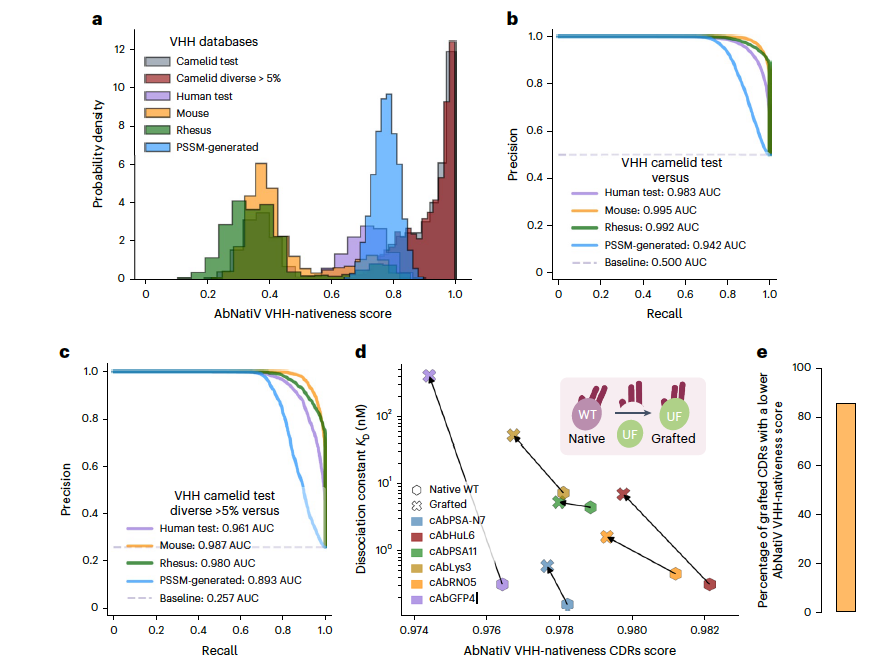
图 4
自2019年首个基于纳米抗体的治疗药物Caplacizumab获批以来,单域抗体的发展势头更加强劲。纳米抗体(VHH)天然存在于骆驼科动物中,它们展现出有利的稳定性和溶解性特性,结合小尺寸能够实现更好的组织穿透,同时保持与完整长度抗体相同的亲和力和特异性。当在VHH序列上训练时,AbNatiV返回一个VHH自然性分数,该分数量化了抗体序列与天然骆驼科单域抗体的相似度。作者发现AbNatiV准确地区分了测试集中的VHH序列和人类(PR-AUC为0.983)、小鼠(0.995)及恒河猴(0.992)的VH序列。使用PSSM生成的人工VHH序列与测试集中真实骆驼科VHH序列之间的PR-AUC为0.942。VHH模型能够将大多数多样化大于5%的VHH序列分类为天然,其性能与测试集上观察到的性能相当。此外,多样化大于5%的VHH序列中有10.4%的分数低于0.8的自然性特征阈值,相比之下,测试VH序列为10.8%。据作者所知,AbNatiV是第一个量化纳米抗体自然性的方法。因此,为了与不同的模型比较,作者在我们的纳米抗体训练集上重新训练了最初为人类VH序列开发的AbLSTM架构。作者发现AbNatiV在所有任务上的分类性能都高于重新训练的AbLSTM模型,尤其是在与VHH多样化大于5%数据集的分类中。
编译 | 曾全晨
审稿 | 王建民
参考资料
Ramon, A., Ali, M., Atkinson, M. et al. Assessing antibody and nanobody nativeness for hit selection and humanization with AbNatiV. Nat Mach Intell 6, 74–91 (2024).
https://doi.org/10.1038/s42256-023-00778-3



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


