今天为大家介绍的是来自清华研究大学团队的一篇论文。单细胞RNA测序(scRNA-seq)数据是理解“生命之语”的强大工具,能为各种生物医学任务提供新见解。近来,大规模语言模型(LLMs)开始用于细胞表征学习。但现有基于BERT架构的细胞表征学习方法存在问题,它们产生的嵌入空间不均匀,导致语义表达效率不高。对比学习通过均匀分布嵌入来解决这个问题。然而,对比学习中更大的批量大小能带来更好的表征,但scRNA-seq数据的高维性和LLMs的大参数量限制了其实际应用。为解决这个问题,作者提出了一种新颖的“分而治之”对比学习方法,它能够解耦批量大小和GPU内存大小的关系,用于细胞表征学习。基于这种方法,作者介绍了单细胞语言模型(CellLM),这是一个大规模的细胞表征学习模型,能够处理包含成千上万基因的高维scRNA-seq数据。CellLM拥有超过5000万个参数,利用200万个scRNA-seq数据进行训练,它是首次尝试从正常细胞和癌细胞中学习细胞语言模型。CellLM在所有评估的下游任务中都达到了新的最先进水平。

单细胞RNA测序(scRNA-seq)数据是理解生命语言的有力工具,可为生物医学任务提供洞见。大规模语言模型(LLMs)最近开始用于解码生命的编码语言,并在此领域取得了一定成功。尤其是近期的研究显示,LLMs在表征单细胞数据方面有效且可行。然而,这些方法完全依赖于BERT架构进行细胞表征,研究发现直接应用BERT可能会因嵌入空间的各向异性而导致表征质量下降。具体来说,低频词在预训练过程中没有得到有效训练,导致它们的嵌入向量在特征空间中分布稀疏。而高频词的嵌入向量则倾向于在特征空间中聚集。这种嵌入空间的不均匀分布限制了衡量高频词和低频词之间语义联系的能力。同样,scRNA-seq数据中基因表达频率的多样性表明,BERT架构的缺陷也会影响scRNA-seq数据的语义表征过程。
通过学习正样本和负样本特征,对比学习通过均匀分布嵌入来解决这个问题。然而,对比学习的一个基本挑战在于确保每个训练批次中有足够多的负样本,这对于模型学习有效特征至关重要。不幸的是,这个挑战由于GPU内存大小的限制而加剧,尤其是在最常见的端到端对比学习方法中。针对scRNA-seq数据,数据维度由细胞中的基因数量决定,可高达19379个已知编码蛋白的基因,如果包括非编码基因则更高。scRNA-seq数据的高维性和LLMs的庞大参数量使得在使用对比学习进行细胞表征任务时,达到大批量大小变得具有挑战性,尤其是在GPU内存大小受限的情况下。因此,将批量大小与GPU内存大小分离至关重要。
为了克服批量大小的限制并确保正负样本由同一编码器生成,作者提出了一种新颖的“分而治之”对比学习方法,将批量大小与GPU内存大小分离用于细胞表征学习。更重要的是,这种方法在数学上严谨,并已被证明与端到端对比学习方法完全等效。这意味着我们可以在不引入任何额外错误的情况下增加批量大小,同时更新正负样本的编码器。通过利用时间换空间的概念,一个大批次被分成几个小批次。然后顺序进行梯度更新计算,允许在不影响正负样本编码器更新同步性的情况下增加适当的批量大小,更有效地利用可用的GPU内存资源,同时保持编码器更新的一致性。
模型部分
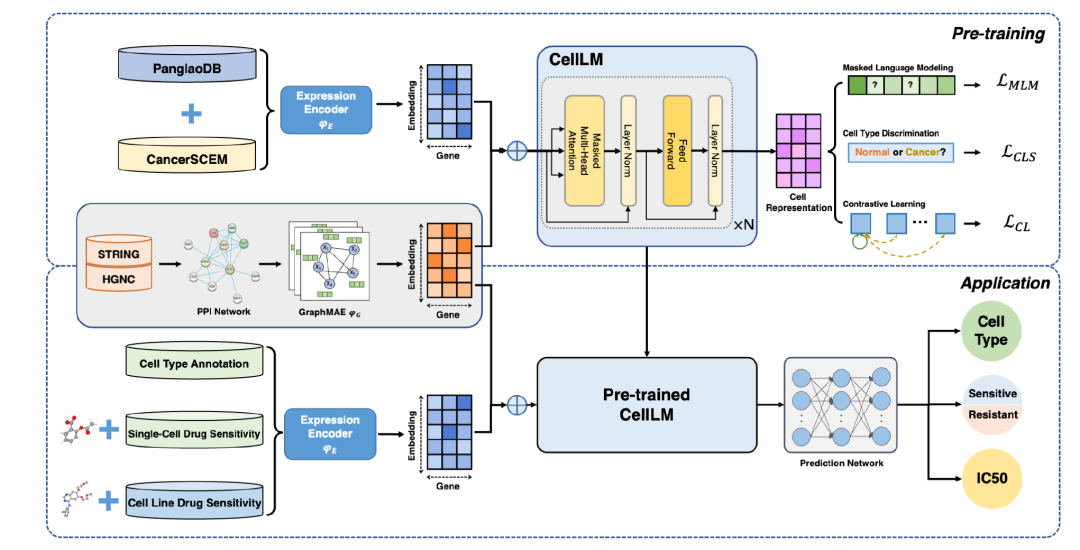
图 1
模型CellLM的结构如图1所示。CellLM在PanglaoDB和CancerSCEM上进行了预训练。在预训练过程中,基因表达数据和基因相互作用数据首先被编码,然后相加在一起,之后再输入模型进行自监督学习。在预训练阶段,作者使用了三种自监督任务:掩码语言建模(MLM)、细胞类型鉴别,以及一种新颖的“分而治之”对比学习方法。在应用方面,作者评估了预训练后的CellLM在单细胞和细胞系数据层面上进行细胞类型注释和药物敏感性任务的效果。这意味着CellLM不仅可以应用于理解单个细胞的特性,还能在更广泛的细胞群体(如细胞系)中进行有效的药物敏感性预测和分类。
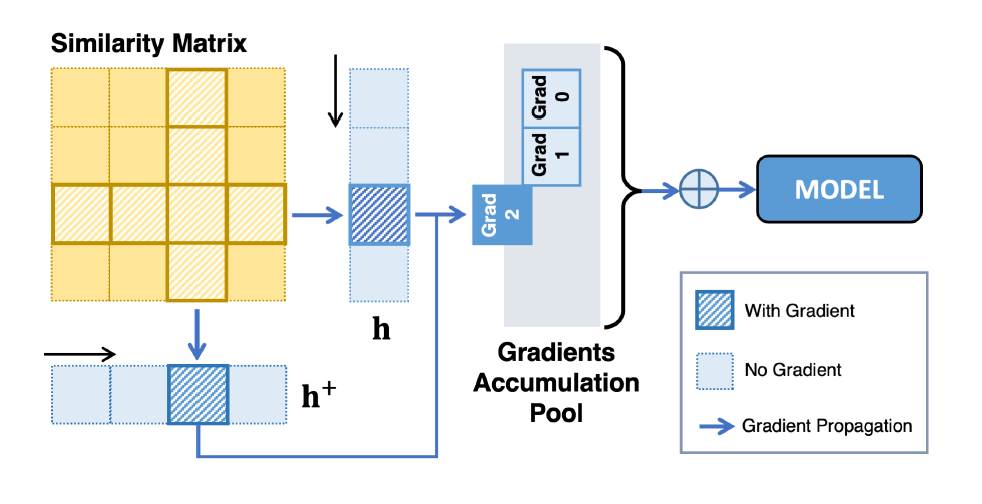
图 2
为了解决基于BERT方法导致的表征退化问题,并增强细胞表征的编码能力,作者引入了对比学习。在对比学习中,构建正负样本以进行数据增强是一个关键步骤,这有助于模型更好地理解数据特征。在单细胞RNA测序(scRNA-seq)数据中,每个基因表达水平都有其独特意义,传统的数据增强方法可能会破坏基因表达的语义。因此,作者认为在特征层面进行扰动更适合scRNA-seq数据的数据增强。为此,作者使用dropout层应用于同一单细胞来构建正样本,而同一批次中的其他单细胞则作为负样本。由于模型的庞大参数量和输入序列的高维度,要在有限的GPU内存大小下用大批量大小训练模型是一个挑战。然而,对比学习需要大批量大小以提供足够的负样本。因此,作者设计了“分而治之”对比学习方法(图2为方法图列),将批量大小与同时处理的实际数据量分离。这使模型能够在保留内存限制的同时,使用更大的有效批量大小来训练模型。与此同时,这种方法保留了端到端对比学习的优势,包括编码器的同步更新和对正负样本的无损比较。
实验部分
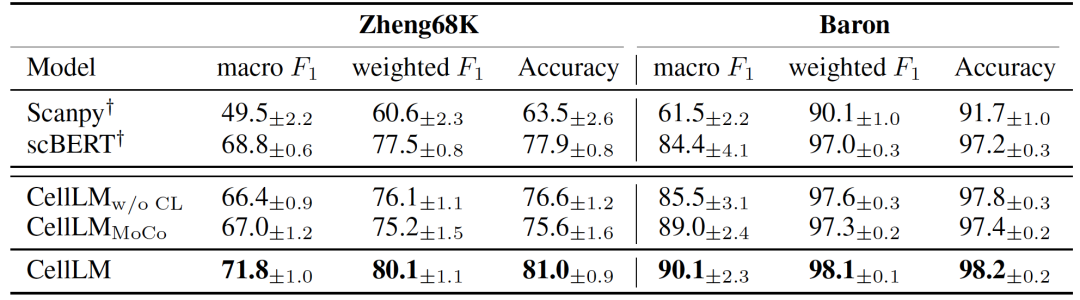
表 1
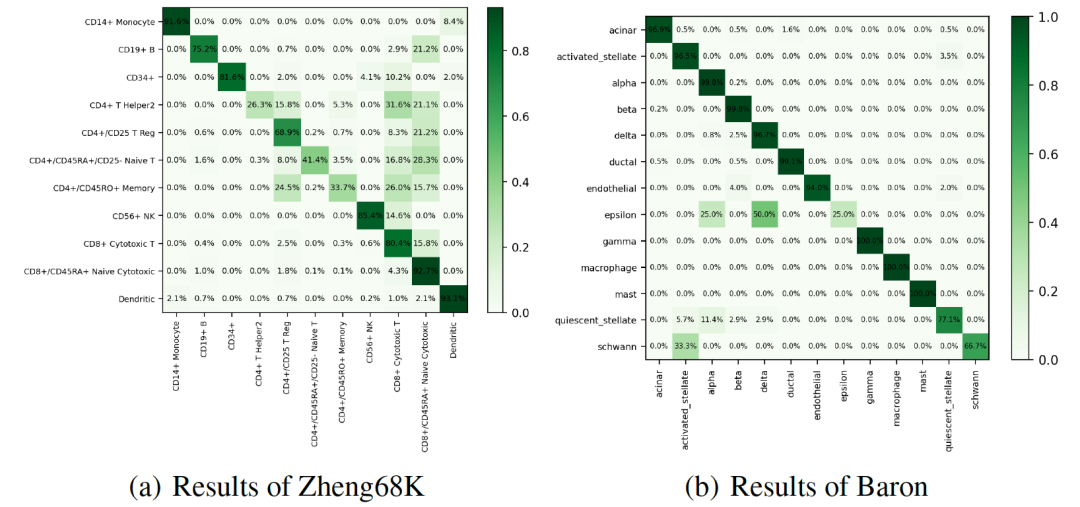
图 3
首先,作者评估了单细胞语言模型(CellLM)在单细胞类型注释任务上的表征能力。表1中的结果和图3中混淆矩阵的热图显示,模型在Zheng68K和Baron数据集上达到了最先进(SOTA)的性能。值得注意的是,与当前领先的模型scBERT相比,F1分数分别提高了3.0%和5.7%。作者的方法成功优化了BERT的原始特征空间,并显著改善了由嵌入空间的各向异性引起的表征退化问题。
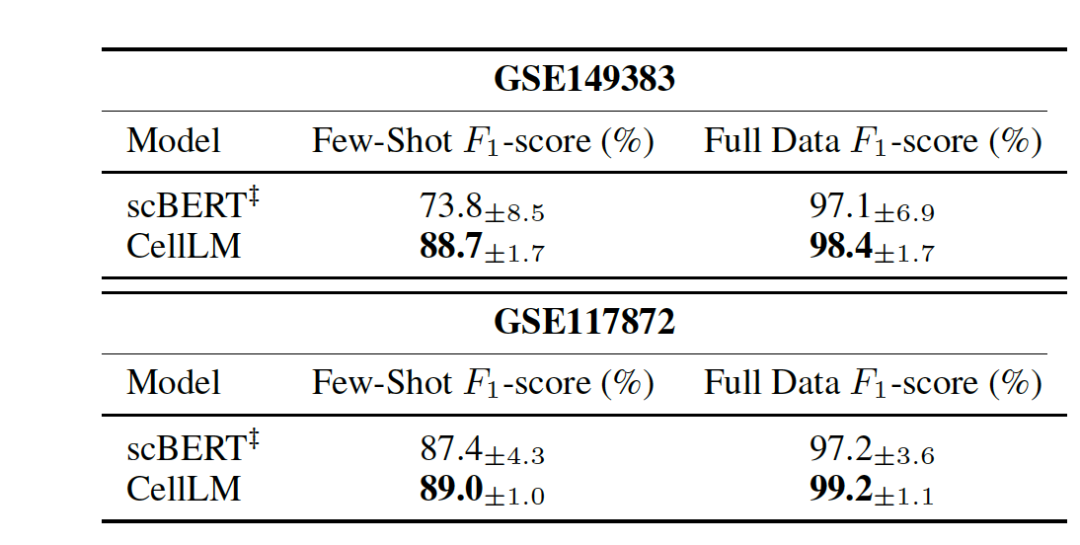
表 2
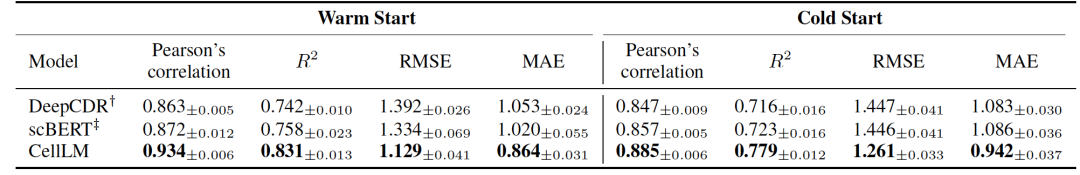
表 3
在单细胞药物敏感性预测实验中,作者既进行了few-shot实验,也进行了全数据情况实验。在few-shot实验中只使用5%的可用数据样本进行训练。相反,全数据实验使用了80%的数据进行训练。实验结果总结在表2中。与scBERT相比,CellLM在两种情况下均显示出显著改进。具体来说,在few-shot情况下,CellLM平均提高了8.25%。在全数据情况下,平均提高了1.65%。这些实验结果突显了CellLM在理解癌症方面的增强,因为它超越了仅在正常人类组织上预训练的scBERT。纳入癌症数据使CellLM能够捕捉癌症特有的特征,从而带来更优越的表征,并在癌症药物虚拟筛选中显著提升性能。表3展示了在细胞系上进行的药物敏感性预测实验结果。作者模拟了两种类似于现实世界药物再利用的情景:细胞系热启动和细胞系冷启动。在热启动情景中,数据被随机分为训练、验证和测试集。相反,在冷启动情景中,测试集包含训练期间未见过的细胞系。这要求模型预测以前从未遇到过的新细胞系的药物反应。与DeepCDR相比,CellLM在两种情境下的皮尔森相关系数中分别显示了6.2%和3.8%的绝对提升。实验结果表明,CellLM从单细胞转录组学中获得的知识在转移到细胞系时仍然有效。此外,CellLM的性能超过了目前单组学数据的方法。
参考资料
Zhao, S., Zhang, J., & Nie, Z. (2023). Large-Scale Cell Representation Learning via Divide-and-Conquer Contrastive Learning. arXiv preprint arXiv:2306.04371.





 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง




