蛋白质功能注释一直是生物学面临的关键挑战之一,在指导新型药物靶标发现中具有重要作用。然而,当前方法在注释那些仅有少量蛋白的功能家族时,仍面临“长尾问题”的严峻挑战。针对此问题,浙江大学药学院朱峰教授与阿里云李兆融工程师组成联合研发团队,在国际权威期刊Genome Biology发表研究工作,提出了一种名为AnnPRO的基于序列的多尺度蛋白质表征和混合深度学习框架的蛋白质功能注释新策略。

研究背景
蛋白质是生命的基本组成单位,它们的功能决定了生物体的结构和活动。然而,目前只有很少一部分的蛋白质的功能是通过实验方法得到验证的,大量的蛋白质的功能还是未知的。因此,如何利用计算方法来预测蛋白质的功能,是生物学领域的一个重要而又困难的问题。
目前,已经有许多计算方法被开发出来,用于预测蛋白质的功能。这些方法主要可以分为两类:一类是基于序列相似性的方法,它们通过比较蛋白质的序列,来推断它们的功能;另一类是基于机器学习的方法,它们通过学习蛋白质的特征,来预测它们的功能。然而,这些方法都存在一个共同的问题,就是在预测那些功能类别较少的蛋白质时表现不佳。这是因为蛋白质在不同的功能家族中的分布是不均匀的,有些功能类别的蛋白质很多,而有些功能类别的蛋白质很少。这种数据分布导致了一个“长尾问题”,即少数的“头部”功能类别占据了大部分的蛋白,而大量的“尾部”功能类别只占据了少数的蛋白。这种“长尾问题”使得现有的方法在训练过程中,往往忽略了“尾部”功能类别的信息,导致它们在预测时,对蛋白的“尾部”功能的预测准确度很低。
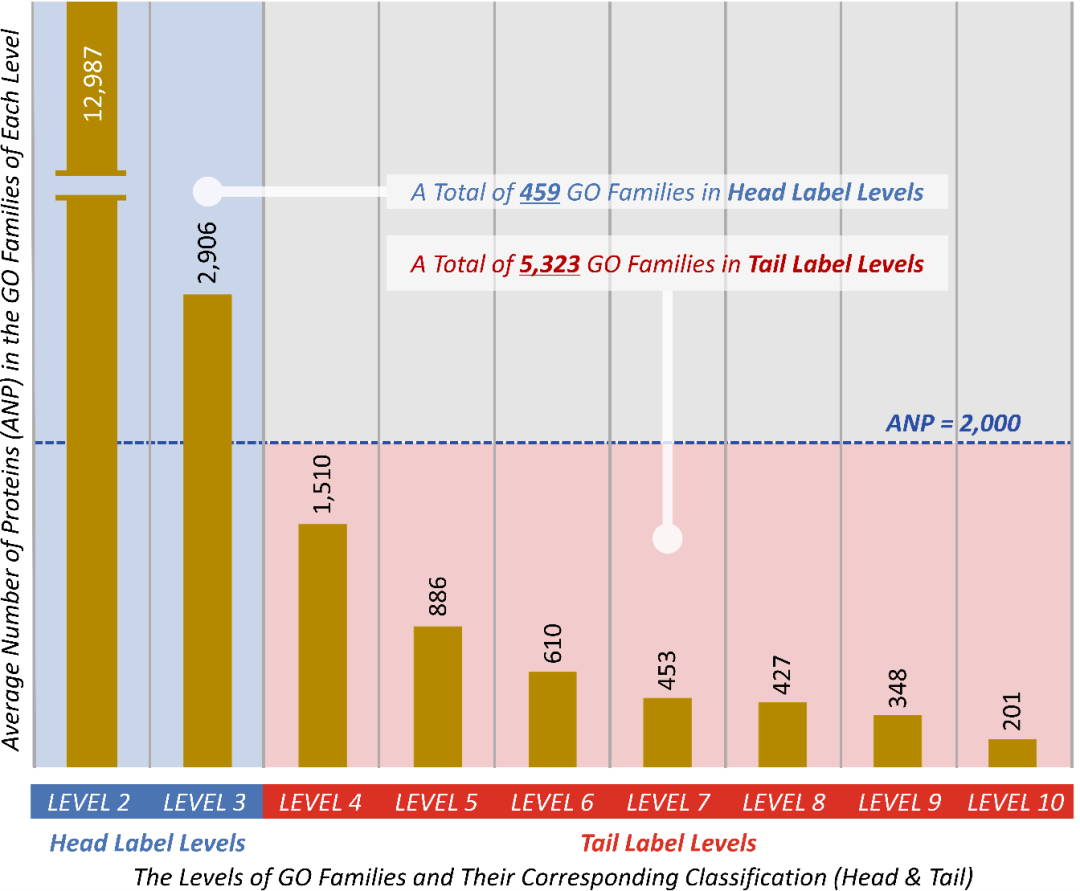
图1. 蛋白质GO层级及数量
模型框架
为了解决蛋白功能预测中的“长尾问题”,一种新的蛋白功能预测方法AnnoPRO被提出。AnnoPRO的核心思想是利用多尺度的蛋白质表征方式,双路径的蛋白质编码网络,和基于LSTM的蛋白质解码模型,来实现对蛋白质功能的预测。具体来说,AnnoPRO首先将蛋白质的序列转换为两种不同的表示形式:一种是基于特征相似性的图像,它能够捕捉蛋白质特征之间的内在关联;另一种是基于蛋白质相似性的向量,它能够考虑蛋白质序列之间的全局关联。然后,AnnoPRO利用一个双路径的编码网络,分别对这两种表征进行编码,得到蛋白质的特征向量。这个编码网络是基于预训练的,即利用已知的蛋白质功能注释数据,来优化网络的参数。最后,AnnoPRO使用一个基于LSTM的解码网络,将蛋白质的特征向量作为输入,输出蛋白质的功能类别。这个解码网络是基于多标签的,即可以同时预测蛋白质属于多个功能类别的概率。
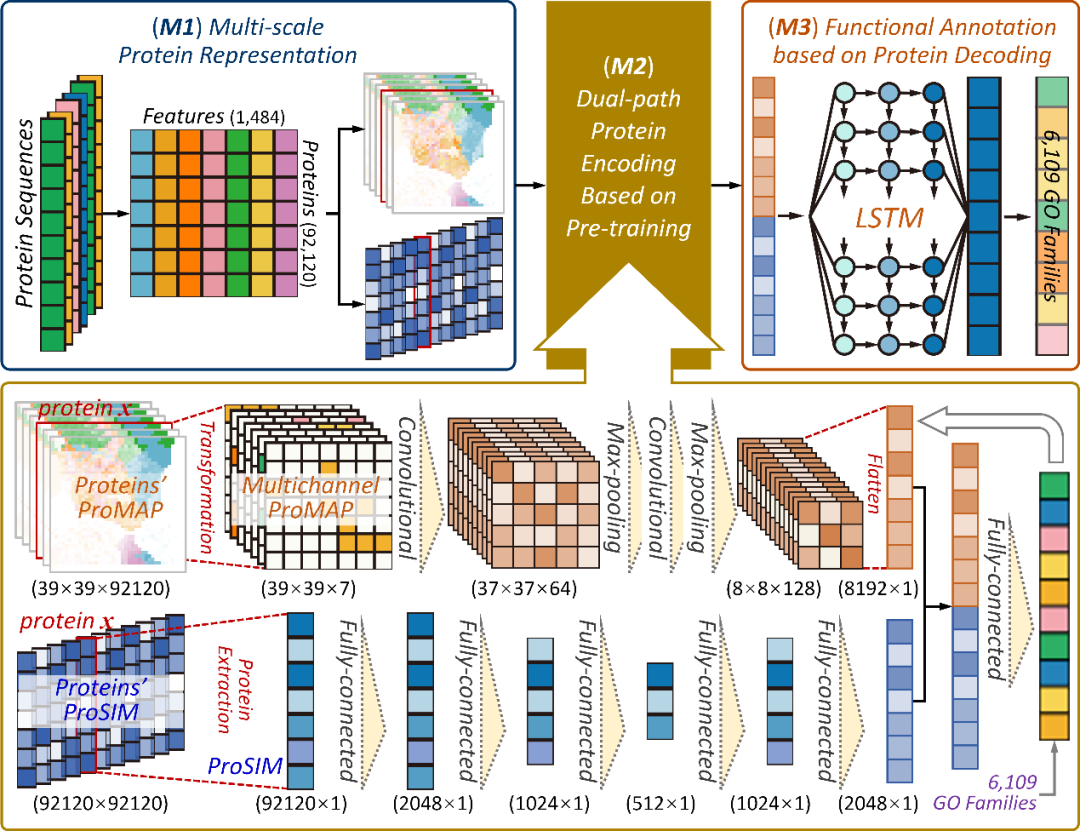
图2. AnnoPRO深度学习混合模型框架
模型效果
为了验证模型性能,研究者们使用了多个基准数据集,来与现有的方法进行比较。结果表明,AnnoPRO在所有的功能类别(生物过程,分子功能,细胞组分)上,都取得了最好的预测效果,显著优于其他的方法。特别是在“尾部”功能类别上,AnnoPRO的预测准确度有了显著的提升,达到了解决“长尾问题”的目标。
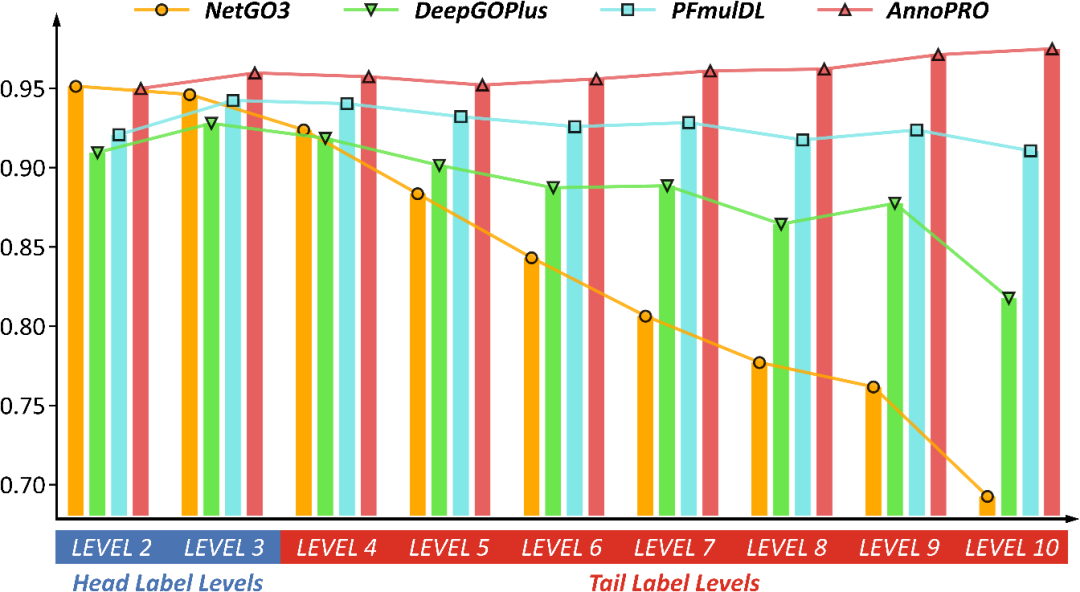
图3. AnnoPro 在不同层级上的性能比较
此外,AnnoPRO还能够成功地捕捉到由于序列变异导致的蛋白质功能变化,例如生长分化因子(GDFs)和热休克蛋白(HSPs)等。如下图所示,GDF8和GDF11之间只有三个氨基酸的差异,导致GDF8具有肝素(herprin)结合功能(GO:0008201),而GDF11的变异残基使其无法与FS288交互,因此失去了这一功能。AnnoPRO可以成功预测两种GDFs的所有GO家族。
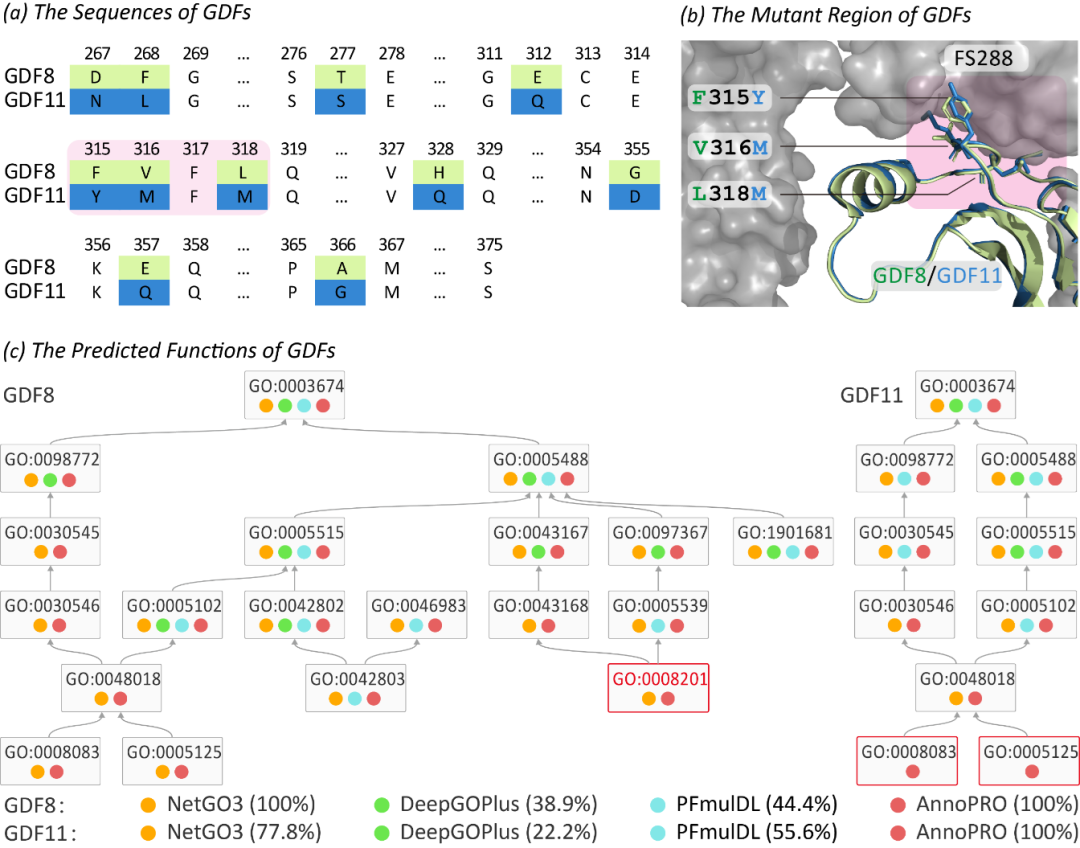
图 4. 不同方法在GDFs的功能预测结果
AnnoPRO是一种基于深度学习的蛋白质功能注释新方法,它能够充分利用蛋白质序列的信息,提高蛋白质功能的预测能力。AnnoPRO的研究成果,为蛋白功能预测领域提供了一个新的思路和方法,也为相关的生物学研究,如新药靶点的发现,疾病机理的揭示等,提供了有力的支持。
参考资料
Zheng, L., Shi, S., Lu, M. et al. AnnoPRO: a strategy for protein function annotation based on multi-scale protein representation and a hybrid deep learning of dual-path encoding. Genome Biol 25, 41 (2024). https://doi.org/10.1186/s13059-024-03166-1



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


