
Uni-Dock 是由深势科技开发的高性能分子对接软件,能在 GPU 上以超过单 CPU 千倍的速度完成虚拟筛选任务。2023 年 6 月,Uni-Dock 以封面文章形式发表在 JCTC 杂志上[1];同年 8 月,Uni-Dock 加入 DeepModeling 社区,在 LGPL 协议下开放源代码,并发布 v1.0 版本。在过去的半年中,Uni-Dock 得到了学术界和工业界的关注、支持和应用。Uni-Dock 已实践于多个早期药物发现项目,被多个软件平台集成和收录,并为药物设计领域的 AI for Science 方法开发提供了高效的数据标记、评估和预测的能力。Uni-Dock 已经成为药物研发工作者和 AI for Science 方法开发者的优选工具。作为 DeepModeling 社区的孵化软件,Uni-Dock 以开放的姿态拥抱开源社区。广大用户的使用反馈和开源社区开发者的贡献,共同推动了 Uni-Dock 的持续优化和进步,Uni-Dock 的开源协作模式也随着软件迭代过程变得更加明确和规范。2024年2月,Uni-Dock 发布 v1.1 版本。新版本合并了近半年以来,包括深势科技、NVIDIA 等在内的开发者贡献的众多新特性,分子对接和虚拟筛选效率进一步提升;同时,Uni-Dock Tools 工具包的发布为广大用户带来了更好的使用体验和优秀的实践案例;另外,新版本中发布了 Uni-Dock Benchmarks 基准测试集、完善了 Uni-Dock CI/CD 流程等开发套件,并明确了 Uni-Dock 的开源协作模式,为更广泛的开源合作提供了基础。Uni-Dock v1.1 代码可从以下 GitHub 仓库获取:
https://github.com/dptech-corp/Uni-Dock/releases/tag/1.1.0

Uni-Dock
LOGO标识首次公开


Uni-Dock 的 LOGO 标识为一个戴着帽子的吃豆人的形象延展,由青色的配体分子、蓝色的受体蛋白和紫色的筛选漏斗组成,展现了 Uni-Dock 在虚拟筛选过程中的主要元素。
Uni-Dock v1.1
更快的分子对接、更快的虚拟筛选
Uni-Dock v1.1 在分子对接和虚拟筛选的计算效率上得到了进一步的显著提升。
分子对接特指单个配体分子向单个受体蛋白的对接计算过程,常常用于受体蛋白-配体分子结合复合物的结构预测,以获得两者的结合模式来开展相互作用分析,指导理性药物设计。在 Uni-Dock v1.1 版本中,分子对接的主要提升如下:
- 实现了单配体单分子对接计算模式:在原有的单配体多分子的计算流程基础上,使用 CUDA stream 轻量化地实现 1:1 docking 的快速计算,并提供了相关计算案例与配置文件结构。
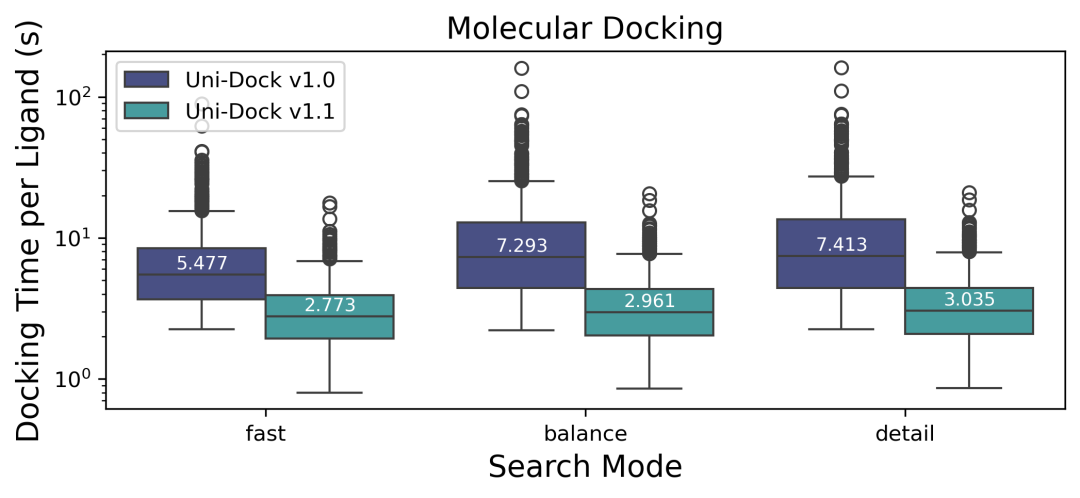
在基准测试机型上,Uni-Dock v1.1 的单分子对接速度分别是 fast 模式下 2.773s/ligand(提升 194%),balance 模式下 2.961s/ligand(提升 246%),detail 模式下 3.035s/ligand(提升 244%)。
虚拟筛选特指一批配体分子向单个受体蛋白的对接计算过程,常常用于虚拟分子库面向特定受体蛋白的快速筛选评估,以获得对该受体蛋白具有潜在活性的配体分子。在 Uni-Dock v1.1 版本中,虚拟筛选的主要提升如下:
-
为 GPU kernel 设定合适的 launch bound 以规避寄存器溢出问题。
-
为对接核心算法蒙特卡洛模拟实现了 Warp-level 并行优化,进一步提升资源利用率以加速计算。
- 优化任务调度策略:按照配体原子数及可旋转键数量分类并使用模版生成相应的计算代码,节省每个配体占用的显存空间,进一步提升单个批次内计算的配体数目;针对此优化更新显存预估方法,提升了显存的有效利用率,对于配体尺寸分散的小分子数据库有明显加速。
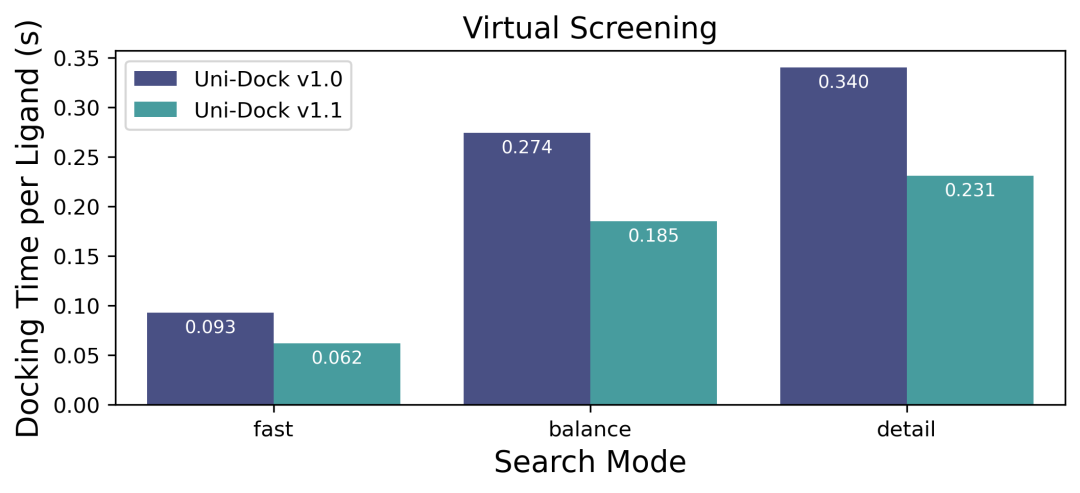
在基准测试机型上,Uni-Dock v1.1 的虚拟筛选速度分别是 fast 模式下 0.062s/ligand(提升 150%),balance 模式下 0.185s/ligand(提升 148%),detail 模式下 0.231s/ligand(提升 147%)。Uni-Dock v1.1 为计算能力(compute capability)更高的 NVIDIA GPU 开展了进一步的适配,在最新消费级旗舰 GPU 上,虚拟筛选的速度能稳定达到 fast 模式 0.030s/ligand,balance 模式 0.067s/ligand,detail 模式 0.082s/ligand。现在,使用 Uni-Dock v1.1 fast 模式开展 1 亿分子的大规模虚拟筛选,100 卡 GPU 集群最快只需要运行 8.3 个小时就能完成全部任务。
Uni-Dock Tools
分子对接工具包
我们发布了 Uni-Dock Tools 分子对接工具包,给广大用户提供了更好的使用体验和优秀的实践案例。
Uni-Dock Tools 整合了受体蛋白准备工具 proteinprep 和配体分子准备工具 ligandprep 等实用工具,补全了 Uni-Dock 使用过程中的前置文件处理方法。Uni-Dock Tools 还整合了 unidock-pipeline 分子对接工作流,能够接受主流格式的受体蛋白和配体分子文件作为输入,并自动执行 proteinprep 和 ligandprep 进行准备工作。
Uni-Dock Tools 和 Uni-Dock v1.1 的使用案例可见:
https://nb.bohrium.dp.tech/detail/48759512168
Uni-Dock Tools 中还收录了基于 Uni-Dock 开发的多构象对接方法(Multi-Conformation Dock,MCDock)。MCDock 充分利用 Uni-Dock 的快速对接能力,通过大量的构象枚举、多构象刚性对接、优秀构象的局部优化,来可以进一步提升对受体蛋白-配体分子复合物结合构象的采样空间覆盖率,来获得更为准确的复合物构象。MCDock 的详细说明和使用案例可见:https://nb.bohrium.dp.tech/detail/91221652314您可以访问: https://github.com/dptech-corp/Uni-Dock/tree/main/unidock_tools
获取分子对接工具包的最新版本。

Uni-Dock Benchmarks
基准测试集
为了在统一标准下评估软件性能和精度,我们整理并发布了 Uni-Dock 使用的基准数据集 Uni-Dock Benchmarks。Uni-Dock Benchmarks 以公开数据集或文献数据为基础整理,面向 Uni-Dock 的适用场景对原始数据进行了清洗,目前包含分子对接基准数据集3个(含有 84 个复合物体系的 Astex 数据集、含有 271 个复合物体系的 CASF-2016 数据集和含有 428 个复合物体系的 PoseBuster 数据集),以及虚拟筛选基准数据集5个(含有 824 个配体分子的 D4 数据集、含有 824 个配体分子的 sigma2 数据集、含有 3580 个配体分子的 NSP3 数据集、含有 7321 个配体分子的 PPARG 数据集和含有 458491 个配体分子的 GBA 数据集)。您可以访问: https://github.com/dptech-corp/Uni-Dock-Benchmarks获取基准数据集的最新版本。
Uni-Dock
开源协作规范更新
Uni-Dock 作为 DeepModeling 社区的孵化软件,依照 LGPL 协议开放源代码。我们欢迎广大用户使用并提出问题、建议和需求,亦欢迎开源社区开发者共同参与 Uni-Dock 的优化升级。为了便于开源社区开发者的协同,我们在此次版本更新中一并发布了社区建设的相关文档。如果您遇到了安装、使用上的问题,或是希望 Uni-Dock 增加一个新的特性,请向 Uni-Dock 的 GitHub 仓库提交一个新的 issue,根据 issue 表单的指引提供必要信息;如果您希望为 Uni-Dock 贡献新功能,欢迎按照贡献指南更新代码。具体如下:
1. 提交issue:
https://github.com/dptech-corp/Uni-Dock-Dev/issues/new
2. 贡献指南:
https://github.com/dptech-corp/Uni-Dock-Dev/blob/main/.github/CONTRIBUTING.md
3. 代码格式规范:
https://github.com/dptech-corp/Uni-Dock-Dev/blob/main/unidock/.clang-format
Uni-Dock 为计算能力(compute capability)为 7.0 及以上的 NVIDIA GPU 提供官方支持。

Uni-Dock CI/CD
更规范的测试部署流程
软件测试有助于发现开发过程中的潜在问题,确保计算的精度与速度符合预期;自动化的部署流程能帮助用户及开发者更便捷地评估最新开发版本;充分的测试体系可以为用户和开发者建立对软件的信任,促进社区成员的协同合作,并帮助开发者实践良好的开发流程。
作为开源软件,Uni-Dock 持续完善测试用例及测试框架,做好迎接更多合作者的准备。此次版本发布,我们完善了 Uni-Dock 及 Uni-Dock Tools 的测试框架,加入了新的单元测试及集成测试用例;基于 GitHub Actions 的工作流会在新 PR 被接收时构建新的 Docker 镜像并发布至 Docker Hub:
dptechnology/unidock;
dptechnology/unidock_tools。
Uni-Dock
其他新特性与错误修复
功能更新
-
Uni-Dock Tools 支持 PDB 格式蛋白解析,支持无氢键格式配体解析 #19
-
添加项目代码标准格式化命令支持 #46
-
优化配体显存估计逻辑 #56
-
更新配体数据扭转构造规则 #58
-
添加氢键计算支持 #64
bug 修复
-
修复编译期警告 #17
-
删除项目冗余代码 #46
-
修复配体索引选项错误 #48
-
修复逻辑判断符号 #49
-
修复缺少 CUDA 内存初始化导致崩溃的问题 #63
-
修复了 Vina 打分函数的内存泄漏 #63

贡献者
以下为各位贡献者的 GitHub ID(依照字母顺序排列)。
@caic99 @envidian @Hong-Rui @hypnopump @lucifer1004 @pkuyyj @prabindh @rongfengzou @UR-Free @ysyecust @YNYuan @zhangjingrong @zhengh96 @zmsunnyday
相关链接
Uni-Dock 开源仓库:https://github.com/dptech-corp/Uni-Dock/Uni-Dock Bohrium®APP:https://app.bohrium.dp.tech/uni-dock/Hermite®药物计算设计平台:https://hermite.dp.tech/
参考文献
[1] Yu, Y., Cai, C., Wang, J., Bo, Z., Zhu, Z., & Zheng, H. (2023). Uni-dock: Gpu-accelerated docking enables ultralarge virtual screening. Journal of Chemical Theory and Computation, 19(11), 3336-3345.
推荐关注
关于深势科技
深势科技是“AI for Science”科学研究范式的引领者和践行者,致力于运用人工智能和多尺度的模拟仿真算法,结合先进计算手段求解重要科学问题,为人类文明最基础的生物医药、能源、材料和信息科学与工程研究打造新一代微尺度工业设计和仿真平台。
我们开创性地提出了「多尺度建模+机器学习+高性能计算」的革命性科学研究新范式,并推出了Bohrium®科研云平台、Hermite®药物计算设计平台、RiDYMO®难成药靶标研发平台及 Piloteye®电池设计自动化平台等工业设计与仿真基础设施,颠覆了现有研发模式,打造“计算引导实验、实验优化设计”的全新范式。
深势科技是国家高新技术企业、国家专精特新“小巨人”企业,总部位于北京,并在上海、深圳等城市布局研发中心。科研技术团队由中国科学院院士领衔,汇集了超百位数学、物理、化学、生物、材料、计算机等多个领域的优秀青年科学家和工程师,其中公司的博士及博士后占比超过35%。核心成员获得过2020年全球计算机高性能计算领域的最高奖项“戈登贝尔奖”,相关工作当选2020年中国十大科技进展和全球AI领域十大技术突破。








 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง





