今天为大家介绍的是来自Jakob Nikolas Kather团队的一篇论文。深度学习(DL)能够从癌症组织病理学中预测生物标志物。目前,已有几种获得临床批准的应用采用了这项技术。然而,大多数方法预测的是分类标签,而生物标志物往往是连续的测量值。作者假设基于回归的深度学习在性能上超过了基于分类的深度学习。因此开发并评估了一种自监督的、基于注意力的、弱监督回归方法。

收集和病理检查组织样本是准确诊断恶性肿瘤患者的一种方法,它提供了有关组织学等级、亚型、分期和其他肿瘤生物标志物的信息。数字病理学描述了以整张切片图像(WSI)形式对组织样本进行计算分析的过程。众多研究已经表明,可以直接从WSI预测个体基因的变化以及单个基因的表达或基因组的表达模式。这一研究领域还使得基因变化与形态学模式(即基因型-表型相关性)之间的关联成为可能,这有助于预测患者的预后。与其临床应用一致,其中几种方法已经被监管机构批准用于临床,以至于基于深度学习(DL)从病理诊断流程中预测生物标志物的方法正变得越来越重要。预测基因型-表型相关性,即从WSI预测遗传生物标志物,是深度学习中一个弱监督问题。为了完成这项任务,深度学习模型需要将WSI中的表型特征与从患者级别的肿瘤组织分子遗传测序获得的单一真值相关联。然而,由于这些WSI具有千兆像素级的分辨率,神经网络处理需要将它们分解成较小的区域。但是,这些区域可能包含如结缔组织或脂肪这样的不太相关的组织,可能不会对生物标志物的可预测性做出贡献。为了解决这个问题,基于注意力的多实例学习(attMIL)成为主要的技术方法。将这些值二值化或划分为两部分会导致信息损失,这可能会限制深度学习(DL)系统从病理切片预测这些生物标志物的性能。相反,在组织病理学WSI(整张切片图像)分析中,一个更适合的分类方法可能是回归。回归是一种用来研究变量之间关系的建模方法,例如从WSI中的形态特征与连续数值变量之间的关系。迄今为止,探索这种方法的数据还很少。几项研究探讨了从WSI预测基因表达水平和空间基因表达的不同方法。然而,他们的分析受到样本量小且仅限于单一癌症类型的限制。Graziani等人最近的一项研究提出了一种使用attMIL形式从病理图像预测连续值的方法,但他们的回归网络并没有系统地与更多分类方法进行比较,且需要更广泛的验证。
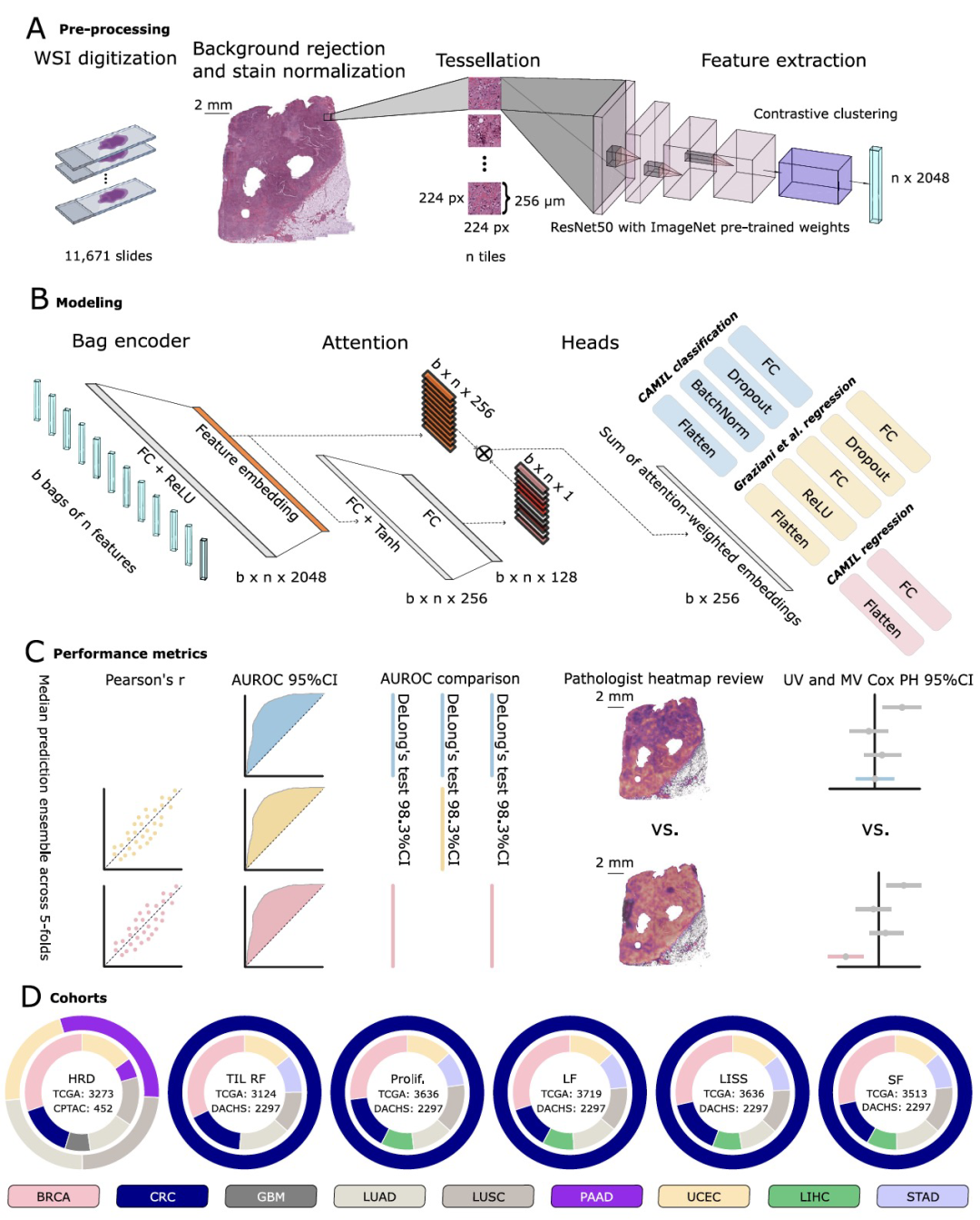
图 1
作者开发了一种基于回归的深度学习(DL)方法,这种方法结合了通过自监督学习(SSL)训练的特征提取器和一个基于注意力的多实例学习(attMIL)模型(图1A, B),称为对比聚类的注意力多实例学习(CAMIL)回归。作者测试了这种方法直接从病理图像预测同源重组缺陷(HRD)的能力。作者选择HRD是因为它是一种泛癌症生物标志物,可以作为一个连续的得分来测量,但在临床验证的截断点可以二值化。作者使用了癌症基因组图谱(TCGA)队列,包括乳腺癌(BRCA)、结直肠癌(CRC)、胶质母细胞瘤(GBM)、肺腺癌(LUAD)、肺鳞状细胞癌(LUSC)、胰腺腺癌(PAAD)和子宫内膜癌(UCEC),来训练每种癌症类型的回归DL模型,并通过交叉验证评估它们的表现(图1C, D)。CAMIL回归模型能够在7种测试的癌症类型中的5种中预测HRD状态,其AUROC超过0.70。AUROC及其95%置信区间(CI)分别是:在BRCA中为0.78 [0.75–0.81],在CRC中为0.76 [0.65–0.87],在GBM中为0.64 [0.37–0.79],在PAAD中为0.72 [0.62–0.81],在LUAD中为0.72 [0.67–0.77],在LUSC中为0.57 [0.52–0.63],在UCEC中为0.82 [0.78–0.86](图2A)。作者在CPTAC上验证了模型,这是一组外部验证队列,在这些队列中,图像和HRD状态对LUSC、LUAD、PAAD、UCEC都是可用的(图2B)。在这些队列中,模型达到了更高的AUROC,分别是在PAAD中为0.68 [0.56–0.79],在LUAD中为0.81 [0.77–0.85],在UCEC中为0.96 [0.93–0.98]。最低的AUROC是在LUSC中为0.62 [0.56–0.67]。这些数据一起表明,基于回归的DL可以单独从病理图像预测HRD状态。
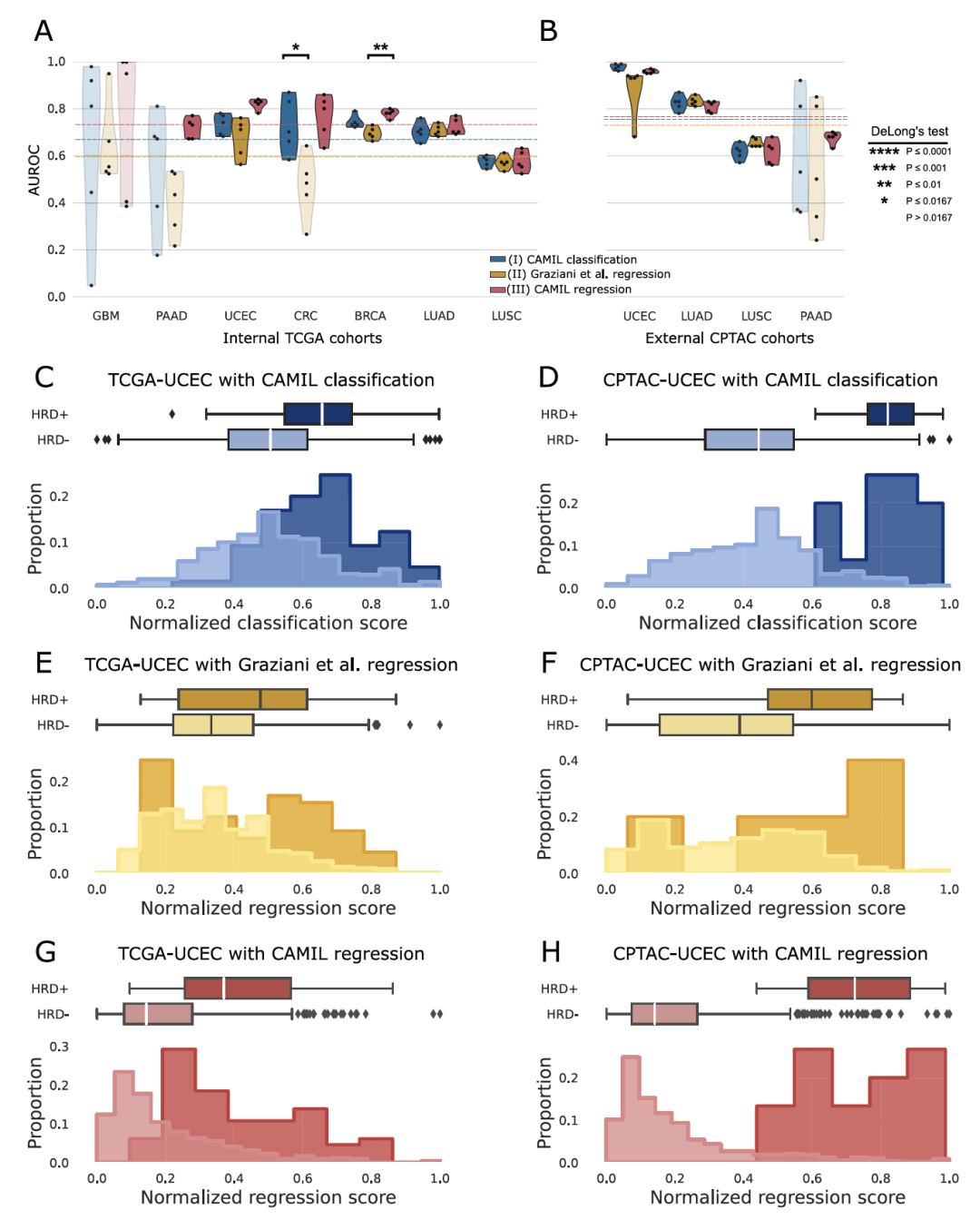
图 2
回归强于分类
作者将CAMIL回归与两种最新方法进行了比较:Graziani等人的回归方法和CAMIL分类方法。为了比较分类和回归,选择了AUROC作为评估指标。在TCGA队列感知分割测试集中,CAMIL回归在7种测试的癌症类型中的5种上超过了之前的两种方法,其中GBM和LUSC展示了相似的AUROC(图2a)。在TCGA-BRCA队列中,CAMIL分类和Graziani等人回归之间观察到显著的性能差异(p ≤ 0.0167)。在外部验证队列中,模型之间的AUROC没有统计学上的显著差异。值得注意的是,CAMIL回归在大多数癌症类型的5折交叉验证中表现出较低的模型性能方差,无论是在内部队列还是外部队列。这些发现表明,与CAMIL分类相比,CAMIL回归在不同患者子集中学习了更鲁棒的特征。因此作者调查了AUROC无法捕捉到的模型性能的其他方面。为此,作者通过量化正负样本的归一化分数中位数之间的绝对距离来比较这三种方法。当可视化模型从HRD预测的分布时,CAMIL分类和Graziani等人的回归方法展示了在HRD+和HRD-患者之间缺乏清晰区分的预测分数。值得注意的是,与其他方法相比,CAMIL回归模型在内部和外部验证队列中展示了HRD+和HRD-患者得分分布之间更明显的分离。作者进一步在所有肿瘤实体中量化了这一点,并发现在所有7个选定的TCGA队列中,这个距离在CAMIL回归中比CAMIL分类大,导致更大的类别可分性。在CPTAC中,与CAMIL分类方法相比,使用CAMIL回归方法在4个队列中的2个上改善了类别可分性。总的来说,与CAMIL分类相比,CAMIL回归方法在TCGA训练队列的测试集上将组间中位数的平均绝对分离距离提高了9.9%,在外部CPTAC测试队列上提高了4.9%。
回归在组织病理学中的生物标志物预测上增强了与临床知识的对应关系
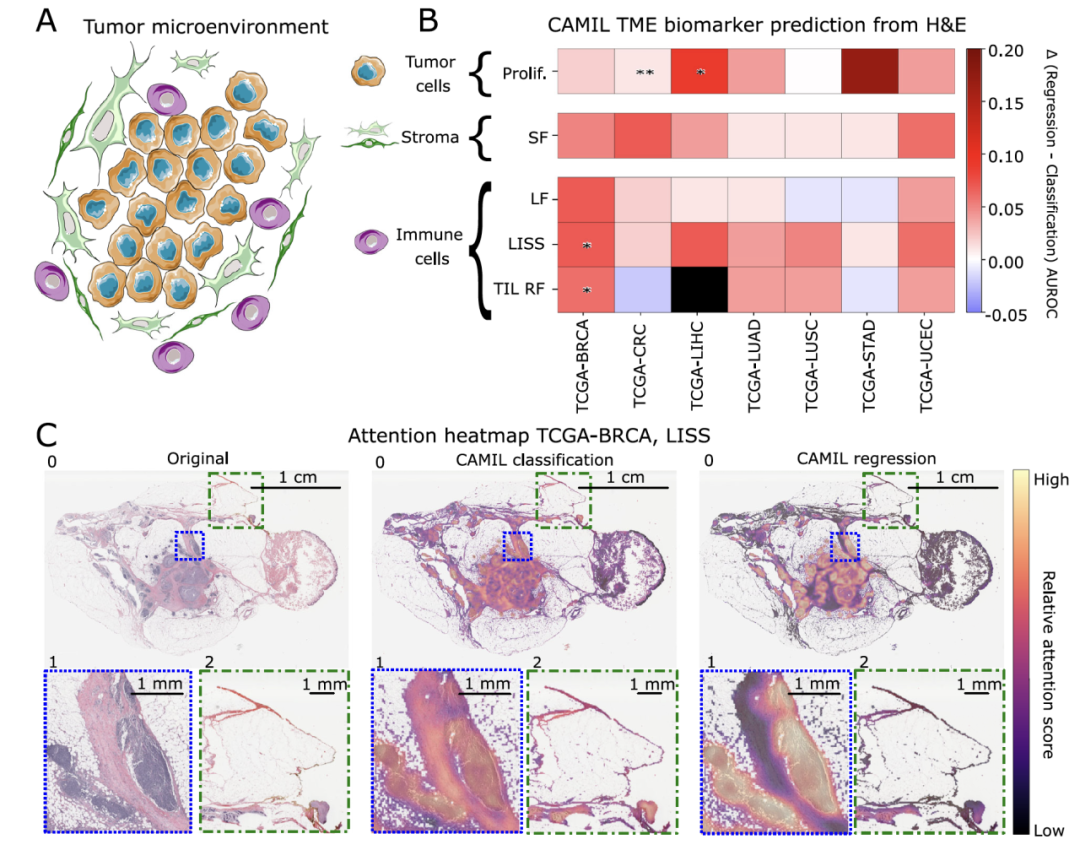
图 3
作者接下来探讨了CAMIL分类模型与CAMIL回归模型在与已知临床相关区域的对应能力方面的差异。为此作者评估了通过部署在TCGA队列测试集上训练的LISS回归模型和LISS分类模型获得的空间预测热图的生物合理性。尽管LISS作为一个弱标签,每个WSI只有一个分数,但一个鲁棒的模型仍应能够突出与LISS相关的区域,这些区域应主要包含淋巴细胞。实际上,可以看到分类模型和回归模型都将注意力放在了富含淋巴细胞的区域(图3c)。然而,在评估的WSI中,LISS回归模型对富含淋巴细胞的区域进行了更清晰的界定,并且对组织特征较不相关的区域关注较少。相比之下,LISS分类模型相对于回归模型在密集淋巴细胞区域的置信度较低,如较低的注意力分数所示。分类模型将重要性赋予无预设临床相关性的区域,模型突出了缺乏高密度淋巴细胞区域的组织边缘。Graziani等人的回归模型产生的热图也有类似的观察,它强调了没有预设临床相关性的区域,同时忽视了富含淋巴细胞的区域。作者通过KJH(一名病理学住院医师)的盲审对42个来自CAMIL分类和CAMIL回归模型的注意力热图进行了量化。基于专家审查,CAMIL回归方法产生的注意力热图在34个案例中更好地对应于已知的临床相关区域。在42个案例中,有6个案例中CAMIL分类方法更受青睐。在42个案例中的2个案例中观察到CAMIL分类和回归方法之间的表现相似。因此,基于盲审,CAMIL回归在81%的案例中胜过CAMIL分类。总的来说,这些数据表明回归方法为研究的生物标志物提供了统计上显著更好的AUROC,并且与已知的临床相关区域的对应能力显著优于CAMIL分类和Graziani等人的回归方法。
基于回归的生物标志物在结直肠癌中提高了生存预测
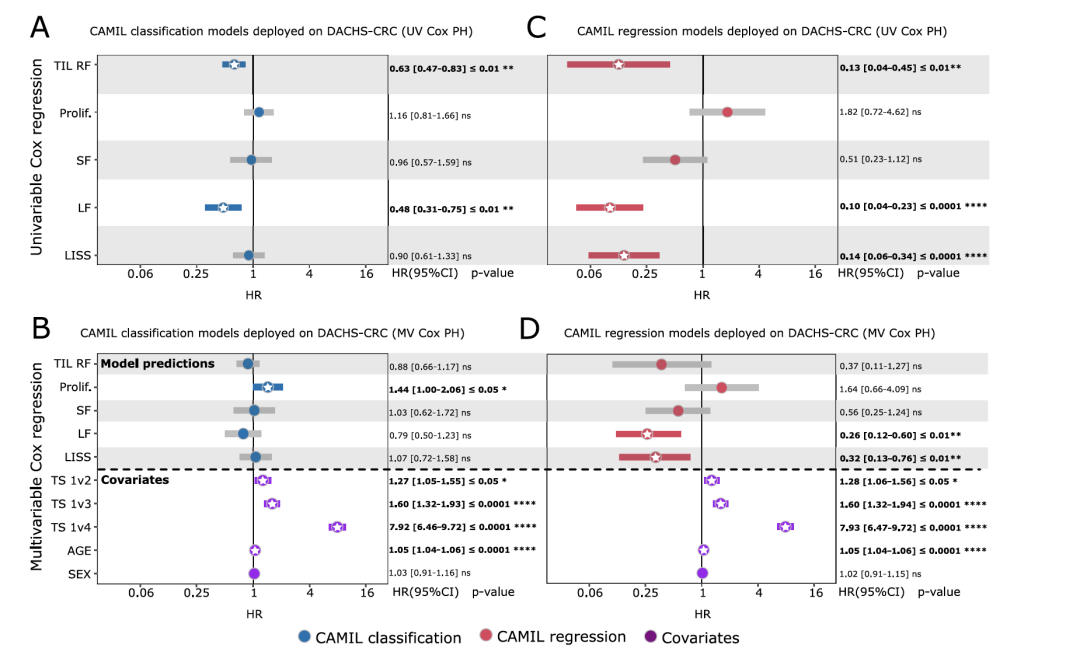
图 4
肿瘤细胞增殖、间质成分沉积和淋巴细胞浸润的生物过程在肿瘤发生和进展过程中具有生物学相关性,且已知与临床结果相关。因此,从H&E病理切片预测淋巴细胞浸润应该对预后评估具有相关性。作者在一个包含2297名结直肠癌患者的大型队列中调查了这一点,该队列来自“大肠癌:通过筛查预防的机会”(DACHS)研究,该研究提供了H&E整张切片图像(WSI)和长期(10年)随访数据,用于总生存分析。首先,作者在TCGA的乳腺癌患者上训练的模型,CAMIL分类和CAMIL回归展示了显著不同的AUROC。然后,作者在DACHS中登记的患者的WSI上部署了CAMIL分类模型。但是,作者没有使用分类模型的预测标签,而是采用了连续的分类得分,这允许与连续的回归得分进行更可比较的生存分析。采用这种方法后,作者使用单变量和多变量Cox比例风险模型评估了预测分类得分对总生存的预后影响,产生了风险比(HR)。在分析中,分类模型在2个生物标志物中观察到显著的风险分组分层(图4A),即TIL RF和白细胞比例显示了统计学上显著的结果。在多变量生存模型中(图4B),分类模型仅在1个生物标志物中展示了显著的预后能力。重复使用CAMIL回归模型获得的连续得分进行此过程后,可以发现回归模型显著提高了生存预测。回归模型在3个生物标志物中展示了显著的风险分组分层(图4A)。此外,在多变量生存模型中(图4B),回归模型在相同的2个生物标志物中展示了显著的预后能力。
编译 | 曾全晨
审稿 | 王建民
参考资料
El Nahhas, O.S.M., Loeffler, C.M.L., Carrero, Z.I. et al. Regression-based Deep-Learning predicts molecular biomarkers from pathology slides. Nat Commun 15, 1253 (2024).
https://doi.org/10.1038/s41467-024-45589-1



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


