今天为大家介绍的是来自Didier Rognan团队的一篇论文。超大型化学空间正在革新早期药物发现中的命中率识别。由于其庞大的规模,这些化学空间无法被完全枚举,因此需要特定的计算工具来导航这些空间,并挑选出可能有趣的命中目标。作者在这里提出一种基于结构的方法来筛选超大型化学空间,首先将商业化学试剂对感兴趣的目标进行对接,然后根据有机化学和拓扑规则以在目标的三维约束条件下枚举出类药化合物。

通常,通过实验或计算方法筛选药物样化合物库来识别能够针对特定大分子的首个命中化合物,进而使用传统药物化学策略进行后续的命中到先导化合物的转化。直到最近,可供筛选的描述药物样化合物的商业化学空间被限制在大约1000万到1500万种化合物,每年增长大约五十万种化合物。按需化合物库彻底改变了这种状况,它们有数十亿种尚未可用但可通过几个步骤轻松合成的化合物,并能进行可重复的并行合成。早期对超大化学空间子集进行虚拟筛选的方法取得了巨大成功,特别是高命中率、非常高的效力和选择性。今天,大约有700亿种化合物可以按需获得,提供快速交付(6-8周)和高纯度(>95%)。由于它们庞大的规模,这些超大化学空间的化合物无法被完全枚举,需要专门的计算工具进行检索。通常,大型化学空间是以组合的方式从构建模块和合成它们所需的有机化学反应来描述的。现在已经有基于配体的方法可以有效查询这些大型化学空间,但是包含宏分子目标信息(例如,结合位点的拓扑)的基于结构的方法仍需要开发,以彻底开采数十亿的化学空间。研究人员先前已经描述了几种这项任务的计算方法,尽管存在中等到严重的计算限制。在这里,作者介绍了一种简单快速的计算方法(SpaceDock),它避免了先前方法的缺点。首先需要将商业可获得的化学试剂与感兴趣的目标进行对接,然后根据标准的有机化学反应将它们结合起来,以构建一步或两步合成的数十亿化合物库。当应用于具有药物研究兴趣的目标时,这种方法能够迅速找到与现有配体化学上完全相同(或非常接近)的命中目标,同时也能提出化学上新颖且有效的配体。
构建基本条件
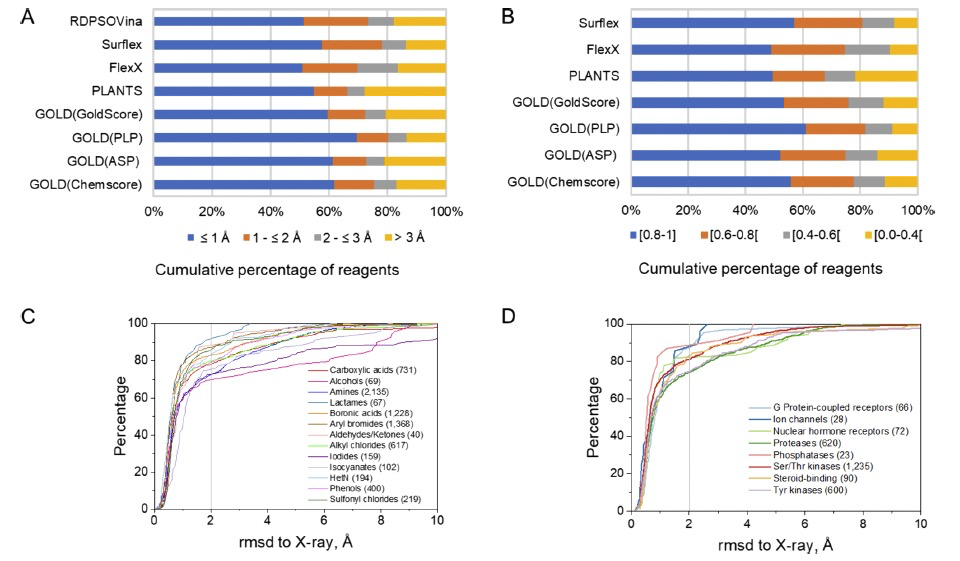
图 1
为了使SpaceDock方法有效工作,首先需要建立一个包含与蛋白质结合的化学试剂的参考3D结构库。因为没有这些数据的实验数据,所以作者通过分割已知的蛋白质-配体X射线结构中的类药物配体,并添加了缺失的活性部分(例如,硼酸、卤素等)的3D坐标,然后为这些试剂创建了一种假想的“X射线姿势”。最终挑选了5845种试剂,这些试剂展示了13种化学功能,主要是那些在药物发现中常用的活性团,如胺、芳卤化物和硼酸。拥有了一组参考试剂,作者接下来验证了最先进的对接算法是否能够复现这些替代X射线姿态。为此目的,使用了依赖不同原理的五种算法(FlexX:增量构建,GOLD:遗传算法,PLANTS:蚁群优化,RDPSOVina:随机漂移粒子群优化,Surflex:基于表面的分子相似性)。由于SpaceDock策略只需要一对互补试剂被适当对接来重构一个完整的配体,作者通过测量对接结果与假想X射线结构之间的根均方偏差(rmsd)来评估对接性能。结果显示,所有的对接工具都能够非常准确地完成对接任务,有70-80%的试剂在2 Å rmsd的精度范围内被成功对接(图1A)。由于rmsd是一个全局测量,它不考虑是否验证了关键的蛋白质-试剂相互作用,作者还计算了X射线姿态之间蛋白质-试剂相互作用指纹(IFPs)的相似性,这通过比较对接姿势和假想X射线姿势之间的相互作用指纹(IFPs)的相似度来实现。结果同样显示了出色的性能,有75-85%的试剂与X射线姿势的相互作用指纹相似度被认为是可接受的,图1B。为了确保所有化学功能都同样适合对接,对每个存在于库中的13个化学组(图1C)重复了相同的分析,专注于最佳的对接策略(GOLD对接和PLP评分)。令人放心的是,对接性能似乎相对独立于试剂的化学功能(图1C)以及目标蛋白质家族(图1D)。
构建化学空间
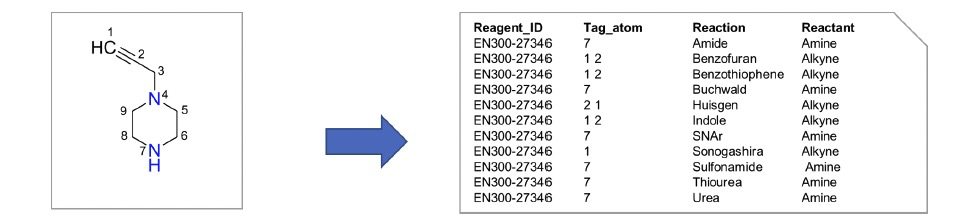
图 2
作者从简单的有机化学反应出发,定义了一个容易获得的超大化学空间。基于Hartenfeller等人的开创性工作,作者选择了36种稳健的、立体和区域选择性的有机化学反应,以定义一个可通过一到两步合成步骤轻松获得的55亿化合物的化学空间。与之前类似的方法不同,这里的化学试剂是从145,705种商业化学试剂的列表中仔细挑选的。此外,通过选择特定化学功能的单功能试剂并避免具有额外化学功能的试剂,从而减少了可能影响合成产率的副反应。总共有134,331种商业反应物被明确地标注了反应类型、反应物角色和反应活性原子,总计产生了713,155个原子标签(图2)。转换为3D原子坐标后,提供了176,824种准备就绪的独特试剂。
人类雌激素受体β(ERβ)激动剂
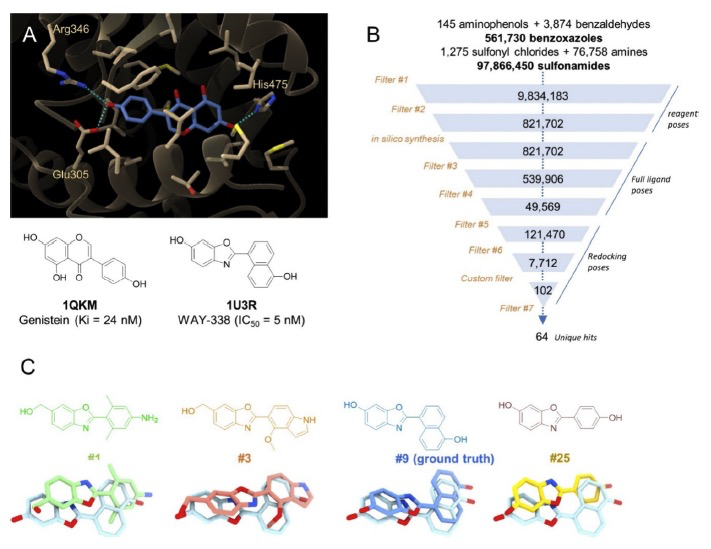
图 3
为了找到能作用于人类雌激素受体β(ERβ)的激动剂,作者对9700万种化合物进行了一次特殊的化学空间对接实验(图3)。作者选择ERβ的活化形式作为目标,主要是因为:(i) 它的配体结合部位既亲水又疏水,很适合药物作用;(ii) 已经有很多与之结合的高亲和力低分子量激动剂,特别是那些基于2-芳基-苯并噁唑结构的化合物,可以通过选定的36种反应之一简单合成。为了避免偏向某一特定化学类型,作者选择了一个与genistein(一种非苯并噁唑高亲和力激动剂)共晶的ERβ结构(PDB 1QKM),作为起点。作者想知道是否能够找到一个与已知苯并噁唑激动剂(WAY-338)或其近似物相似的化合物,方法是先对接必要的反应物(2-氨基苯酚和苯甲醛),然后促使苯并噁唑环在蛋白质结合位点形成。因此作者对145种2-氨基苯酚和3874种苯甲醛进行了3D建模和对接,探索了561,730种可能的苯并噁唑。由于这个空间相对较小,作者又考虑了一个更大的9700万个可合成磺胺酮的化学空间。在使用了之前最有效的对接协议(GOLD对接,PLP评分)处理所有必要反应物后,作者通过一系列越来越复杂的过滤步骤,从可能的反应物对开始,逐步筛选到质量检查过的重对接姿态。首先使用化学和拓扑过滤器快速去除不可能的反应。为了更加保险,作者只保留了那些与genistein结构相似度高的反应物对。经过一系列转换和最小化处理后得到了539,906个合适的姿态。通过更严格的过滤标准最终留下了49,569个姿态进行进一步处理。为了确保这些被选中的姿态真的有意义作者对所有剩余的潜在命中目标进行了重新对接。通过最后一轮质量检查保留了121,470个姿态,并通过一个特殊的过滤器进一步筛选,最终留下了102个姿态,来自64种独特的化合物,包括54种苯并噁唑和10种磺胺酮。尽管这些化合物在最初的化学空间中只占很小一部分(0.57%),但在最终的命中列表中,作者成功富集了84%的目标化学类型。在检查了这些命中目标的结构和结合方式,可以发现SpaceDock能够找到高排名的已知ERβ激动剂和其他2-芳基苯并噁唑化合物,几乎完美地模拟了它们的结合方式。
编译 | 曾全晨
审稿 | 王建民
参考资料
Sindt, F., Seyller, A., Eguida, M., & Rognan, D. (2024). Protein Structure-Based Organic Chemistry-Driven Ligand Design from Ultralarge Chemical Spaces. ACS Central Science.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


