今天为大家介绍的是来自Shengyong Yang团队的一篇论文。基于深度学习的分子生成技术在许多领域都有广泛的应用,尤其是在药物发现中。然而,目前大多数深度生成模型都是基于配体的,并没有在分子生成过程中考虑化学知识,这通常导致较低的成功率。在这里,作者提出了一个基于结构的分子生成框架,它明确考虑了化学知识(命名为PocketFlow),能够在蛋白质结合口袋内生成新的配体分子。

创新药物发现是一个极其复杂且成本高昂的过程,主要包括活性先导化合物检索、命中/先导优化、临床前评估和临床试验。其中,命中/先导化合物的检索是第一步也是关键步骤,因为它是启动新药开发项目的基础,而且能够显著影响后续药物开发步骤。传统上,命中/先导化合物的发现是通过对已知化合物库进行高通量筛选来完成的。然而,现有化合物库的有限结构多样性以及各药物开发机构或公司长期不断的筛选,使得检索新活性化合物和建立知识产权越来越困难。深度生成模型(DGMs),在生成图像、文本和声音方面取得了巨大成功,提供了一种高效的方法来生成全新的先导化合物。通常用于分子生成的DGMs主要是基于配体的,它们首先使用神经网络学习大量已知活性化合物的结构信息概率分布,然后通过采样学到的分布生成新的分子结构。然而,大多数这样的基于配体的DGMs没有考虑目标蛋白的结构信息,直到最近,这样的基于配体的DGMs被扩展到了结构基础的方法,纳入了目标蛋白的显式信息;考虑受体的结构信息被认为有助于提高药物设计的准确性,因为药物分子必须在体内精确地绑定到其对应的目标蛋白上,以发挥其特定的治疗效果。此外,许多蛋白质只有很少或没有已知的配体分子,对于这些蛋白,基于配体的DGMs不能用来生成分子。在蛋白质结合口袋内生成新配体分子的基于结构的DGMs有望克服基于配体方法的缺点,并且越来越受到关注。
尽管基于结构的深度生成模型(DGMs)可以基于蛋白质口袋条件生成新的分子结构,但仍存在许多挑战性问题,总结如下。首先,由已知实验蛋白质-配体复合结构组成的现有数据集较小,不足以训练生成模型。其次,当前基于结构的DGMs仍然是一种数据驱动的方法;越来越多的观点认为,将领域知识或规则引入深度学习模型可以有效解决数据不足、鲁棒性差和可解释性差的问题。第三,目前大多数深度分子生成模型在训练和生成过程中不考虑化学键信息。相反,它们输出一组无连通性的离散原子,然后通过第三方方法(如OpenBabel)组装这些原子形成分子。这种策略可能会导致许多不希望的亚结构,给化学合成带来困难或降低药物类似性。第四,尽管生成的分子在理论上已被验证,但其生物活性和结合模式并未通过湿实验室实验进行验证。受到上述挑战的启发,作者提出了一个由数据和化学知识驱动的基于结构的分子生成框架,命名为PocketFlow。
模型结构
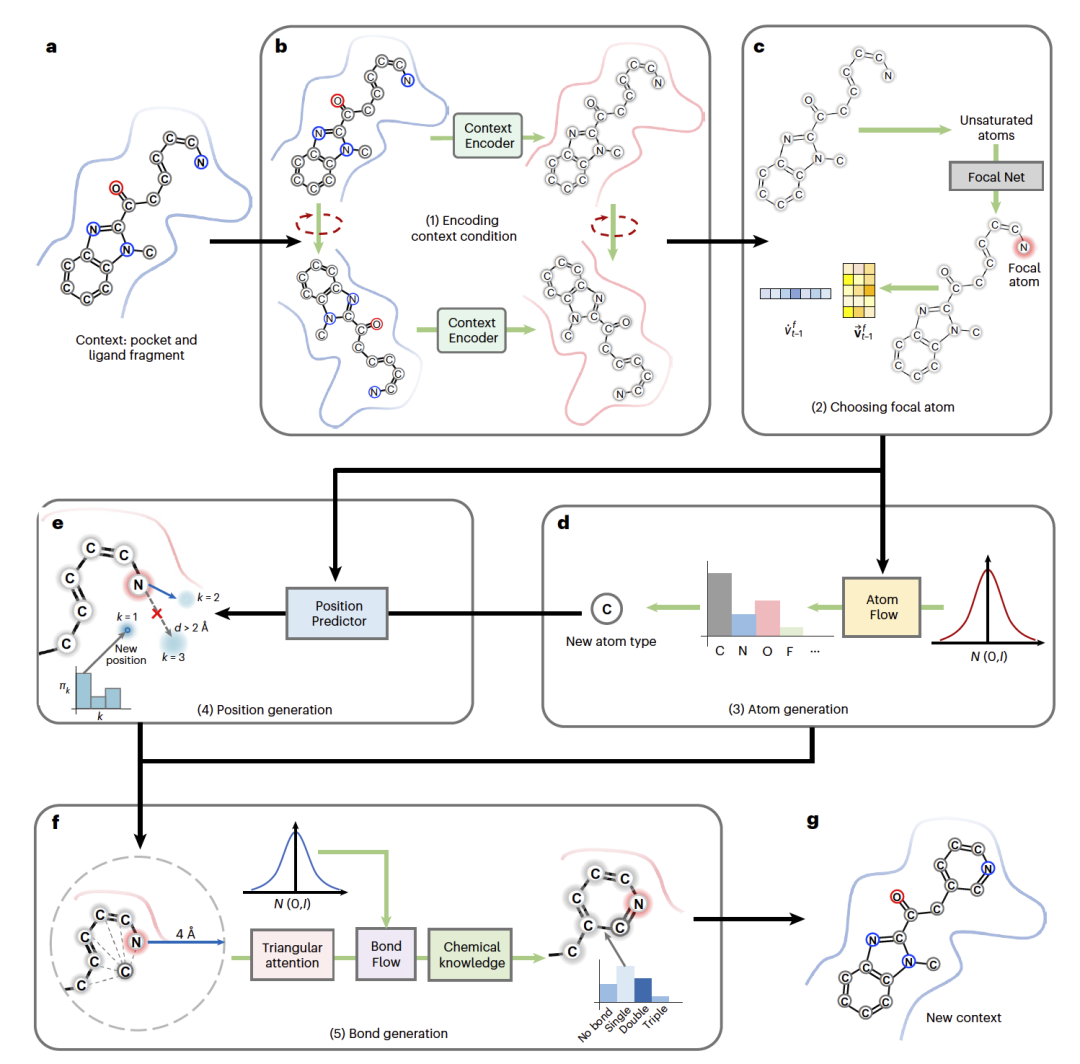
图 1
PocketFlow是一种自回归流(autoregressive flow)的生成模型,它能够逐步在蛋白质口袋内生成小型有机分子。本质上,它通过模型训练在基础分布(如正态分布)与给定3D口袋中的原子类型和共价键分布之间建立了一种可逆映射。在生成过程的每一步中,模型从基础分布中抽样以生成一个新的原子,连同其对应的坐标和共价键。模型将结合位点和已生成的分子片段作为环境信息(图1a)。环境编码器模块编码环境信息以提取环境特征C(t-1)(图1b)。在生成新组件之前,一个辅助网络,焦点网(Focal Net),用于从环境特征中选择一个焦点原子作为生成新原子的参考点(图1c)。基于这些环境特征,PocketFlow采用序列依赖策略来生成一个新的原子:原子类型a(t)、坐标r(t)和共价键e(t)(图1d-f),即a(t)→r(t)→e(t)。在生成过程中明确包含了化学知识约束,以确保生成分子的拓扑(图1f)和构象(图1e)是合理的。如果新生成的组件不满足化学知识约束,模型将重新抽样并生成其他组件以满足这些约束。在生成步骤结束时,a(t)、r(t)和e(t)被添加到环境中,作为下一个生成步骤的输入(图1g)。如果满足以下任一条件,则终止生成过程:(1) 不能预测到任何原子作为焦点原子,(2) 生成的原子数量达到预定义的最大值,以及(3) 重采样次数达到预定义的最大数量。
初步评估部分
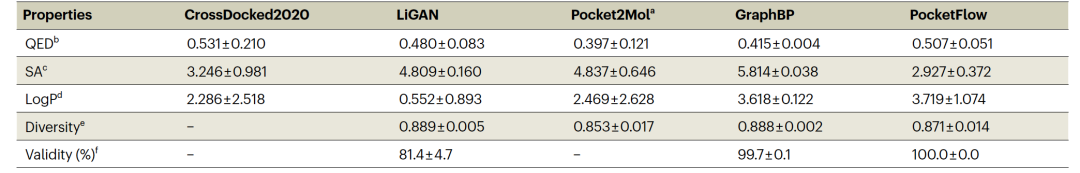
表 1
作者首先评估了由PocketFlow生成的分子的常见属性,包括药物类似性的定量估计(QED)、LogP(辛醇-水分配系数)、合成可及性(SA)、多样性和有效性。为了进行比较还计算了当前最先进的基于结构的深度生成模型生成的分子的五个属性。这里,使用了与评估LiGAN相同的测试集,包括具有PDB ID的十种不同类型的目标蛋白质。对于每个蛋白质口袋使用PocketFlow和三个基线模型生成了10,000个分子。为了比较目的作者还计算了CrossDocked2020数据集分子的属性,该数据集包含约13,000个真实的、类药物的小分子配体与蛋白质口袋结合。不同DGMs生成的分子和CrossDocked2020分子的五个属性的平均值显示在表1中。对于QED,只有PocketFlow的平均值在所有模型中超过0.5,非常接近CrossDocked2020分子的QED值,这表明PocketFlow具有更好的生成类药物分子的能力。由PocketFlow和三个基线模型生成的分子的平均LogP值介于0.552和3.719之间,位于类药物分子公认的LogP范围内。PocketFlow的SA分数为2.927,接近CrossDocked2020分子的SA分数(3.246)。
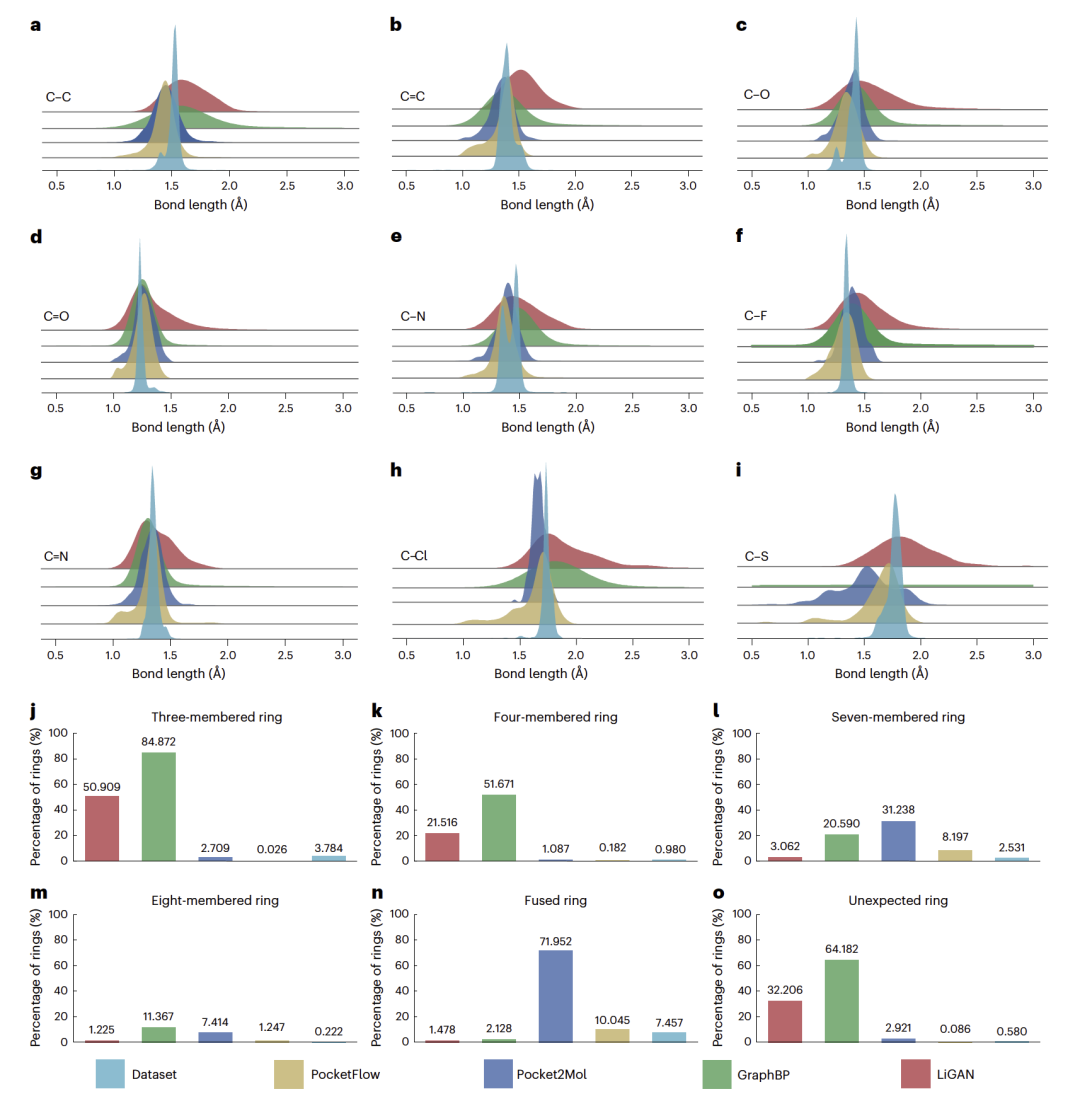
图 2
接下来作者评估了生成分子的化学结构的合理性,包括键长、键角和环结构。分析了九种常见的共价键。如图2a-i所示,对于所有九种共价键,由PocketFlow生成的分子的键长分布比三个基线模型生成的分子更接近CrossDocked2020分子的键长分布。对于八种常见的键角,由PocketFlow生成的分子的键角分布比三个基线模型生成的分子更接近CrossDocked2020分子的键角分布。对于环结构,五元和六元环及其融合环(如6+6、6+5和5+5)在类药物分子中最为常见,而三元、四元、七元及更大的环或多环结构(≥3个环融合)在类药物分子中不常见或不受青睐,这是由于它们的合成可及性差、化学稳定性低、毒性高和代谢不稳定。如图2j-o所示,含有上述不常见或不受青睐环的分子在由PocketFlow生成的分子中的百分比非常低,与CrossDocked2020分子非常相似(图2j-o中的青色)。相比之下,由三个基线模型生成的分子中含有不常见或不受青睐环的分子更多。
生成分子的结合位点以及结合亲和力/配体效率(LE)
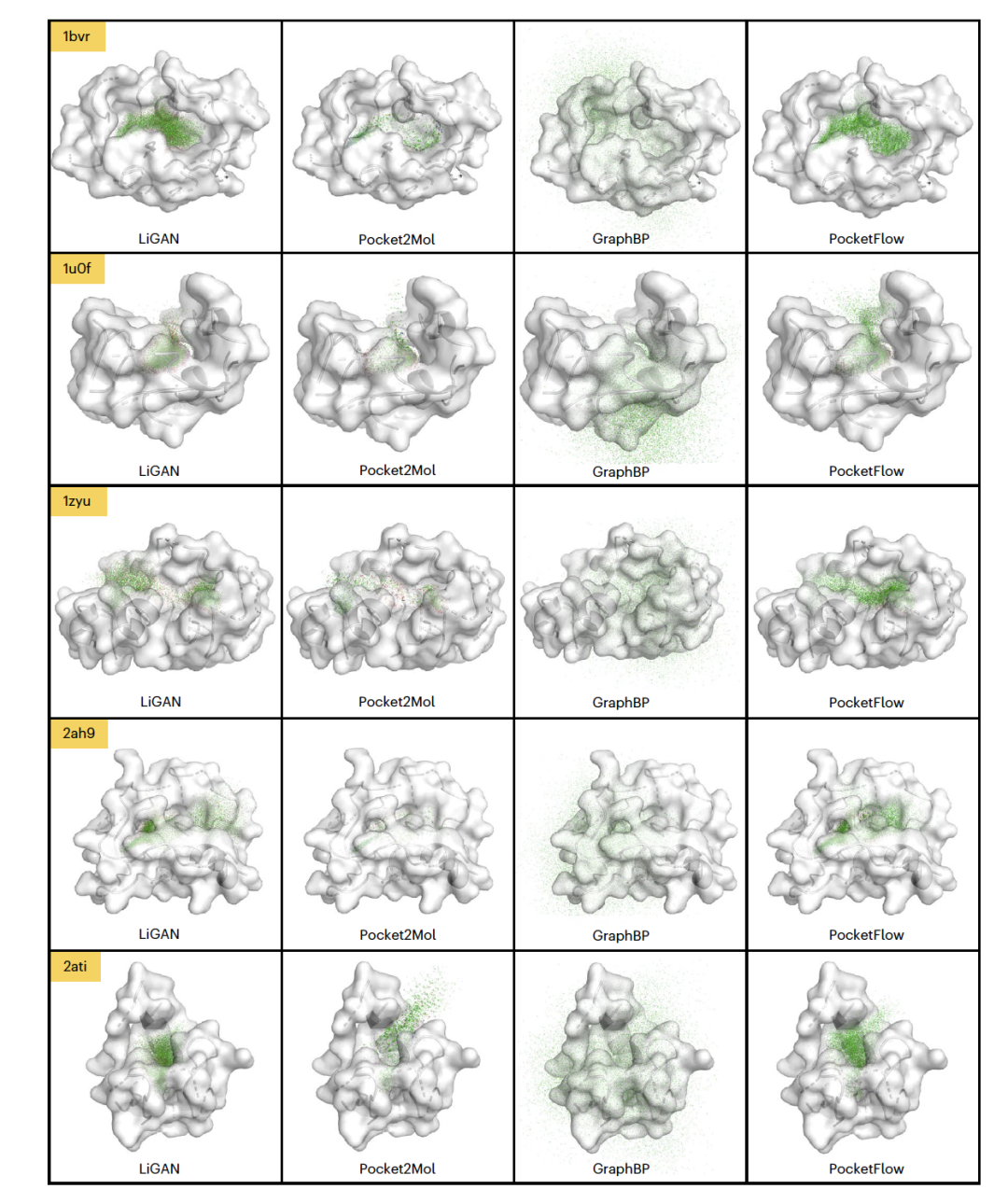
图 3
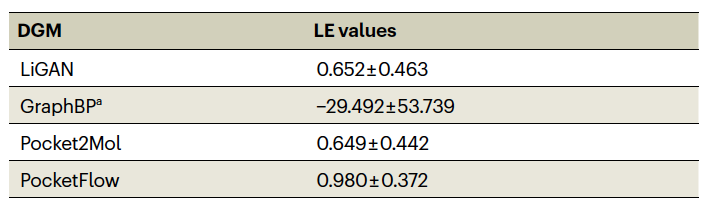
表 2
为了分析生成分子的详细结合位点作者从上述生成的10,000个分子中,针对每个靶标随机选取了1,000个分子进行统计分析。如图3所示,对于所有十种不同口袋形状的蛋白质,由PocketFlow生成的分子主要位于蛋白质口袋的内部,而由GraphBP生成的分子则分散地分布在蛋白质口袋周围,有大量分子位于口袋外部。由Pocket2Mol生成的分子也主要位于口袋内部,但Pocket2Mol的原子分布可能比其他三个模型更稀疏,这可能是因为Pocket2Mol生成的分子多样性较低,不同分子之间的原子坐标过于接近,导致许多原子在空间中重叠或聚集。与GraphBP和Pocket2Mol相比,LiGAN在某些靶标蛋白(例如,1bvr、2ati)上表现更好,可能的原因是LiGAN强制引入了一个额外的生成边界,将生成范围限制在一个盒子内。为了评估生成分子的结合亲和力和LE,作者使用ChemScore直接估计生成分子与相应靶标蛋白之间的结合亲和力。PocketFlow和Pocket2Mol在平均ChemScore值方面表现最佳,其中Pocket2Mol略优于PocketFlow。然而,在LE值方面,PocketFlow优于Pocket2Mol(表2),这是通过将ChemScore值除以重原子数得到的。一般来说,选择一个具有较大LE值的分子作为命中/先导化合物,可能意味着后续结构优化的空间较大。ChemScore值的分布显示,PocketFlow和Pocket2Mol有更高的概率生成具有高结合亲和力的分子。LE的分布表明,PocketFlow倾向于生成具有更高LE的分子。
编译|曾全晨
审稿|王建民
参考资料
Jiang, Y., Zhang, G., You, J., Zhang, H., Yao, R., Xie, H., … & Yang, S. (2024). PocketFlow is a data-and-knowledge-driven structure-based molecular generative model. Nature Machine Intelligence, 1-12.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


