
点击蓝字 关注我们

微末生物
Nanomega BioAI
作为当前结构生物学领域最热门的蛋白结构成像技术,冷冻电镜主要包括电子衍射晶体学(Micro-ED)、单颗粒冷冻电镜(Cryo-EM SPA)和电子冷冻断层扫描(Cryo-ET)三大类技术。其中,冷冻电镜单颗粒分析已逐渐成为结构生物学的重要工具,被广泛用于研究难以结晶的大分子复合物,以前所未有的方式深刻地改变着结构生物学领域,促进了生物学领域重大新发现的产生。
在这篇综述中,来自加州大学洛杉矶分校的周正洪教授围绕单颗粒冷冻电镜技术,从样品处理、仪器成像、重建以及模型构建等方面进行了详细的介绍,最后以3.8Å 分辨率解析的细胞质多角体病毒(CPV)的三维结构为例介绍了该技术在解决生物学问题方面的应用。
本文以Towards atomic resolution structural determination by single-particle cryo-electron microscopy为题,于2008年发表在结构生物学领域的国际期刊Curr. Opin. Struct. Biol.上(编者注:本文发表在2013年发生的冷冻电镜领域的“分辨率革命”之前,因此一些细节跟现在的冷冻电镜有所区别,但总体技术思路不变。读者可以将其当作冷冻电镜发展早期的里程碑式综述进行看待,也可以以此为契机首次详细了解冷冻电镜这一强大的蛋白质结构成像技术)。
一、单颗粒冷冻电镜技术
单颗粒冷冻电镜技术在在确定蛋白质高达亚纳米级别分辨率的 3D 结构方面发挥着越来越重要的作用,目前分辨率可达3.8 Å。在此分辨率下,可以解析蛋白质螺旋链的转角和深沟、β折叠中的链分层、无序环的密度和庞大的氨基酸侧链等细节结构特征。此外,该技术也可以用于分析CPV 、Epsilon 15 噬菌体和轮状病毒等生物体的结构特征,进一步扩展了其在生物学领域的应用价值。与传统经典的X射线晶体衍射方法构建出的蛋白结构模型相比,冷冻电镜的分析结果与X射线衍射的分析结果间存在良好的一致性。
二、实验流程
1、样品制备
单颗粒冷冻电镜技术对样品质量要求较高。冷冻电镜样品制备的目标是从新鲜制备的样品中获得网格上的冷冻水合样品,以尽量减少所研究的大分子复合物可能的变形或损坏。因此,结构完整的样品对于单粒子冷冻电镜至关重要。
冷冻剂的选择仍备受争议。很多研究在做CPV和轮状病毒时采用的是液氮冷却,分辨率为3.8Å,而在分析GroEL结构和Epsilon 15噬菌体结构时候选择的是液氦,分辨率为4.5Å。当同时使用液氮和液氦分析相同样品进行结构分析时发现,液氮的效果更好。主要原因是因为液氦温度下冰和生物样品之间的低分辨率对比度降低。作者使用FEI Polara G2 双温冷冻电子显微镜比较了液氮和液氦温度下冷却同一样品所获得的图像,从图像对比度、剂量耐受性和潜在分辨率极限等方面分析数据质量差异,最终发现液氦来冷却样品实现高分辨率成像并没有总体优势。
2、仪器和成像
当向超大粒子的高分辨率分析迈进时,景深(depth of filed,指在相机拍摄的图像中,处于清晰状态下的最近物体和最远物体之间的距离)问题成为一个限制因素。目前基于中心投影定理,在当前大多数3D重建程序中实现蛋白质三维重构的方法,在景深受到限制的情况下可能不再成立。使用更高的电压(更短的电子波长)缓解了这个问题。但对于直径高达800Å(80nm)或更大的粒子,使用300keV电子将分辨率提高到4Å以上,需要对传递函数进行完全校正。
除了视野仍然有限外,CCD相机作为记录介质比胶片具有许多显著优势,并且已被严格证明是单粒子冷冻电镜成像的首选记录设备,分辨率高达亚纳米级。
首先,宽动态范围、线性度和低噪声水平使这些器件成为记录二维晶体衍射强度的理想选择。
其次,CCD相机可提供有关图像质量的即时反馈,从而可以有效地优化样品制备和显微镜对准。
再次,无需在显微镜真空中装卸照相胶片的步骤,冷冻电镜网格的使用寿命可以从采用胶片时期的一天显著延长到 CCD方法的数天或数周。第四,尽管由于CCD相机的点扩散功能较差,高分辨率数据受到的抑制更严重,但CCD图像在低分辨率区域具有更好的信噪比,从而提供了更好的图像“对比度”,这对于对齐单粒子图像至关重要。
最后,与传统的照相胶片相比,CCD成像曝光的样品面积要小得多。
因此,它最大限度地减少了类似于“点扫描”方法的可能的光束引起的样品移动或带电,并减少了每个图像中散焦变化的影响。对于近原子分辨率的冷冻电镜重建,应严格筛选原始冷冻电镜图像,仅选择功率谱中可见对比度传递函数环高达5Å的图像。这些图像的功率谱中也不应有可见的标本漂移、电荷和散光,以确保在最终的 3D 重建中仅使用高质量的图像。
3、重建
单颗粒结构重建的数据处理包括两个基本步骤:取向中心参数确定和3D 重建。结构细化是作为这两个步骤的迭代过程进行的,通过逐步向更高的分辨率推进。各种软件包,包括EMAN、FREALIGN和IMIRS,已成功用于近原子分辨率结构重建。这些软件包中使用的方向中心参数确定原理是相似的,包括搜索公共线和匹配计算投影。对于3D重建步骤,EMAN 和 FREALIGN 使用直接傅里叶反演方法,这是计算效率最高的方法,但可能具有较大的内存要求(例如,800Å 粒子的内存要求为30GB),并且通常对噪声更敏感。相比之下,在IMIRS中实现的计算效率较低的傅里叶-贝塞尔合成方法和球谐波合成方法对图像噪声的敏感度较低,对存储器的要求也较低。总体而言,这些研究中使用的总计算时间相差几个数量级,并且可能与实现细节和用户程序的差异有关。
单颗粒冷冻电镜重建的有效分辨率可以通过严格评估密度图中解析的结构特征或标志以及使用独立重建图的统计分析来估计。通过使用EMAN中实现的高斯滤波器过滤原子分辨率模型,显示了三种类型的二级结构单元(α螺旋、β片和连接环)在不同分辨率下的模拟密度。在3.8–4.0Å分辨率下,理想的螺旋密度图应显示深凹槽和清晰的螺距。链间距离为4.4Å的β片中的链(氢键密度区域除外)和相距3.8Å的Cα原子的锯齿形图案也被解析。较大的氨基酸的侧链的密度也应该开始出现。在研究人员获得的 CPV 冷冻电镜密度图中,这些特征都存在,包括相邻 Cα 原子之间的 3.8 Å 距离和许多侧链密度数据明确、结构精准、令人信服(评估有效分辨率的常用统计标准包括傅里叶壳相关和频谱信噪比,二者被证明是等效的)。
通过检查图中的一些可分辨特征(例如α螺旋结构中轴的空隙、侧链的密度、β折叠片层之间的清晰分离)来评估模型质量至关重要。由于重建方法中可能存在系统误差,在使用传统的FSC 曲线(Fourier shell correlation)方法评估冷冻电镜图的有效分辨率时需要谨慎,尤其是当分辨率接近原子尺度时更是如此。
人们已经认识到,由于使用互相关方法(cross correlation)来进行基于模板匹配的数据优化的过程中存在模型偏差(model bias),可能存在的“噪音相关性”会导致误导性的“高分辨率”FSC评估结果(编者注:这一问题实际上反映了实验人员追求单项数据的结果。真正的评估数据应该落实到蛋白质结构模型与其冷冻电镜拍摄结果的对应效果上。然而,如果仅仅是为了追求分辨率数值而对目标结构进行调整,反而会使得结果产生更加严重的偏差。)种模型偏差问题对于低信噪比的图像数据(例如在近聚焦条件下的图像数据)或非对称、低对称性的目标物体以及小蛋白颗粒更为严重。
因此,有必要使用结构特征或立体化学作为内部控制来判断在模型优化的过程中是否发生了这种偏差。当分辨率达到近原子尺度时,上述的那些“可分辨特征”可用于判断FSC评估是否受到模型偏差的影响。另一种监测FSC评估可能偏差的方法是,根据两个不同的初始模型来优化整个图像数据集,并监测两种起始点优化结果的收敛性。最后,使用特定方法(如聚焦对方法focal-pair approach和无模型共线方法model-free common lines method)进行方向-中心估计(orientation-center estimation),可以消除模型偏差。后续地,通过对「方向」或「中心」变化的范围做出限制,基于模型的方向-中心优化可以作为一种“局部”搜索的监督方法发挥作用,降低在评估和调整分辨率的过程中发生“模型偏差”的风险。
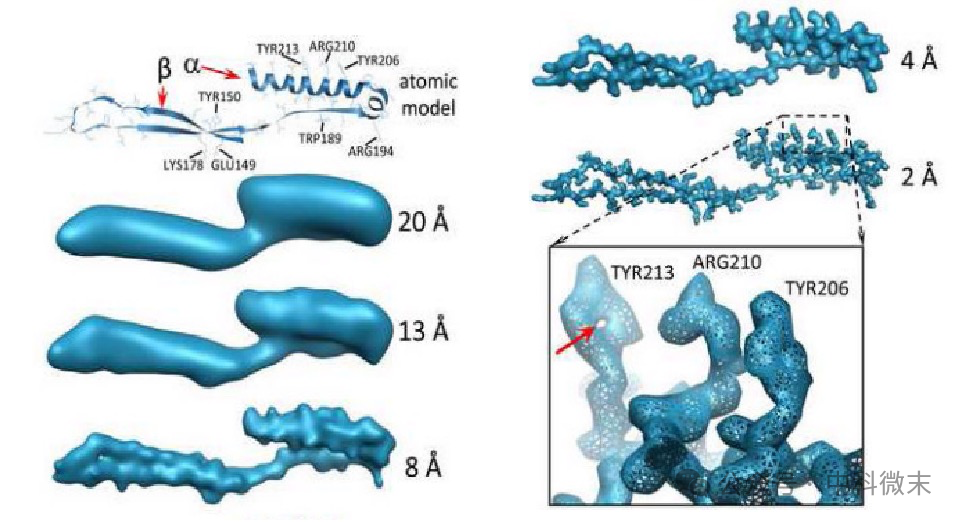
图 1.不同分辨率的二级结构元件。该片段是从HK97衣壳蛋白的原子模型中提取的,包括一个α螺旋和一个β发夹,它们通过环连接在一起,并使用EMAN过滤到不同的分辨率。在这种理想情况下,β发夹中的股线开始分离,较大的氨基酸的侧链在4Å处清晰可见。在2Å分辨率下,酪氨酸分子侧链芳香环上的一个孔被解析(红色箭头)。所有密度图均使用Chimera软件显示。
4、细分和模型构建
大体积蛋白复合物结构数据的渲染对计算要求非常高,并且可能是结构解释中的速率限制步骤。因此,研究人员希望从整个蛋白复合物群体中剖析或分割单个结构组件,以便方便地识别和比较详细的特征。对于二十面体病毒,只有不对称单元内的结构成分在结构上是唯一的,值得检查,而其他的部分不过是这一结构按特定规律进行重复。单个组分的分割还允许对结构相似的组分进行非二十面体平均,以提高信噪比并进一步提高平均亚基的分离度。例如,在由8400个颗粒重建的轮状病毒的不对称单元中,VP6分子(病毒蛋白6)的13个亚基的非二十面体平均显著提高了图谱的分辨率。通过平均降低VP6三聚体中的噪声,研究人员能够获得更好的侧链密度分辨率。
适用于解释近原子分辨率冷冻电镜结构的模型构建工具的选择有限。使问题进一步复杂化的事实是,冷冻电镜图通常包含许多亚基,具有超过 5000 个氨基酸,但其中只有不到10-20% 具有可识别的庞大侧链。这是有问题的,因为广泛使用的原子模型构建工具,最著名的是O和Coot,采用自下而上的方法,即从侧链到氨基酸,再到最终的原子模型。这种自下而上的方法对图谱质量和分辨率有严格的要求,并且不容易适用于近原子分辨率的冷冻电镜图。新的集成工具,如骨架化和图优化,可以与其他建模工具一起使用,以构建粗糙的蛋白质主链原子骨架模型。
同源结构的比对对基于近原子分辨率数据的模型构建有很大帮助。例如,CPV病毒,CSP病毒和正呼肠孤病毒λ1的蛋白序列(及其在BTV和RDV中的结构同源物)的序列同一性小于10%,这大约是两条随机之间的预期水平。因此,使用当前的序列比对程序,这些蛋白质没有可检测到的序列同源性。然而,冷冻电镜图清楚地显示,CSP与λ1具有相似的整体拓扑结构(或折叠),尽管螺旋有明显的插入和缺失。因此,在构建CSP-A和CSP-B模型时,可以使用已知的正呼肠孤病毒λ1结构作为拓扑参考。
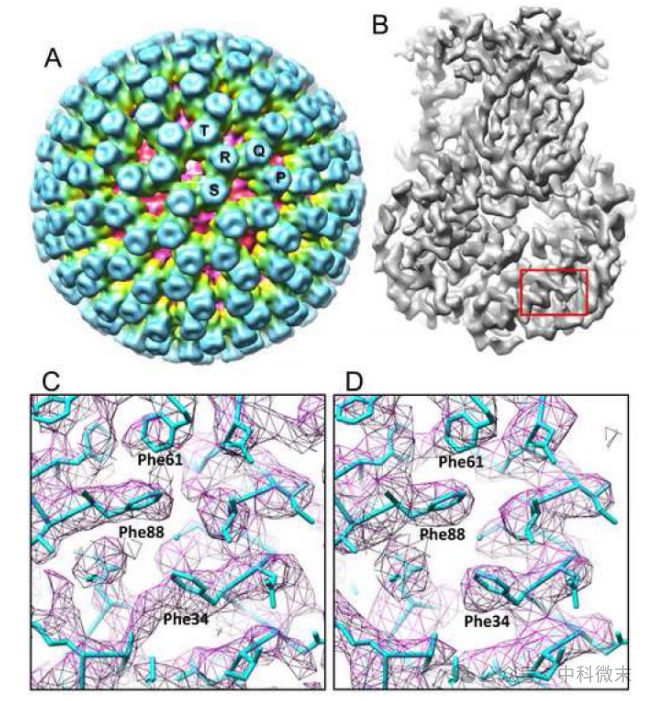
图2:轮状病毒结构显示了13倍非二十面体平均的能力。(a)轮状病毒DLP。(b)VP6 的三聚体经过13倍平均。在(c)平均之前和(d)平均之后,密度板((b)中的红色矩形)与x射线模型叠加。后者的效果明显较好。
三、应用举例
由于细胞质多角体病毒(Cytoplasmic Polyhedrosis Virus,CPV)和Epsilon 15噬菌体没有可用的X射线晶体结构数据,从其冷冻电镜结构得出的原子模型为功能意义提供了线索。CPV 的3.88-Å结构说明了近原子分辨率的冷冻电镜结构如何解决有趣的生物学问题。CPV在呼肠孤病毒科中是独一无二的,因为它在多面体包涵体中仅包含单层衣壳,但仍然完全能够进入细胞和复制。先前对CPV的冷冻电镜研究提供了有关其结构组织、转录酶复合物和基因组RNA组织以及分子相互作用和互补性的大量信息。然而,由于缺乏完整CPV衣壳的原子模型,许多基本问题仍然难以捉摸,包括RNA包装、转录、加工和释放的结构基础,以及CPV多面体包埋独特性质的机制。尽管与高分辨率X射线晶体结构相比,3.88 Å的分辨率仍然有限,但它让我们能够明确追踪主链并构建CPV蛋白的Cα模型,以帮助解决分子相互作用并提供功能解释。
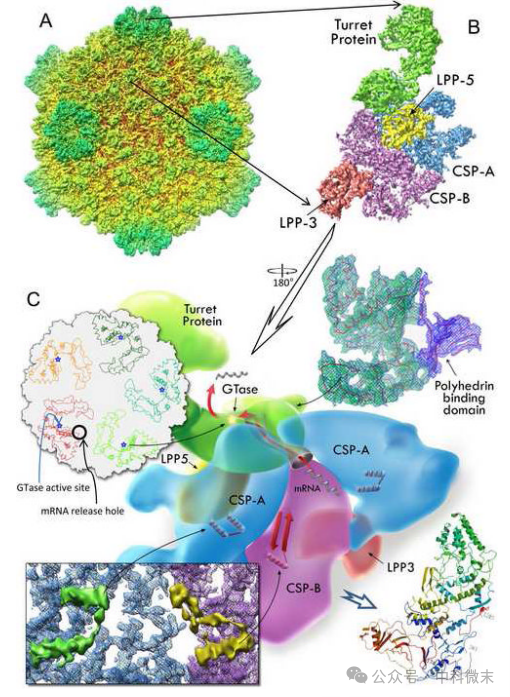
图3. CPV结构说明(3.88Å)解决三个生物学问题。(a)根据粒子半径着色的全图。(b)由蛋白质亚基着色的不对称单元,显示分子相互作用。(c)突出说明三个生物学问题的结构的示意图。顺时针方向为Turret Protein中多面体结合结构域的识别(右上),CSP亚基结构模型中对应位置从CSP- A的螺旋结构到CSP-B的β发夹结构的剧烈变化(下),以及GTase结构及其活性位点与mRNA释放孔的偶联(左上)。
在冷冻电镜的“分辨率革命”之前,冷冻电镜的分辨率远不及传统的晶体衍射技术,很难实现原子级的结构解析。在这样的背景下,仍旧有许多科研团队深耕冷冻电镜领域,想要发掘其潜力。来自加州大学洛杉矶分校的周正洪教授团队正是如此,他的团队在2008年首次在3.88Å超高分辨率条件下获得了CPV的完整三维结构,实现了病毒个体原子级解析,这是在冷冻电镜分辨率革命前的一项重大突破,也让科研工作者们看到了冷冻电镜的潜力。在前期的推文中,我们也介绍过2008年的这篇研究突破。
 点击文章标题阅读原文
点击文章标题阅读原文
目前,中科微末团队在生物大分子三维结构解析方面技术成熟、经验丰富,能够在原子级分辨率解析多种生物大分子材料的三维结构。我们致力于为药企和高校医院科研工作者提供一站式、自动化、原子级分辨率的结构解析服务,助力创新药物研发和科研成果转化,帮助科研人员实现“原子结构自由”。
期刊介绍
《结构生物学评论》(Current Opinion in Structural Biology)是一本专注于生化与分子生物学领域的学术期刊,创刊于1991年,由CURRENT BIOLOGY LTD出版商出版,出版周期Bimonthly。该刊发文范围涵盖生化与分子生物学等领域,旨在及时、准确、全面地报道国内外生化与分子生物学工作者在该领域的科学研究等工作中取得的经验、科研成果、技术革新、学术动态等。该刊已被SCIE、SCI数据库收录,2022年影响因子为6.8。
微末生物
Nanomega BioAI

请关注微末生物
期待与您交流




 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง





